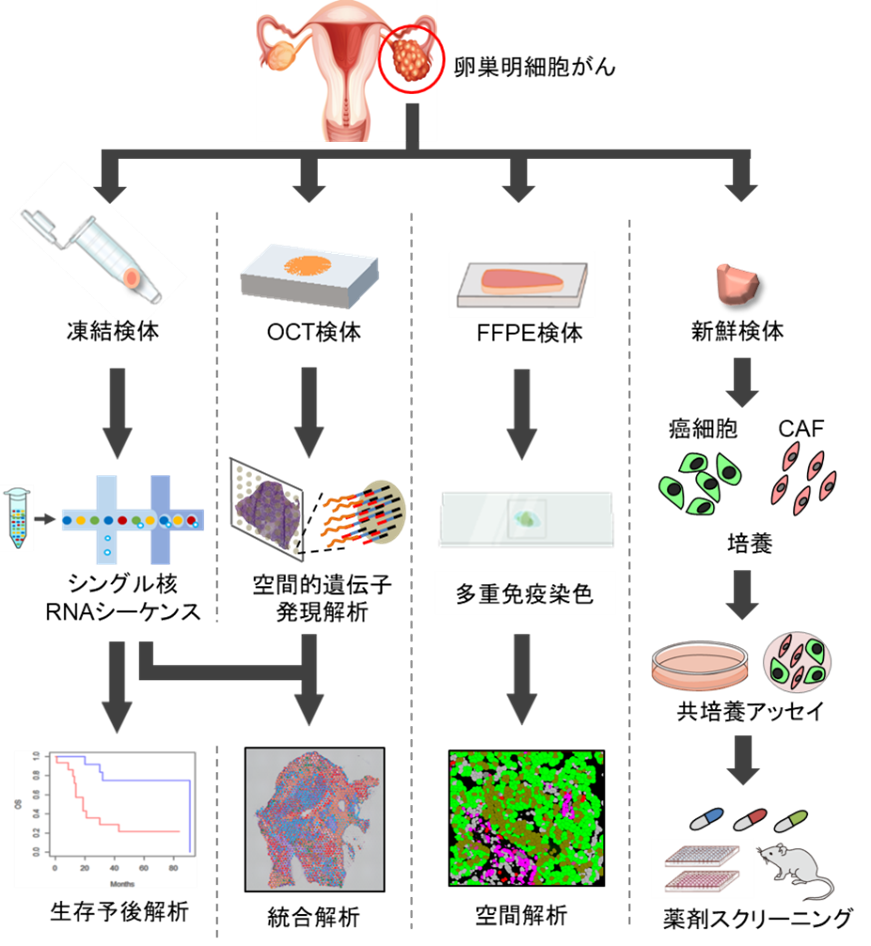
2024-04-25 理化学研究所,バイオ産業情報化コンソーシアム
理化学研究所(理研)生命機能科学研究センター 生体分子動的構造研究チームの嶋田 一夫 チームリーダー(バイオ産業情報化コンソーシアム(JBIC)特別顧問)、外山 侑樹 研究員の研究チームは、RNAヘリカーゼ[1]の一種であるDEAD-Box RNA helicase 3 X-linked(DDX3X)[1]タンパク質がRNAの複雑な高次構造[2]をほどく仕組みの一端を明らかにしました。
DDX3Xの機能不全はがんの一種である髄芽腫(ずいがしゅ)[3]などの疾患の要因になることが知られています。本研究成果は、これらの疾患の分子メカニズムの理解や新たな分子機序を持つ治療法の開発に貢献すると期待できます。
DNAから転写されることで生じるRNAは、立体的に折り畳まれたさまざまな高次構造を形成することによって、RNAにコードされたタンパク質の翻訳速度や、他のタンパク質との相互作用、細胞内における局在などを精緻に制御しています。DDX3Xは、このようなRNAの高次構造をほどく活性を有するタンパク質であり、RNA高次構造の形成と解消を介した細胞内RNA動態の制御に重要な役割を担っています。
今回、研究チームは溶液核磁気共鳴分光法(溶液NMR法)[4]を用いて、溶液中で1本鎖あるいは2本鎖の構造平衡[5]にあるRNAのうち、DDX3Xはほどかれた構造である1本鎖RNAに対して優先的に結合し、安定化すること、そしてこの結合特性によって多彩な高次構造を有するRNAを認識してほどくことを明らかにしました。
本研究は、科学雑誌『Nature Communications』オンライン版(4月25日付:日本時間4月25日)に掲載されました。
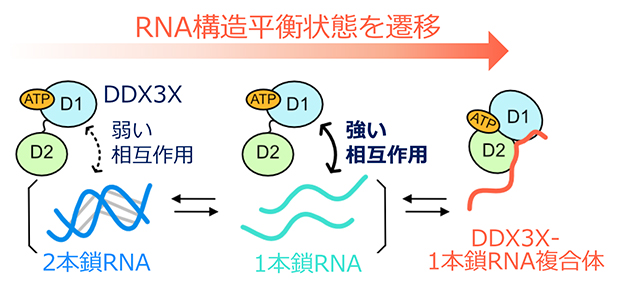
DDX3XがRNAの高次構造をほどく分子機構の模式図
背景
生物は遺伝情報をコードするDNAからRNAへの転写、RNAからタンパク質の翻訳という過程を経ることで、複雑な立体構造を持つタンパク質を合成し、生命活動を維持します。DNAが二重らせん構造をとることは有名ですが、DNAから転写されるRNAもまた折り畳まれた高次構造を形成することで、RNAがコードするタンパク質の翻訳速度、他のRNA結合タンパク質との相互作用、そしてRNAの細胞内局在などを精緻に制御しています。RNAの高次構造は、DNAの二重らせんに典型的に見られるような1本鎖が相補的に組み合わさった2本鎖構造[2]だけではなく、より多様なバリエーションを有しています。細胞内には、このようなRNAの高次構造をアデノシン三リン酸(ATP)[6]に依存してほどく活性を有するタンパク質、RNAヘリカーゼが存在し、RNAの高次構造の形成と解消を介してさまざまな生物学的過程の制御に関与し、重要な役割を果たしています。
DDX3Xは、RNAヘリカーゼの中でも最も大きなタンパク質ファミリーであるDEAD-Box RNAヘリカーゼの一種です。遺伝的な変異によるDDX3Xの機能不全ががんの一種である髄芽腫や神経疾患の要因となることからも、その機能はヒトの正常な生命活動に必須です。従って、DDX3XがRNA高次構造をほどく分子機構の解明は、RNAヘリカーゼが関与する生命現象を分子レベルで理解し、DDX3Xの機能不全による疾患に対する新規治療戦略を確立する上で重要です。
これまでにDDX3Xやその類縁タンパク質を対象として、RNAヘリカーゼとRNAが結合した複合体の立体構造がX線結晶構造解析[7]により明らかにされてきました。しかし、すでにほどかれた1本鎖構造を持つRNAとの複合体、もしくは構造がほどかれる前の2本鎖構造を持つRNAとの複合体構造が明らかとなっているのみで、その中間段階でDDX3Xがいかにして多彩なRNAの高次構造の変化を引き起こしているのか、そしてその駆動力は何であるかは十分に明らかになっていませんでした。
研究手法と成果
今回、研究チームは、ヒト由来DDX3Xタンパク質を対象とし、ヘリカーゼ活性を担うコア領域(残基番号132-607)を用いて、さまざまなRNAとの相互作用を詳細に解析しました。DDX3Xのコア領域はD1、D2と呼ばれる二つのドメイン構造から構成されており、D1ドメイン上にはRNAをほどく活性に必要なATPの結合部位が存在します(図1A)。DDX3XのRNAの結合能、および結合に伴う構造変化は、DDX3Xの溶液中における部位特異的な構造・運動性の情報を与える溶液核磁気共鳴分光法(溶液NMR法)を用いて解析しました。
まずは、ATP結合状態のDDX3Xを用いて、ほどかれる前の構造に対応する2本鎖RNA(グアノシン/シチジンから成る14塩基対のRNA)、およびほどかれた後の状態に対応する1本鎖RNA(10塩基のポリウリジン)との相互作用を解析しました。13C[8]により標識されたイソロイシン、およびメチオニンの側鎖メチル基を対象とした13C-1H相関NMRスペクトル[8]変化を指標に、それぞれのRNAの結合親和性および相互作用に伴う構造変化を評価したところ、DDX3Xは2本鎖RNAに対しては弱くしか相互作用しない一方で、1本鎖RNAとはより強固に結合し、複合体形成に伴ってDDX3Xに大きな構造変化が生じていることが分かりました(図1B)。また、DDX3X変異体の解析や、これまでに明らかとなっている結晶構造との比較から、1本鎖RNA結合状態はATPが両ドメインにまたがって結合し、D1とD2ドメインが閉じた構造に対応していることを明らかにしました(図1C)。同様の解析を、RNAをほどく活性が失われ髄芽腫の原因となるDDX3Xの2種類の病因性変異体(G302V変異体およびG325E変異体)についても適用したところ、これらの変異体では1本鎖RNAに対する結合能が失われていました。この結果はDDX3Xの1本鎖RNAに対する結合がRNAの高次構造をほどく活性の駆動力であり、この駆動力の喪失は疾患の発症と深く関連していることを支持しています。
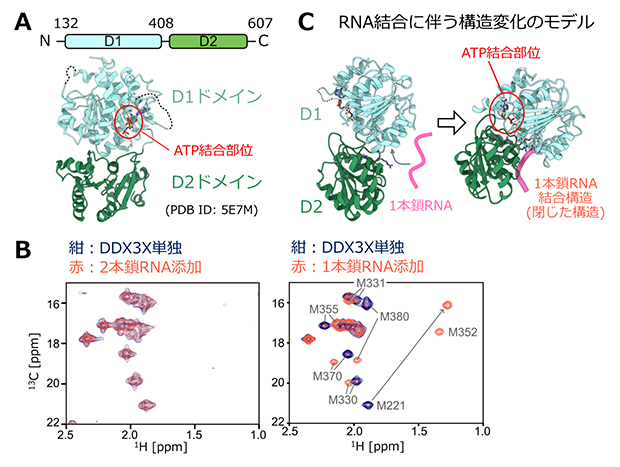
図1 DDX3Xタンパク質の構造と1本鎖、および2本鎖RNAとの相互作用
A.DDX3Xコア領域のドメイン構造(上)とATP結合状態の結晶構造(下)。立体構造はデータベース上で公開されているもの(PDB ID: 5E7M)を参照し、ATP結合部位(赤)、およびD1(水色)、D2(緑)ドメインを色分けして示した。なおATPはDDX3Xと結合するとADPに分解されてしまうため、このような構造解析では分解を受けない構造を持つATPアナログ(AMPPNP)がATPの代わりに用いられる。
B.DDX3Xのメチオニン側鎖メチル基の13C-1H相関2次元NMRスペクトル。DDX3Xに対して2等量の2本鎖RNA(左)、もしくは1本鎖RNA(右)を添加したときのNMRスペクトルの変化を示した。DDX3X単独状態のスペクトルを紺、RNA添加時のスペクトルを赤とし、重ねて表示した。1本鎖RNA添加時のスペクトルには、各メチオニンシグナルの帰属と、シグナルの変化の方向を灰色で示している。これらのシグナルの変化は、DDX3Xに大きな構造変化が生じていることを示す。
C.DDX3Xの単独状態(左)、および1本鎖RNAが結合して閉じた状態(右)に対応するDDX3Xの立体構造を示した。単独状態の構造はAMPPNP結合状態の結晶構造(PDB ID: 5E7M)、1本鎖RNA結合状態は予測による構造(ホモロジーモデル構造)。
さらに、DDX3Xの結合に伴い、RNA側にはどのような変化が生じているのかを明らかにするために、RNAのリボースの2’位に部位特異的に導入された19Fプローブ[9]を観測するNMR実験を行いました。この解析には、ウリジンとアデノシンから成る12塩基対の自己相補RNAを用いました。このRNAは単独状態において、自己会合によって生じた2本鎖構造と、解離した1本鎖構造との間の構造平衡状態にあり、二つの構造状態の存在割合は2個に分裂して観測される19Fシグナルの強度比から解析することが可能です(図2A)。DDX3Xの結合がRNAの構造、もしくはRNAの複数の構造間の平衡状態にどのような摂動を与えるかを調べたところ、DDX3Xの添加に伴い1本鎖構造の存在割合が増大したことに加え、結合状態に対応する新たな19Fシグナルが観測されました。そして、その化学シフト[10]値からDDX3X結合状態のRNAは1本鎖構造であることが分かりました(図2B)。このことは、DDX3Xは1本鎖構造を持つRNAを選択的に認識し、その構造を安定化することでRNAの構造平衡状態を遷移させ、その結果として2本鎖構造を1本鎖構造へとほどいていることを示しています(図2C)。
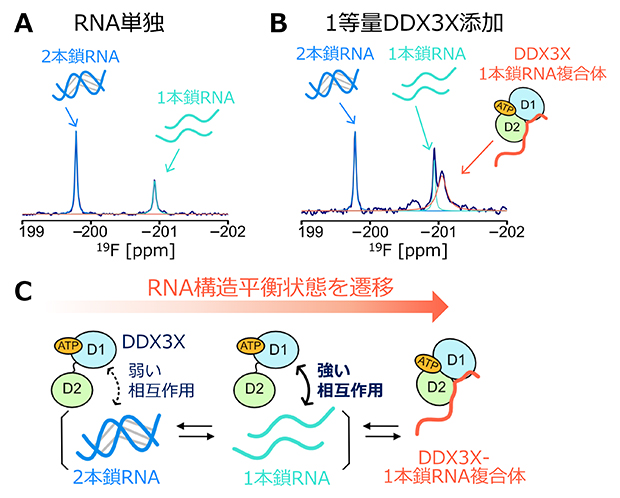
図2 DDX3Xとの相互作用によるRNAの構造平衡状態の遷移
A.11番目のアデノシンのリボース2’位置に19Fを導入した自己相補RNA(12塩基対)の19F NMRスペクトル。2本鎖構造(青)と1本鎖構造(水色)に対応する2個のシグナルが観測され、両状態の構造平衡状態にあることが分かる。
B.DDX3Xを添加したときの自己相補RNAのスペクトル。DDX3X結合状態に対応するシグナル(赤)が新たに観測され、化学シフト値から1本鎖RNAとの複合体状態であることが分かった。
C.DDX3XがRNAの高次構造をほどく分子機構の模式図。1本鎖構造のRNAに対してより強く相互作用することで、RNAの構造平衡状態を1本鎖構造へと遷移させ、RNAをほどいていることを表している。
DDX3XによるRNAの1本鎖構造への優先的な結合が、2本鎖構造だけではなく、RNAの多彩な高次構造をほどくことを可能にしているかを検証するため、同様の解析を分子内で塩基対を組むことで形成されるテトラループ[2]RNAを用いても検証しました(図3A)。14塩基から成るテトラループRNAとの相互作用を、DDX3Xの側鎖メチル基のNMRシグナルの変化から解析したところ、テトラループの結合に伴い、結合状態に起因するシグナルが新たに観測されました(図3B)。興味深いことに、これらのテトラループ結合状態に由来するシグナルの化学シフト値は、DDX3Xが1本鎖RNAと結合した閉じた状態に対応する化学シフト値とほぼ完全に一致することが明らかとなりました(図3B)。このことは、テトラループに結合した状態でも、2本鎖RNAの場合と同様、1本鎖構造に対する結合状態を形成することで、テトラループ構造をほどいていることを示しています。以上の結果から、DDX3Xは多様な高次構造を持つRNAに対しても、1本鎖構造を優先的に認識・安定化することで、その高次構造をほどくことが明らかとなりました。
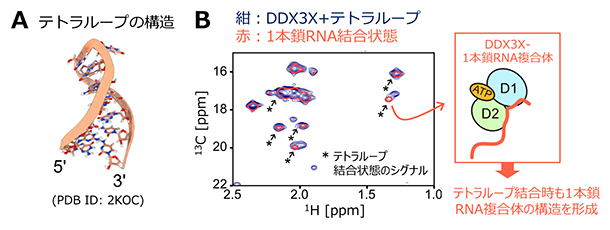
図3 テトラループRNAとDDX3Xとの相互作用
A.テトラループ構造を持つRNA(14塩基)のNMR構造(PDB ID: 2KOC)。
B.DDX3Xのメチオニン側鎖メチル基の13C-1H相関2次元NMRスペクトル。DDX3Xに対して6等量のテトラループRNAを添加したときのNMRスペクトルを紺で示した。テトラループRNA添加に伴い新たに観測された、結合状態に由来するシグナルをアスタリスクと矢印にて示している。DDX3Xと1本鎖RNAが結合した状態のスペクトルを赤色(単一等高線にて表示)で重ねて表示した。テトラループ結合状態のシグナル(アスタリスク)が1本鎖RNAが結合した状態(図1B)と同一の化学シフト値を持つことが分かった。
今後の期待
本研究では、DDX3Xとさまざまな高次構造を有するRNAとの相互作用、および相互作用に伴ってRNAに誘起される構造変化を、溶液NMR法を活用することにより解析しました。そして、DDX3XのRNAの1本鎖構造に対する優先的な結合が、RNAの構造平衡状態を遷移させることで高次構造をほどく駆動力となっていることを示しました。本研究から明らかとなったDDX3Xの分子機構は、DEAD-Box RNAヘリカーゼ全般に適用可能であることが予想され、RNAの高次構造の形成と解消によって制御されるさまざまな生命現象の分子レベルでの理解に貢献します。そして、これらの分子レベルでの知見は、RNA分子の構造や機能を制御するような、新たな創薬目標や治療戦略の指針となることが期待できます。
補足説明
1.RNAヘリカーゼ、DEAD-Box RNA helicase 3 X-linked(DDX3X)
RNAヘリカーゼは、RNAのヘリックス(らせん)構造などの高次構造を解消する機能を持つ酵素。DEAD-Box RNAヘリカーゼは、活性部位にアスパラギン酸-グルタミン酸-アラニン-アスパラギン酸(D-E-A-D)から成る保存された配列モチーフを持つことを特徴とし、RNAの高次構造をATPに依存して、ほどくRNAヘリカーゼのファミリー。ヒトにおいて最も大きなファミリーを構成する。DEAD-Box RNA helicase 3 X-linked(DDX3X)はX染色体上にその遺伝子が存在するDEAD-Box RNAヘリカーゼの一種である。
2.高次構造、2本鎖構造、テトラループ
アデノシン(A)、シチジン(C)、グアノシン(G)、ウリジン(U)の4種類のリボヌクレオシドから構成されるRNAが、分子間もしくは分子内で相互作用することで形成される立体構造をRNAの高次構造と呼ぶ。RNAの2本鎖構造は、分子間でAとU、もしくはGとCとの間で相補的な塩基対を組むことで形成される高次構造である。DNAの二重らせん型構造と類似しているが、リボースのコンフォメーションが異なるA型のらせん構造を典型的に形成することを特徴とする。ループはRNAに一般的に見られる高次構造の一つで、同じ分子内でRNA鎖が逆平行に向かい合い塩基対を形成する輪のような構造。このうち、塩基対を結ぶ領域が4塩基から構成される構造をテトラループと呼ぶ。
3.髄芽腫(ずいがしゅ)
小脳に発生する脳腫瘍の一種であり、特に小児における脳腫瘍で最も多い。小児における髄芽腫のサブグループであるWNTタイプではおよそ50%の頻度でDDX3Xの遺伝的変異が見いだされることが報告されている(Northcott et al., Nat Rev Cancer. 2012 12 (12) 818-834)。
4.溶液核磁気共鳴分光法(溶液NMR法)
溶液中の化合物や生体分子などを対象に、強い磁場中に置くことで生じる原子核の共鳴現象を観測することで、その化学構造や運動性といった情報を原子レベルで解析できる分光法。NMRはNuclear Magnetic Resonanceの略
5.構造平衡
タンパク質やRNAなどの分子が、一つではなく、複数の異なる構造間の交換状態にあること。
6.アデノシン三リン酸(ATP)
生物全般に存在するリン酸化合物の一つで、塩基と糖が結合した化合物(ヌクレオシド)に三つのリン酸が結合していることから、三リン酸と呼ばれている。加水分解により一つのリン酸が解離するとアデノシン二リン酸となる。この加水分解で得られるエネルギーはさまざまな生体機能に利用されることから、「エネルギー通貨」とも呼ばれる。
7.X線結晶構造解析
結晶化させた試料にX線を照射したときの回折パターンから電子密度分布を取得し、分子の立体構造を解析する方法。
8.13C、13C-1H相関NMRスペクトル
13Cは炭素の同位体の一つ。天然存在比(天然に存在する比率)約99%の12Cに対して、約1%の13Cが自然界には存在する。12C核はNMRで観測されないため、NMRでは13C核が対象となる。13C-1H相関NMRスペクトルは、隣接した炭素(13C)と水素(1H)間の情報をNMRで取得し、これらの情報から化合物や生体高分子の構造、部位特異的な化学環境の違いを解析する手法。
9.19Fプローブ
NMR法では多くの場合、電荷分布が対称で高分解能なシグナルを与えるスピン量子数が1/2の核種を対象とする。フッ素の同位体フッ素19(19F)はスピン量子数が1/2であり、またNMRの感度を決定する磁気回転比が軽水素(1H)と同程度と高いことから、そのシグナルを高感度、かつ高分解能に検出することが可能である。さらに、19Fは自然界の生体分子にはほとんど存在しないため、19Fを化学的に導入することでバックグラウンドのないシグナルを得られることが利点である。
10.化学シフト
NMRの共鳴周波数は、同じ核種であっても周辺の化学環境の違いに起因する磁場の遮蔽(しゃへい)効果が異なるため、その違いを反映してわずかに(100万分の1程度)異なる周波数を示す。この周波数の違いは化学シフトと呼ばれ、基準物質からのずれを指標としたparts per million(ppm)単位で表記される。化学シフトの変化を解析することで、構造変化や相互作用部位を同定することが可能となる。
研究チーム
理化学研究所 生命機能科学研究センター 生体分子動的構造研究チーム
チームリーダー 嶋田 一夫(シマダ・イチオ)
(バイオ産業情報化コンソーシアム 特別顧問、広島大学 副学長)
研究員 外山 侑樹(トヤマ・ユウキ)
研究支援
本研究は、理化学研究所運営費交付金(生命機能科学研究)で実施し、日本医療研究開発機構(AMED)「次世代治療・診断実現のための創薬基盤技術開発事業」(RNA標的創薬技術開発)による助成を受けて行われました。
原論文情報
Yuki Toyama, Ichio Shimada, “NMR characterization of RNA binding property of the DEAD-box RNA helicase DDX3X and its implications for helicase activity”, Nature Communications, 10.1038/s41467-024-47659-w
発表者
理化学研究所
生命機能科学研究センター 生体分子動的構造研究チーム
チームリーダー 嶋田 一夫(シマダ・イチオ)
(バイオ産業情報化コンソーシアム 特別顧問)
研究員 外山 侑樹(トヤマ・ユウキ)
報道担当
理化学研究所 広報室 報道担当
バイオ産業情報化コンソーシアム問い合わせ窓口