2024-05-10 理化学研究所
理化学研究所(理研)計算科学研究センター 粒子系生物物理研究チームの杉田 有治チームリーダー(開拓研究本部 杉田理論分子科学研究室 主任研究員)、ジョン・ジェウン 研究員(開拓研究本部 杉田理論分子科学研究室 専任技師)、タン・チェン 研究員の研究チームは、多成分の生体分子系の大規模粗視化分子動力学(MD)シミュレーション[1]を効率よく行うための新規アルゴリズムとソフトウェアを開発しました。
アミノ酸残基を1粒子として扱う粒度の粗視化分子モデル[2]は、分子動力学シミュレーションを用いて大規模生命現象を解析し、そのメカニズムを理解するための貴重なツールの一つです。しかし、このようなモデルを用いた超並列計算をスーパーコンピュータ上で行うためには、その計算アルゴリズムの大きな改善が必要でした。本研究では、動的な負荷分散を伴う不均一領域分割手法を開発し、スーパーコンピュータ「富岳」[3]などでの計算効率を大きく改善することに成功しました。この計算手法を「CGDYN」と名付け、理研を中心に開発しているマルチスケール分子動力学ソフトウェアGENESIS[4]に実装しました。本研究では、「CGDYN」を用いたシミュレーションを「富岳」を用いて行い、天然変性タンパク質(IDP)[5]から成る凝縮体(ドロプレット)の融合過程を観察し、小さなドロップレットの溶解と大きなドロップレットへの再融合に特徴付けられるオストヴァルト成長[6]を直接観察することに成功しました。今後、「CGDYN」を用いた粗視化分子モデルは、実験的に観察可能な生命現象を、シミュレーションを用いて計算機中に再現し、そのメカニズムの詳細を解明するための強力なフレームワークとして発展することが期待されます。
本研究は、科学雑誌『Nature communications』オンライン版(4月20日付)に掲載されました。
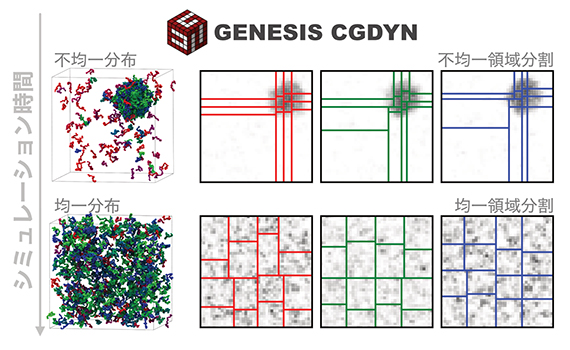
GENESIS CGDYNの動的負荷分散
背景
分子動力学(MD)シミュレーションは、生体分子の構造、ダイナミクス、安定性などの詳細な情報を提供し、生命現象を理解するための非常に強力な手法です。粗視化(CG)分子モデルは、複数の原子を単一の粒子として表現することで自由度を減少させ、演算量を減らします。
残基程度の粒度を持つCGモデルでは、アミノ酸に含まれる複数の原子を一つの粒子として表現し、大規模な生体分子系の長時間シミュレーションを行うために用いられてきました。このようなシミュレーションでは、水分子やイオンなどの溶媒を粒子として含まずに物理モデルとして溶媒効果を考慮した陰溶媒モデルがよく用いられます。このモデルでは、タンパク質などの分子を多く含む空間には多数の粒子が、そうでない空間には少数の粒子が分布します。このような不均一な粒子分布を持つ系の並列計算効率を高めるためには、各CPUコアに可能な限り均等に演算を振り分けることが重要です。多くのMDプログラムでは、均等なサイズのセルという単位で領域(空間)を分割し、各コアにセル内の演算を振り分けます。しかし、陰溶媒モデルを用いた粗視化MDシミュレーションで不均一な粒子分布を持つ系の超並列計算を効率的に行うためには、均等な空間分割では十分ではありませんでした。
研究手法と成果
本研究では、不均一な粒子分布を持つ生体系のMDシミュレーションを効率よく行うために新たな動的な負荷分散を用いた領域分割手法を開発しました。そして、理研を中心に開発しているMDソフトウェアGENESISにプログラム(CGDYN)として実装しました。新しい領域分割手法では、ターゲットとなるタンパク質などの分子を含む空間を分割する際に、分割したドメイン内の粒子数がほぼ同数になるように2分割することを繰り返します(図1a)。またシミュレーションを実行している間に、タンパク質などの分子構造が大きく変化したり、複数の分子の相対配置が変わったりすることで、空間の密度分布が急速に変化することがあり得ます。その場合、CGDYNでは自動的に不均一な領域分割を実行し、計算速度が低下することを避けることができます(図1b)。
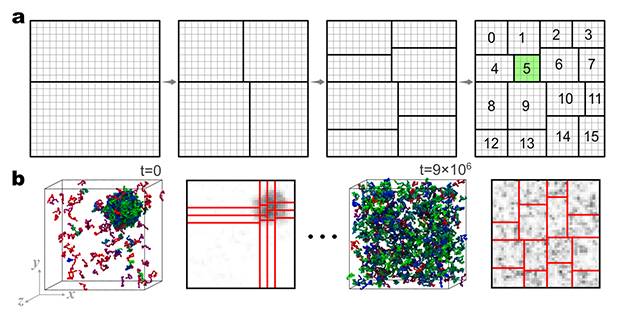
図1 GENESIS CGDYNの負荷分散アルゴリズム
(a)領域分割の概念図。分子を含む空間をドメインに分ける際に、ドメイン内の粒子数がほぼ同数になるように2分割することを自動的に繰り返す。
(b)動的負荷分散の概念図。t=0では、ボックスの中に大きな粒子密度の不均一性があるため、濃度の高い空間により多くのドメインを含むように分割されている。t=9×106ステップ後には密度がより均一になっているので、その密度に合うようにより均一なドメイン分割になる。
研究チームは、「富岳」および理研Hokusai-BigWaterfall[7]の二つのスーパーコンピュータを用いて、新しい動的領域分割手法を実装したCGDYNと、従来の均等なサイズのドメインに分割するSPDYN-likeおよび領域分割を使わないATDYNの二つのアルゴリズムの性能を比較しました。その結果、CGDYNは他の二つよりも計算速度に優れ、特にATDYNと比べ3~30倍高い性能を示しました(図2)。また、CGDYNでは高密度および低密度システム間でほぼ同一の計算性能を与え、この負荷分散手法が粒子密度によらず有効であることを示しています。CGDYNを使用したMDシミュレーションは、SPDYN-likeと比較して最大で7.5倍高速化されました(図2)。さらに、「富岳」上で他のMDソフトと性能を比較することで、CGDYNがより高い計算速度を持つことも実証しました。
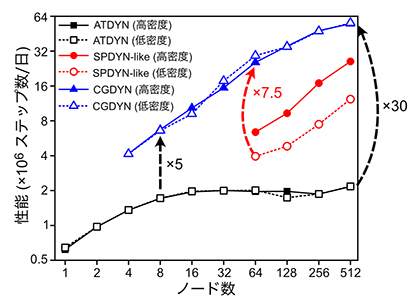
図2 GENESIS CGDYNのベンチマークによる計算速度の向上
新たに開発したCGDYNと、均等なサイズのセルへ分割するSPDYN-likeおよび領域分割を使わないATDYNの二つのアルゴリズムの性能を比較した。性能を1日で計算できるステップ数(x106)で表現し、ノード数(あるいはCPU数)を変えることでどのように計算量(ステップ数)が増えるかを示した。
CGDYNを用いた応用例として、さまざまなタンパク質凝縮体(ドロップレット)の融合過程の計算を行いました。16,647個の天然変性タンパク質から成る超大規模システムを構築し、50個以上の小さなドロップレットが0.1マイクロメートル(μm、1μmは100万分の1メートル)の直径を持つ単一の大きなドロップレットに融合する過程を観察しました(図3)。その結果、ドロップレット数の減少は、小さなドロップレットが大きなものに融合するだけでないことが分かりました。つまり、一部の小さなドロップレットは溶解し、そのタンパク質が希薄相に拡散した後、大きなドロップレットに吸収されることが明らかになりました(図3)。このプロセスは、表面張力と分子運動エネルギーの競合から生じるオストヴァルト成長を連想させます。ドロップレットのオストヴァルト成長は光学顕微鏡を通じて直接観察することが可能ですが、私たちの知る限り、残基レベルの粗視化モデルを用いたMDシミュレーションでこのような現象の観測に成功したのはこの研究が初めてです。

図3 超大規模タンパク質ドロップレットの融合過程の計算
時間(t)の経過に伴い、一部の小さなドロップレットは溶解し、そのタンパク質が希薄相に拡散した後、大きなドロップレットに吸収されることが分かる。シミュレーションは周期的境界条件を用いているため、2087×2067×2077Å3(1Åは100億分の1メートル)の直方体の中でのドロプレットの融合過程を計算した。
今後の期待
本研究で開発された手法とソフトウェアは、生体分子の凝縮体のより深い理解を提供するとともに、大規模な分子動力学シミュレーションシステムの長時間ダイナミクスを通じてメゾスコピック(ミクロとマクロの中間領域的)な生物学的現象を理解するための重要な計算基盤となることが期待されます。
補足説明
1.分子動力学(MD)シミュレーション
原子間に働く力を計算し、運動方程式を繰り返し解くことで、分子の動きを追跡する方法。
2.粗視化分子モデル
分子を構成する全ての原子(全原子モデル)の動きを追う分子動力学シミュレーションでは、計算コストが大きくなりすぎるため、扱える系の時間的・空間的サイズが限られている。より長時間で大規模な分子系の分子動力学シミュレーションを低コストで実行するために、一つのアミノ酸残基を1粒子で近似して(粗視化して)扱うことがよく行われており、これを残基粒度の粗視化分子モデルという。
3.スーパーコンピュータ「富岳」
スーパーコンピュータ「京」の後継機。社会的・科学的課題の解決で日本の成長に貢献し、世界をリードする成果を生み出すことを目的とし、2021年3月に共用が開始された。電力性能、計算性能、ユーザーの利便性・使い勝手の良さ、画期的な成果創出、ビッグデータやAI(人工知能)の加速機能の総合力において世界最高レベルのスーパーコンピュータ。15万8976個の中央演算装置(CPU)を搭載し、1秒間に約44京2010兆回の計算が可能。2020年6月から2021年11月にかけて、世界のスパコンランキング「TOP500」「HPCG」「HPL-AI」「Graph500」で4期連続の世界1位を獲得した。現在「富岳」は日本が目指すSociety 5.0を実現するために不可欠なHPC(ハイパフォーマンス・コンピューティング)インフラとして活用されている。
4.分子動力学ソフトウェアGENESIS
理研の粒子系生物物理研究チームを中心に開発されているソフトウェア。細胞内環境を含む大きな生体分子系のシミュレーションやレプリカ交換分子動力学法などの構造探索手法を利用することができる。詳細はGENESISウェブサイトを参照。
5.天然変性タンパク質(IDP)
生理的条件下で、固有の立体構造を保持しないタンパク質。完全にランダムな構造から部分的には二次構造を持つタンパク質まで幅広く存在する。天然変性タンパク質が持つ機能は近年、生物学において注目されている。
6.オストヴァルト成長
小さな粒子やドロップレットが溶解し、その溶質がより大きな粒子に移動して成長する現象。これは表面張力と分子の運動エネルギーの競合により生じる。
7.理研Hokusai-BigWaterfall
超並列演算システム、大容量メモリ演算サーバーと、フロントエンドサーバー、2種類のストレージから構成される。理研での科学技術研究などの推進と発展に資する研究のために使われている。
研究支援
本研究は、理化学研究所計算科学研究センターと開拓研究本部で実施し、日本学術振興会(JSPS)科学研究費助成事業基盤研究(S)「マルチスケール分子動力学シミュレーションによる細胞内分子動態の解明(19H05645、研究代表者:杉田有治)」、学術変革領域研究(A)「クロススケール細胞内分子構造動態の実験データ融合モデリング(21H05249、研究代表者:杉田有治)」「非ドメイン型バイオポリマーの分子動力学計算(21H05282、研究代表者:依田隆夫)」、文部科学省 データ創出・活用型マテリアル研究開発プロジェクト事業「バイオ・高分子ビッグデータ駆動による完全循環型バイオアダプティブ材料の創出(JPMXP1020230327、研究代表者:沼田圭司)」、文部科学省HPCI一般課題「Phase behavior of biomolecules studied with molecular dynamics simulations」(課題番号:hp200028)、「富岳」成果創出加速プログラム「全原子・粗視化分子動力学による細胞内分子動態の解明」(課題番号:hp200135、hp210177、hp220170)、「天然変性蛋白質の機能発現機構解明のための大規模シミュレーション」(課題番号:hp220101、hp230072)などによる助成を受けて行われました。
原論文情報
Jaewoon Jung, Cheng Tan, Yuji Sugita, “GENESIS CGDYN: large-scale coarse-grained MD simulation with dynamic load balancing for heterogeneous biomolecular systems”, Nature Communications, 10.1038/s41467-024-47654-1
発表者
理化学研究所
計算科学研究センター 粒子系生物物理研究チーム
チームリーダー 杉田 有治(スギタ・ユウジ)
(開拓研究本部 杉田理論分子科学研究室 主任研究員)
研究員 ジョン・ジェウン(JUNG Jaewoon)
(開拓研究本部 杉田理論分子科学研究室 専任技師)
研究員 タン・チェン(TAN Cheng)
報道担当
理化学研究所 広報室 報道担当