2024-07-30 ペンシルベニア州立大学(PennState)
<関連情報>
- https://www.psu.edu/news/research/story/genes-or-environment-new-model-understanding-disease-risk-factors/
- https://www.nature.com/articles/s41467-024-49566-6
MarketScanの5,000万人超を用いた遺伝性、環境リスク、大気汚染の因果関係の解明 Dissecting heritability, environmental risk, and air pollution causal effects using > 50 million individuals in MarketScan
Daniel McGuire,Havell Markus,Lina Yang,Jingyu Xu,Austin Montgomery,Arthur Berg,Qunhua Li,Laura Carrel,Dajiang J. Liu & Bibo Jiang
Nature Communications Published:25 June 2024
DOI:https://doi.org/10.1038/s41467-024-49566-6
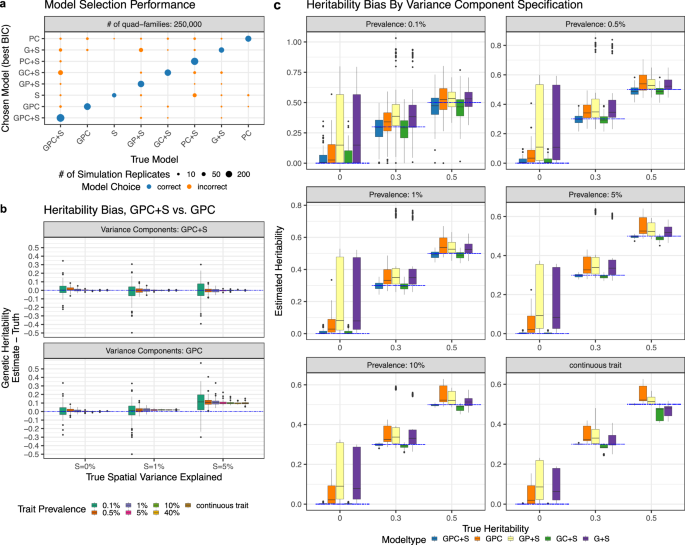
Abstract
Large national-level electronic health record (EHR) datasets offer new opportunities for disentangling the role of genes and environment through deep phenotype information and approximate pedigree structures. Here we use the approximate geographical locations of patients as a proxy for spatially correlated community-level environmental risk factors. We develop a spatial mixed linear effect (SMILE) model that incorporates both genetics and environmental contribution. We extract EHR and geographical locations from 257,620 nuclear families and compile 1083 disease outcome measurements from the MarketScan dataset. We augment the EHR with publicly available environmental data, including levels of particulate matter 2.5 (PM2.5), nitrogen dioxide (NO2), climate, and sociodemographic data. We refine the estimates of genetic heritability and quantify community-level environmental contributions. We also use wind speed and direction as instrumental variables to assess the causal effects of air pollution. In total, we find PM2.5 or NO2 have statistically significant causal effects on 135 diseases, including respiratory, musculoskeletal, digestive, metabolic, and sleep disorders, where PM2.5 and NO2 tend to affect biologically distinct disease categories. These analyses showcase several robust strategies for jointly modeling genetic and environmental effects on disease risk using large EHR datasets and will benefit upcoming biobank studies in the era of precision medicine.