2024-08-26 理化学研究所,東京大学
理化学研究所(理研)開拓研究本部 岩崎RNAシステム生化学研究室の岩崎 信太郎 主任研究員、七野 悠一 研究員、戸室 幸太郎 大学院生リサーチ・アソシエイト、水戸 麻理 テクニカルスタッフⅠ、藤 博貴 学振特別研究員PD、河本 尚大 基礎科学特別研究員、東京大学 生産技術研究所の池内 与志穂 准教授、周 小余 特任助教らの共同研究グループは、細胞内で「翻訳[1]」を行うリボソーム[2]の数やタンパク質がつくられる速度を初めて網羅的に計測しました。
本研究成果は、高効率なRNAワクチンの設計や、神経変性疾患やがんなどの翻訳異常を伴う疾患病態の理解へとつながるものと期待されます。
メッセンジャーRNA(mRNA)[3]からタンパク質を組み立てるプロセスである翻訳は生物種間において高度に保存されたメカニズムです。翻訳ではリボソームと呼ばれる巨大な分子複合体がmRNA上の分子暗号を解読しながら、タンパク質を組み立てます。しかし、これまで、mRNA上で翻訳を行っているリボソームの個数や、それぞれの翻訳の開始頻度といった基本的なパラメータですら計測することが難しく、その動態の全貌は不明でした。
今回、共同研究グループは、新たに「Ribo-Calibration法」を開発し、細胞内の翻訳パラメータを全遺伝子で同定しました。その結果から、ヒト細胞ではおよそ五つのリボソームがmRNA上で翻訳を行い、約22秒に1回翻訳が起きていること、mRNAは分解されるまで約1,800回使われることなどを明らかにしました。
本研究は、科学雑誌『Nature Communications』オンライン版(8月26日付:日本時間8月26日)に掲載されました。
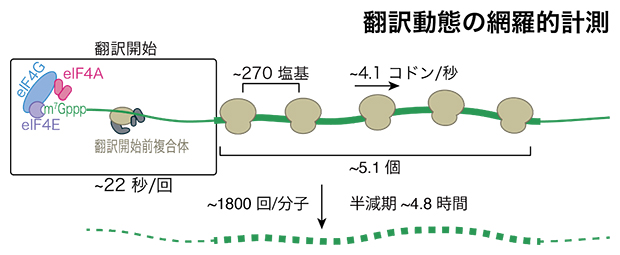
本研究で明らかになった細胞内翻訳速度
背景
DNAに書かれた遺伝情報は、mRNA、タンパク質と形を変え、実際に生体としての機能を果たします。中でもmRNAからタンパク質がつくられる過程は「翻訳」と呼ばれ、リボソームという分子によって実際にタンパク質が組み立てられていく重要なステップです。しかし、それぞれのmRNAが何個のリボソームによって同時に翻訳されているのか、翻訳を開始する頻度や速度はどれくらいなのかといった基本的なパラメータを測定することは困難でした。翻訳の脱制御はがんや神経変性疾患を含む疾患を誘導することが知られており、病態の理解につなげる上でも、これらのパラメータを知ることは重要な意味があります。
近年、mRNA上のリボソームの状態や翻訳動態を網羅的に解析する手法として「リボソームプロファイリング法[4]」が広く用いられています。リボソームプロファイリング法では、リボソームフットプリントと呼ばれる短いRNA断片を生成・回収し、次世代シーケンサー[5]を用いてその配列を読み解くことで、mRNA上の「どこに」、「どのくらい」リボソームが存在しているのかといった情報を網羅的に得ることができます。しかし、既存のリボソームプロファイリング法から得られる情報は翻訳の相対的な情報のみであり、絶対的な数値であるリボソームの数や翻訳開始[6]速度を知ることはできませんでした(図1)。
そこで共同研究グループは新規の手法を開発し、以上の問いにアプローチすることを試みました。
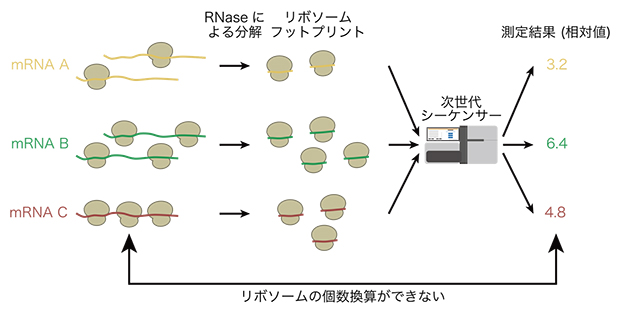
図1 リボソームプロファイリング法の特徴とその課題
既存のリボソームプロファイリング法ではmRNA上のリボソームの数について相対的な量しか知ることができなかった。
研究手法と成果
共同研究グループは既存のリボソームプロファイリング法を改変して「Ribo-Calibration法」を開発しました。Ribo-Calibration法では、既知のリボソーム数(モル比)から成る「mRNA・リボソーム複合体」を細胞に添加し、外部標準[7]として用います。すなわち、細胞由来のmRNAから得られたリード[8]をその外部標準由来のリードを用いて補正することで、それらを絶対的なリボソーム数に読み替えることが可能になりました。
まず、共同研究グループは、試験管内翻訳系を用いて、レポーターmRNA[9]を試験管内で翻訳した後、反応溶液を密度勾配遠心によって分画し、特定の個数のリボソームを含む分画のみを精製し、外部標準として準備しました(図2左)。これと細胞抽出液と混合しリボソームプロファイリング法とRNA-Seq法[10]を行いました(図2右)。
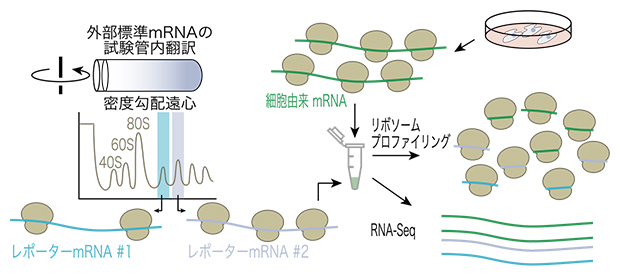
図2 Ribo-Calibration法の模式図
(左)二つのレポーターmRNAを試験管内で翻訳し、密度勾配遠心によって特定の個数のリボソームが結合したmRNAを含む分画を精製した。
(右)作成した外部標準と細胞抽出液(細胞由来mRNA)と混合し、リボソームプロファイリング法とRNA-Seq法を行った。
解析の結果、実験から得られたデータに基づいて計算されたリボソーム数が、理論的なリボソーム数とよく一致し、Ribo-Calibration法の正確性が確認されました(図3A)。そこで、計算を細胞内の全遺伝子に拡張したところ、ヒト細胞において約5個のリボソームがmRNA上で翻訳を行っていることが分かりました(図3B)。平均的にmRNAの長さが長いほど、翻訳を行っているリボソームの数も多く(図3C)、それぞれのリボソーム間の距離は270塩基程度でした(図3D)。さらに興味深いことに、一部のmRNAは非常に短いリボソーム間距離(およそ30塩基)を持っていました(図3E)。これはおよそリボソーム一つ分に相当する長さであることから、このようなmRNAではリボソーム同士が非常に近接しながらうまく翻訳を行っていると考えられます。
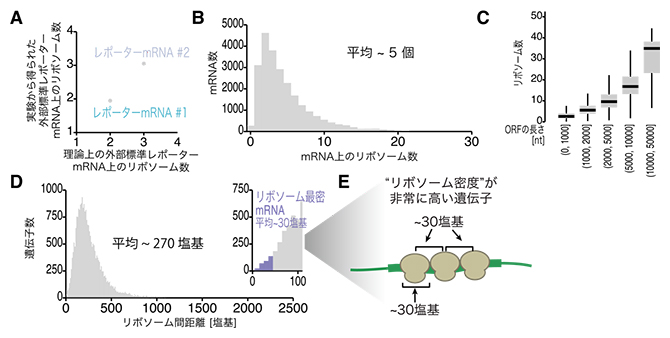
図3 Ribo-Calibration法によるリボソーム数の網羅的測定
(A)Ribo-Calibration法によって実験的に決定されたリボソーム数(縦軸)と理論的なリボソーム数(横軸)。
(B)細胞内のリボソーム数の分布。平均五つのリボソームが翻訳を行っていることが示された。
(C)細胞内のリボソーム数と遺伝子長(ORF(Open Reading Frame)長)の関係。ORF長が長いmRNAほど、多くのリボソームによって翻訳されている。
(D)細胞内のmRNA上のリボソーム同士の平均距離(塩基)。最も密度が高いものではおよそ30塩基ごとにリボソームが点在して翻訳していることが示唆された。
(E)非常に高い密度のリボソームによる翻訳の概念図。
次に共同研究グループは、Ribo-Calibration法とリボソームの伸長速度を測定することができる「リボソームランオフアッセイ法[11]」を組み合わせることで、翻訳開始速度を計算することを試みました。まず、mRNA上のリボソームの数は、1回の翻訳にかかる時間(翻訳開始速度)と一つのリボソームの滞留時間によって規定されるという数理モデルを作成しました。これを基に実験と解析を行った結果、ヒト細胞においては、翻訳伸長[12]速度が平均4コドン/秒であることが測定され(図4A)、平均すると22秒に1回の頻度で翻訳が起こっていました(図4B)。
また、mRNAの半減期を測定する手法であるRNA-Seq法とRibo-Calibration法から計算された翻訳の開始速度を組み合わせることで、1分子のmRNAが転写されてから分解されるまでに翻訳される回数が平均およそ1,800回であることを、明らかにしました(図4C)。
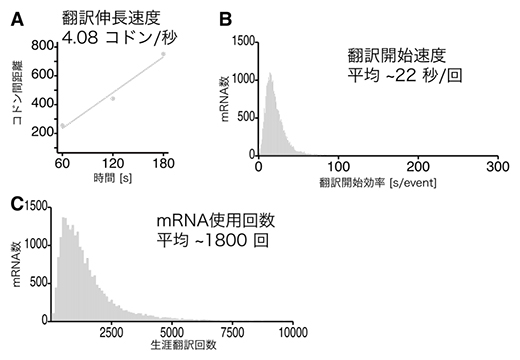
図4 Ribo-Calibration法による翻訳速度の網羅的測定
(A)リボソームランオフアッセイ法によって計測された翻訳伸長速度。
(B)翻訳開始効率の分布。平均およそ22秒に1度翻訳が開始されることが示された。
(C)生涯翻訳回数の分布。mRNAは分解されるまでにおよそ1,800回翻訳される。
さらに、共同研究グループは解析対象をiPS細胞、ケラチノサイト(表皮を形成する細胞)などの皮膚細胞、若年・老化マウスの脳組織まで拡張し、それぞれの細胞、組織におけるリボソーム数を網羅的に測定しました(図5)。これらの結果から、細胞種ごとにリボソーム数の多様性や、老化時における翻訳量の低下は、翻訳の効率よりもmRNAの転写量に起因していることなどが明らかになりました。
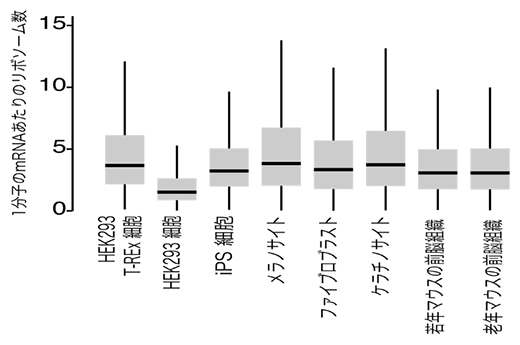
図5 さまざまな細胞種間におけるリボソーム数の多様性
Ribo-Calibration法によって計測されたさまざまな細胞種や組織間におけるリボソーム数。HEK293はヒト胎児腎由来細胞。T-RExはその亜種の一つを指す。メラノサイトはメラニン合成細胞。ファイブロブラストは線維芽細胞。
今後の期待
本研究では、細胞内の翻訳パラメータを網羅的に決定することができるRibo-Calibration法を開発しました。Ribo-Calibration法は細胞種、組織を問わず、広く応用が可能です。今後、翻訳異常と深い関連のあるがんや神経変性疾患に対して適用することで疾患病態の理解へつながるほか、高効率なmRNAワクチンの設計・評価などに活用できる可能性も期待されます。
補足説明
1.翻訳
mRNAへコピーされた塩基配列をアミノ酸配列へ変換して、リボソームでアミノ酸を順番につなげてタンパク質を合成すること。
2.リボソーム
リボソームRNA(rRNA)とリボソームタンパク質から構成される超巨大複合体。リボソームはメッセンジャーRNA(mRNA)にコードされているコドンを読み取り、タンパク質を合成する。
3.メッセンジャーRNA(mRNA)
タンパク質のアミノ酸の並び方の情報(コドン)を持つRNAのこと。リボソームによってそのコドンが読み取られ、タンパク質が合成される。
4.リボソームプロファイリング法
組織から翻訳装置であるリボソームを抽出し、リボソームと結合しているRNA配列を同定することで、どの遺伝子がどの程度の効率で翻訳されているかを知る解析法。リボソームは大きな複合体であるため、一定のmRNA領域を覆うように結合する。これらのリボソームとmRNAの複合体をRNA分解酵素で処理すると、リボソームが保護するmRNA断片だけが分解されずに回収される。
5.次世代シーケンサー
DNAの塩基配列を決定するための装置。複数のDNA断片の塩基配列を同時並行で、高速・高精度に決定できる。
6.翻訳開始
リボソームがmRNA上の開始コドン上に結合するまでの一連の素過程を指す。
7.外部標準
既に濃度などの量があらかじめ分かっている標準物質のことを指す。外部標準に照らし合わせ解析することでサンプル間のデータのばらつきを標準化することができる。
8.リード
次世代シーケンサーで決定されたDNA断片の個数のこと。リードの個数の増減で、それに相当するDNAあるいはRNAがどれくらいの量であったかを相対的に定量することができる。
9.レポーターmRNA
遺伝子の発現を簡便に確認するために用いられる、外来性のmRNA。代表例としてはルシフェラーゼや緑色蛍光タンパク質(GFP)が挙げられる。
10.RNA-Seq法
次世代シーケンサーを使ってRNAの量を網羅的に計測する手法。RNAを逆転写によりDNAに変換し解析する。
11.リボソームランオフアッセイ法
時間を置きmRNA上で翻訳反応(ラン/run)しているリボソームに、翻訳を終結させ、乖離(かいり)(オフ/off)させる実験手法。翻訳開始を阻害することで実験が可能になる。
12.翻訳伸長
mRNA上にリクルートされたリボソームが、コドンを認識し、ペプチドを伸長させる反応を指す。
共同研究グループ
理化学研究所 開拓研究本部 岩崎RNAシステム生化学研究室
主任研究員 岩崎 信太郎(イワサキ・シンタロウ)
研究員 七野 悠一(シチノ・ユウイチ)
大学院生リサーチ・アソシエイト 戸室 幸太郎(トムロ・コウタロウ)
テクニカルスタッフⅠ 水戸 麻理(ミト・マリ)
学振特別研究員PD 藤 博貴(トウ・ヒロタカ)
基礎科学特別研究員 河本 尚大(カワモト・ナオヒロ)
東京大学 生産技術研究所
准教授 池内 与志穂(イケウチ・ヨシホ)
特任助教 周 小余(チャウ・シウユ)
京都大学大学院 薬学研究科
教授 土居 雅夫(ドイ・マサオ)
助教 三宅 崇仁(ミヤケ・タカヒト)
研究支援
本研究は、RIKEN Pioneering Projects「Biology of Intracellular Environments(研究分担者:岩崎信太郎、七野悠一)」で実施し、日本学術振興会(JSPS)科学研究費助成事業基盤研究(B)「ミトコンドリア特異的リボソームプロファイリング法によるオルガネラ翻訳網羅解析(研究代表者:岩崎信太郎)」、同基盤研究(A)「細胞内構造を支えるヘテロポリマー間相互作用の網羅的解析(研究分担者:岩崎信太郎)」、同基盤研究(C)「細胞内顆粒P-bodyによるmRNAの空間的制御を介した細胞周期調節機構(研究代表者:七野悠一)」、同若手研究「翻訳開始因子eIF4A1とグルタミン代謝による協調的な発現制御機構(研究代表者:七野悠一)」「リボソームプロファイリングを応用したリボソーム構造動態の解明(研究代表者:藤博貴)」、同研究活動スタート支援「溶連菌毒素による宿主翻訳ハイジャック機構の解明(研究代表者:藤博貴)」、同特別研究員奨励費「細胞侵入型病原細菌による宿主翻訳の撹乱(研究代表者:藤博貴)」「青色光適応メカニズムから概日的遺伝子発現制御の意義を理解する(研究代表者:河本尚大)」、同学術変革領域研究(B)「新規Disome-Seq法:パラメトリックなリボソーム渋滞の網羅的探索(研究代表者:岩崎信太郎)」「柔軟な神経らしさを作り出すパラメトリック翻訳制御の解明(研究代表者:池内与志穂)」、同学術変革領域研究(A)「時間タンパク質学:翻訳速度の大規模並列網羅解析(研究代表者:岩崎信太郎、研究分担者: 池内与志穂)」「APEX-Ribo-Seq:近傍標識による非典型局所翻訳の網羅解析(研究代表者:七野悠一)」「非典型局所翻訳を包括的に解明するAPEX-Ribo-Seq法の確立(研究代表者:七野悠一)」、国立研究開発法人日本医療研究開発機構(AMED)革新的先端研究開発支援事業ユニットタイプ(CREST)「プロテオスタシスの理解と革新的医療の創出」研究開発領域「神経変性疾患におけるアグリゲーションと翻訳の陰陽(研究代表者:岩崎信太郎、研究分担者:池内与志穂)」による助成を受けて行われました。
原論文情報
Kotaro Tomuro, Mari Mito, Hirotaka Toh, Naohiro Kawamoto, Takahito Miyake, Siu Yu A. Chow, Masao Doi, Yoshiho Ikeuchi, Yuichi Shichino, and Shintaro Iwasaki, “Calibrated ribosome profiling assesses the dynamics of ribosomal flux on transcripts”, Nature Communications, 10.1038/s41467-024-51258-0
発表者
理化学研究所
開拓研究本部 岩崎RNAシステム生化学研究室
主任研究員 岩崎 信太郎(イワサキ・シンタロウ)
研究員 七野 悠一(シチノ・ユウイチ)
大学院生リサーチ・アソシエイト 戸室 幸太郎(トムロ・コウタロウ)
テクニカルスタッフⅠ 水戸 麻理(ミト・マリ)
学振特別研究員PD 藤 博貴(トウ・ヒロタカ)
基礎科学特別研究員 河本 尚大(カワモト・ナオヒロ)
東京大学 生産技術研究所
准教授 池内 与志穂(イケウチ・ヨシホ)
特任助教 周 小余(チャウ・シウユ)
報道担当
理化学研究所 広報室 報道担当
東京大学 生産技術研究所 広報室