2024-09-10 東京医科歯科大学
ポイント
- 一細胞トランスクリプトームデータから一細胞レベルで各遺伝子のスプライシング速度と分解速度の推定を行う手法、DeepKINETを開発。
- RNA結合タンパク質の機能やスプライシング因子の変異の影響の推定が可能。
- 遺伝子発現制御メカニズムの解明により、創薬標的の探索や治療法開発への応用が期待。
名古屋大学医学部附属病院卒後臨床研修・キャリア形成支援センターの水越周良研修医、国立がん研究センター研究所計算生命科学ユニットの小嶋泰弘独立ユニット長(東京医科歯科大学難治疾患研究所計算システム生物学分野連携研究員)、東京医科歯科大学難治疾患研究所計算システム生物学分野/名古屋大学大学院医学系研究科システム生物学分野の島村徹平教授らの研究グループは、各遺伝子の代謝を解析するための新規の情報解析手法「DeepKINET」を開発しました。
この手法は、一細胞トランスクリプトームデータと、RNA速度モデル、深層生成モデルの枠組みを利用して、遺伝子のスプライシングと分解の速度を一細胞解像度で分析することを可能にする技術です。本解析手法を神経細胞、乳癌、骨髄異形成症状群のデータに適用し、各遺伝子のmRNAのスプライシングや分解を網羅的に解析することにより、RNA結合タンパク質の機能や、スプライシング因子の変異の影響の推定を行いました。
mRNAのスプライシングや分解の理解は、遺伝子発現制御メカニズムの解明において不可欠であり、特にがんの発生や進行について新たな知見を提供します。DeepKINETは、一細胞トランスクリプトームデータを用いて、一細胞レベルでmRNAの動態を解析することに成功しました。本解析手法は、遺伝子発現制御の分子メカニズムに関する網羅的なデータに基づく仮説の提案を可能にし、新規の治療標的の探索に役立つと期待されます。
本研究成果は、国際学術誌Genome Biologyに2024年9月6日にオンライン掲載されました。
研究の背景
近年、シングルセルRNAシークエンス※1の登場により、疾患の根底にある細胞の不均一性の理解が進んでいます。この技術では、網羅的に計測される個々の細胞のRNAプロファイルを基にして、生体組織内の細胞タイプの分類だけでなく、より細かな個々の細胞の状態を調べることができます。さらにRNA速度モデル※2においてはスプライシングされていないRNAとスプライシングされているRNAを別々に定量し、数理モデルに当てはめることで、RNAの変化速度や細胞の分化方向の推定を行うことができます。一方で従来のRNA速度モデルはスプライシングや分解の速度を全ての細胞で一定と仮定しており、細胞間の真の生物学的変動を捉えられないという課題がありました。また、mRNAのスプライシングと分解は、遺伝子発現の精密な制御において重要な役割を果たしており、これらのプロセスの異常はがんなどの疾患の発症や進行と密接に関連しています。しかし、これまでRNA速度モデルを用いて推定されるスプライシング速度や分解速度を用いた詳細な生物学的解析は十分に行われていませんでした。
研究成果の概要
本研究において、一細胞レベルでのmRNAのスプライシング速度と分解速度の推定を実現し、一細胞レベルでの遺伝子発現制御を解明するための革新的な手法である「DeepKINET」を開発しました。DeepKINETは、一細胞トランスクリプトームデータから得られる詳細な分子プロファイルと、RNA速度モデルを組み合わせ、各遺伝子のスプライシング速度と分解速度を明らかにする深層生成モデル※3です。このモデルは、ニューラルネットワークを用いて、潜在的な細胞状態からRNA速度モデルを満たすようにスプライシングと分解の速度を学習します。人工的なデータセットへの適用では、推定されたスプライシングと分解の速度が設定値と高い相関性を示し、推定の正確性を確認しました。さらに、mRNAにラベリングを行った実験のデータセットへ適用し、実際の生物データにおける推定の正確性を確認しました。さらにDeepKINETは、神経細胞においてスプライシング速度と分解速度を用いた遺伝子の分類を行い、特定のRNA結合タンパク質がターゲット遺伝子のmRNAのスプライシングまたは分解を制御することを示しました。乳癌データセットにおける解析では、がんの転移に関与するスプライシングや分解の異常が明らかになりました。骨髄異形成症候群のデータセットにおいては、スプライシング因子の変異が標的に与える影響を分析しました。
研究成果の意義
一細胞レベルでのスプライシングおよび分解速度の推定は、遺伝子発現制御メカニズムの解明と、がんをはじめとする疾患における新たな治療標的の発見に重要な役割を果たします。DeepKINETは一般的に入手可能な一細胞トランスクリプトームデータを利用できるため、将来的には、この手法を様々な細胞集団に適用することで、幅広い疾患における遺伝子発現の調節機構が解明されることが期待されます。今後、DeepKINETはさらなる改良が加えられ、転写速度の推定や異なるmRNAアイソフォームの区別など、より複雑な解析にも対応できるよう発展させていく予定です。これにより、遺伝子発現制御の複雑なネットワークや、細胞間・集団間の遺伝子発現の変動について、より包括的な全体像が明らかになるでしょう。このような進歩は、生物学の深い理解だけでなく、がんを含む様々な疾患の新たな治療戦略の開発にも大きく貢献し、医学分野における画期的な発見につながる可能性を秘めています。
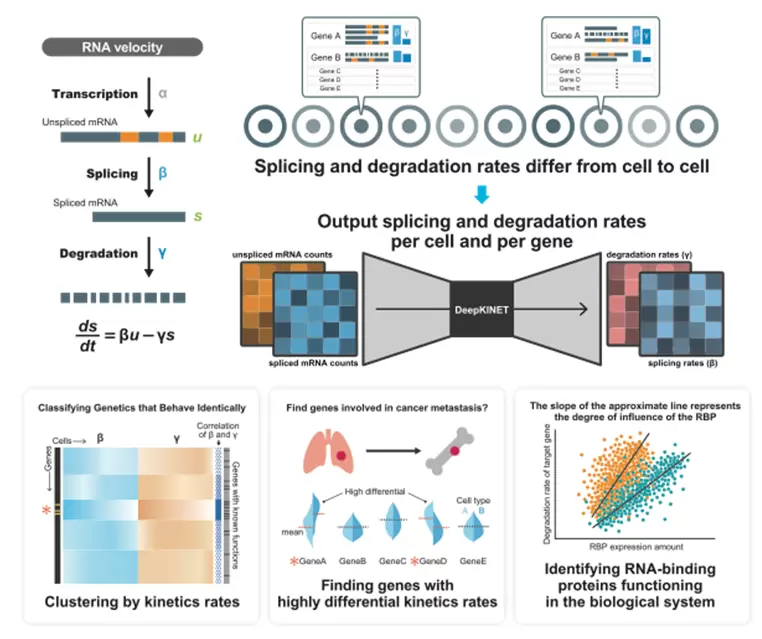
図1 DeepKINETの概要
用語解説
※1シングルセルRNAシークエンス・・・・・一細胞ごとの区別をつけた状態でRNA分子の網羅的なシークエンシングを行うことにより、細胞ごとの遺伝子発現量の網羅的な定量化を行う手法。
※2RNA速度モデル・・・・・シングルセルRNAシークエンスデータを用いて、細胞の状態変化や分化の方向性を予測するための手法。細胞内のRNAの動態、特に転写、スプライシング、分解過程に着目している。
※3深層生成モデル・・・・・深層学習によりデータの生成過程を記述した確率モデル。近年、一細胞トランスクリプトーム解析への応用が進んでいる。
研究助成
本研究は、日本学術振興会(JSPS)(課題番号20H04281、20K22839、22H04839、22H04925、23H04938c)、日本医療研究開発機構(AMED)(課題番号JP22ama221215、JP22ama221501、JP22ek0109488、JP23wm0325068、JP22wm0425007、JP23tm0424226)、科学技術振興機構(JST) ムーンショット型研究開発事業(目標2)(課題番号JPMJMS2025)および戦略的創造研究推進事業(課題番号JPMJAX20AB)、産業技術総合研究所(AIST) AI橋渡しクラウド(ABCI)、東京工業大学のスーパーコンピュータシステムTSUBAME、東京大学ヒトゲノム解析センターのスーパーコンピュータシステムSHIROKANE等の支援を受けて行いました。
論文情報
掲載誌:Genome Biology
論文タイトル:DeepKINET: a deep generative model for estimating single-cell RNA splicing and degradation rates
DOI:https://doi.org/10.1186/s13059-024-03367-8
研究者プロフィール
水越 周良 (ミズコシ チカラ) Mizukoshi Chikara
名古屋大学医学部附属病院
卒後臨床研修・キャリア形成支援センター 研修医
・研究領域
シングルセルオミクス、バイオインフォマティクス、人工知能
小嶋 泰弘 (コジマ ヤスヒロ) Kojima Yasuhiro
東京医科歯科大学 難治疾患研究所
計算システム生物学分野 連携研究員
国立がん研究センター研究所
計算生命科学ユニット 独立ユニット長
・研究領域
バイオインフォマティクス、データ科学、人工知能
島村 徹平 (シマムラ テッペイ) Shimamura Teppei
東京医科歯科大学 難治疾患研究所
計算システム生物学分野 教授
名古屋大学 大学院医学系研究科
システム生物学分野 特任教授
・研究領域
システム生物学、バイオインフォマティクス、データ科学、人工知能
問い合わせ先
<研究に関すること>
東京医科歯科大学 難治疾患研究所
計算システム生物学分野 島村 徹平(シマムラ テッペイ)
名古屋大学 大学院医学系研究科
システム生物学分野 島村 徹平(シマムラ テッペイ)
<報道に関すること>
東京医科歯科大学 総務部総務秘書課広報係
名古屋大学医学部・医学系研究科 総務課総務係