2024-09-13 がん研究センター,理化学研究所
発表のポイント
- 肺腺がんのサブタイプ、なかでも、分子標的治療の対象外である症例について、全ゲノムデータ、エピゲノムデータ(クロマチン免疫沈降解析シークエンスおよびDNAメチル化)、トランスクリプトームデータを活用して、統合解析を行いました。
- 治療標的となる遺伝子変異(ドライバー変異)が見つからない肺腺がん症例において、MAML2遺伝子の発現が有意に低下していることが明らかとなりました。
- MAML2遺伝子との相関解析から、患者予後予測に関連するマーカー遺伝子を同定しました。
- 今回の発見は、肺腺がんの新たな予後マーカーとして創薬に応用できる可能性を示唆するとともに、機械学習を用いることでより精緻に患者を層別化できることを示しました。
概要
国立研究開発法人国立がん研究センター(東京都中央区、理事長:中釜斉)研究所(所長:間野博行)医療AI研究開発分野の浅田健外来研究員(理化学研究所 革新知能統合研究センター・上級研究員)、浜本隆二分野長、ゲノム生物学分野の河野隆志分野長、国立がん研究センター中央病院、理化学研究所からなる共同研究チームは、Notchシグナル(注1)に関わることが知られているMAML2遺伝子(注2)が、分子標的薬の治療対象とならない肺腺がん症例(ドライバー遺伝子変異/転座陰性症例(注3))において、発現が低下していることを見出しました。
研究グループはH3K27Ac(注4)と呼ばれるヒストン修飾を検出するクロマチン免疫沈降シークエンス(ChIP-seq)(注5)解析などのデータを活用して、マルチオミックス解析(注6)を行いました。その結果、腫瘍部位ではMAML2遺伝子の発現抑制と、MAML2遺伝子領域におけるゲノム変異の蓄積、H3K27Ac修飾の低下および高DNAメチル化が起こっていることを見出しました。本研究により、ドライバー変異(注7)が見つかっていない肺腺がん症例の発がんメカニズムと予後予測に関連するマーカー遺伝子が明らかとなったことから、分子標的治療のない肺腺がんでの創薬開発につながることが期待されます。
この研究成果は、国際学術雑誌「Molecular Cancer」オンライン版(9月2日付)に掲載されました。
背景
日本における死因の第一位は悪性腫瘍であり、その中でも肺がんは部位別死亡数の第一位です。肺腺がんは肺がんの中でも最も発症頻度が高く、EGFR、KRAS、ALKなどの遺伝子に変異が生じることが知られています。これらのドライバー変異を調べることは、分子標的薬を用いた治療法の選択において重要ですが、残念ながら全ての症例で変異が見つかるわけでありません。約30%の症例ではドライバー変異が見つからないため、分子標的薬を用いることができません。さらに、肺腺がんには人種差があることが知られており、日本人の肺腺がん研究には、日本人のデータを利用した統合的な解析が求められています。
近年、次世代シーケンシング(注8)の普及によりゲノム・エピゲノム・トランスクリプトームといった様々なオミックスデータが収集され、解析がなされています。当研究では、日本人の肺腺がん症例を対象に、ドライバー変異が見つかっていない症例に着目して、全ゲノム解析とChIP-seq解析から、ゲノム構造異常とスーパーエンハンサー形成によるHER2遺伝子の発現上昇を報告しています*。本研究では引き続きドライバー変異が未同定の症例を対象として、ゲノム・エピゲノム・トランスクリプトームを含む複数のオミックスデータを活用した統合マルチオミックス解析とクラスター分析を行いました。
* 2024年7月2日プレスリリース
肺腺がんに新たな治療標的となる遺伝子を発見 統合的な全ゲノムシークエンスにより、肺腺がんの個別化医療の発展に寄与
<https://www.ncc.go.jp/jp/information/pr_release/2024/0702/index.html>
研究成果
1.ドライバー変異未同定の肺腺がんで転写に関与する遺伝子の発現低下を発見
ドライバー変異が確認されていない肺腺がん症例において、腫瘍部位と非腫瘍部位のサンプルを用いて解析を行ったところ、腫瘍部位では、転写に関与するとされるMAML2遺伝子の発現が低下していることが明らかとなりました。MAML2は粘表皮がん(Mucoepidermoid Carcinoma)において、CRTC1との融合遺伝子がドライバー変異として知られています。また、MAML2遺伝子は種を超えて保存されており、Notchシグナル下流の遺伝子発現を制御することが報告されています。本研究ではマルチオミックス解析から、MAML2の遺伝子領域においてゲノム変異が認められ、同時にエンハンサー活性の低下と高DNAメチル化が見出されました(図1)。
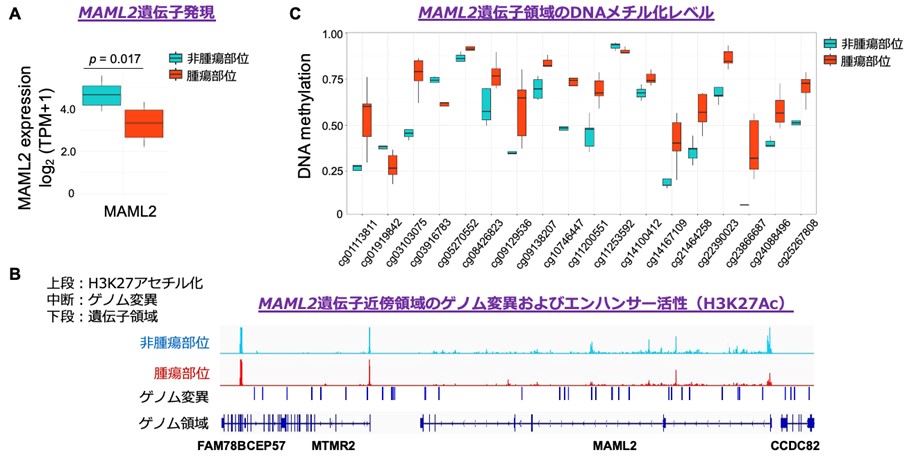
図1:MAML2遺伝子発現の低下とオミックス解析結果
(A) 非腫瘍部位と腫瘍部位におけるMAML2遺伝子発現。腫瘍部位ではMAML2遺伝子の発現が低下している。
(B) MAML2遺伝子とその近傍領域における非腫瘍部位のヒストン修飾(上段:水色)と腫瘍部位におけるヒストン修飾(上段:赤色)、ゲノム変異(中段:青色)、および解析対象としたMAML2遺伝子近傍領域(下段:青色)。
腫瘍部位でエンハンサー活性の低下とゲノム変異の蓄積が明らかとなった。
(C) MAML2遺伝子領域のDNAメチル化。非腫瘍部位と腫瘍部位で有意にメチル化レベルに差があった箇所を図示。
2.相関解析による予後予測マーカーの同定
MAML2遺伝子と他遺伝子との相関解析から、発現の相関が高かった上位15個の遺伝子を抽出し、発現レベルに応じて高発現・低発現の2群にわけて生存時間分析を行ったところ、FAT4、 HMCN1、 CD302、 UTRN、 FOXN3の5つの遺伝子が予後予測のバイオマーカーであることが明らかとなりました。今後、同定した予後バイオマーカーが関わるシグナルパスウェイを詳細に解析することで、ドライバー変異が見つかっていない日本人肺腺がん症例の生存に関わる機序解明が期待されます。
3.機械学習を活用したより精緻な患者層別化
5つのマーカー遺伝子ごとの生存時間分析の結果を元に、予後良好群と予後不良群に応じてラベルをつけて、機械学習に利用できるデータを作成し、クラスター分析を行いました。CD302、FAT4、FOXN3の生存時間分析結果を活用した解析により、さらに精緻に患者層別化できることが明らかとなりました(図2)。また、予後良好群と予後不良群との間で遺伝子の発現変動解析(注9)を行うことで、有意に発現が上昇している遺伝子および低下している遺伝子を明らかにしました。これらの遺伝子は、将来的な創薬標的になる可能性があると考えられます。
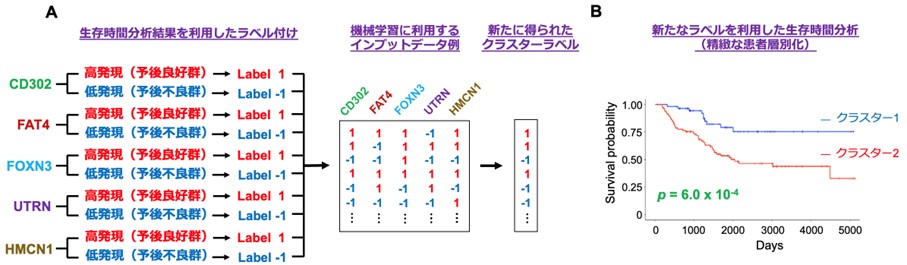
図2:生存時間分析結果と機械学習を活用した精緻な患者層別化
(A) 予後バイオマーカーの生存時間分析結果を利用したラベル付けとクラスター分析に用いるインプットデータの作成(左側および真ん中)。
クラスター分析後に得られた新規クラスターラベル(右側)。(B) (A)で得られたクラスターラベルを利用した生存時間分析の結果。
展望
本研究により、ドライバー変異が見つかっていない日本人の肺腺がん症例において、発がんに関係する新たな分子メカニズムが解明され、今後の創薬への可能性が見出されました。また、機械学習を活用した解析により、精緻な患者層別化ができることも明らかとなりました。これらの研究成果は今後の肺がん研究の進展に寄与するとともに、新たな創薬標的探索の基盤となることが期待されます。
発表論文
雑誌名
Molecular Cancer
タイトル
Multi-omics and clustering analyses reveal the mechanisms underlying unmet needs for patients with lung adenocarcinoma and identify potential therapeutic targets
著者
Ken Asada (* Corresponding Author), Syuzo Kaneko, Ken Takasawa, Kouya Shiraishi, Norio Shinkai, Yoko Shimada, Satoshi Takahashi, Hidenori Machino, Kazuma Kobayashi, Amina Bolatkan, Masaaki Komatsu, Masayoshi Yamada, Mototaka Miyake, Hirokazu Watanabe, Akiko Tateishi, Takaaki Mizuno, Yu Okubo, Masami Mukai, Tatsuya Yoshida, Yukihiro Yoshida8 Hidehito Horinouchi, Shun-Ichi Watanabe, Yuichiro Ohe, Yasushi Yatabe, Takashi Kohno (* Corresponding Author), Ryuji Hamamoto (* Corresponding Author)
掲載日
2024年9月2日付 オンライン・プレ・リリース
DOI
10.1186/s12943-024-02093-w
URL https://molecular-cancer.biomedcentral.com/articles/10.1186/s12943-024-02093-w(外部サイトにリンクします)
研究費
- 科学技術振興機構(JST)・AIPプロジェクト(AIP-PRISM)「人工知能技術を活用した革新的ながん創薬システムの開発」(研究代表者名:浜本隆二)
- 日本医療研究開発機構(AMED)・革新的がん医療実用化研究事業「機械学習/深層学習技術を活用したpan-negative肺がん症例のバイオマーカー探索と臨床的有用性の検証」(研究代表者名:浜本隆二)
- 内閣府科学技術・イノベーション推進事務局 研究開発とSociety5.0との橋渡しプログラム(BRIDGE)「医療デジタルツインの発展に資するデジタル医療データバンク構想」(研究総括:浜本隆二)
- 日本学術振興会・科学研究費助成事業 基盤研究(C)(一般)「エンハンサー領域に着目した肺がんpan-negative症例の統合オミックス解析」(研究代表者名:浅田健)
- 武田科学振興財団・医学系研究助成「Pan-negative肺がん症例における特異的なsignature探索とマルチオミックス解析を用いた分子メカニズムの解明」(研究代表者名:浅田健)
発表者
国立がん研究センター
研究所 医療AI研究開発分野:金子修三、小林和馬、浜本隆二(責任著者)
ゲノム生物学研究分野:白石航也、島田陽子、河野隆志(責任著者)
中央病院 呼吸器内科:立石晶子、水野孝昭、吉田達哉、堀之内秀仁、大江裕一郎
呼吸器外科:大久保祐、吉田幸弘、渡辺俊一
放射線診断科:三宅基隆、渡辺裕一
病理診断科:谷田部恭
医療情報部:向井まさみ
内視鏡科:山田真善
理化学研究所革新知能統合研究センター
がん探索医療研究チーム:浅田健(筆頭著者、責任著者)、高澤建(研究当時)、新海典夫、高橋慧、町野英徳、Bolatkan Amina、小松正明、浜本隆二(併任)
用語解説
注1 Notchシグナル
膜タンパク質のNotchは受容体として接触した細胞間でのシグナル伝達に関わり、細胞分化や細胞増殖、幹細胞維持など、多くの細胞プロセスにおいて重要な役割を担っています。そのため、Notchシグナルとその調節因子はがん化をはじめ疾患の発症に関連することが知られています。
注2 MAML2遺伝子
種を超えて保存されているMastermind Like Transcriptional Coactivator 2遺伝子のことで、ヒトではMAML1、2、3のファミリーが知られています。
注3 ドライバー遺伝子変異/転座陰性症例
pan-negative症例とも呼ばれ、臨床的に有効な遺伝子異常(CAGA: Clinically Actionable Genetic Alteration)を持たない肺腺がんのサブタイプを指します。CAGAとは特徴が異なり、従来の治療法が適用できないため、別の治療法が必要となる場合があります。このため、ドライバー遺伝子変異/転座陰性である肺腺がん症例に対しては、新たな治療標的の発見や個別化医療の展開が求められています。
注4 H3K27Ac
遺伝子の転写開始点付近を中心にみられる、ヒストンH3の27番目のリジン残基がアセチル化された修飾です。塩基性アミノ酸のリジンが酸性のアセチル基と結合することにより、ヒストンの塩基性が弱まります。その結果、ヒストンとDNAとの間の相互作用が弱まるために、高次構造の変化につながります。つまりこの修飾は活性化したエンハンサー領域を示し、遺伝子の転写活性を示すマーカーとして解析に用いられます。
注5 ChIP-seq
Chromatin Immunoprecipitation Sequencingの略。クロマチン免疫沈降法と次世代シーケンシング(注7)を組み合わせた手法で、エピゲノム解析の一つとして知られています。DNAと結合しているタンパク質の結合部位を全ゲノムスケールで解析する技術です。
注6 マルチオミックス解析
オミックス(Omics)とはゲノム(genome)、エピゲノム(epigenome)、トランスクリプトーム(transcriptome)、プロテオーム(proteome)、メタボローム(metabolome)などのように生体分子に関連したデータ(情報)を意味します。これらオミックスデータを複数(マルチ)活用した解析をマルチオミックス解析と呼びます。
注7 ドライバー変異
がんの発生や進行に直接関与する遺伝子をドライバー遺伝子と呼び、その変異をドライバー変異といいます。変異はがん細胞の成長や生存に必要な変化です。ドライバー遺伝子に変異があるがんでは、ドライバー遺伝子を標的とした治療が有効です。
注8 次世代シーケンシング
Next-Generation Sequencing(NGS)。DNA配列を一つずつ解読するサンガーシーケンスの次の技術との位置付けから、次世代と呼ばれています。“次世代”の技術は常にアップデートしていますが、定義としては一度に多くのDNA配列を高速に解読できるハイスループット技術を指し、現在の医学・分子生物学研究に必須な技術の一つです。
注9 発現変動解析
異なる条件(投与薬剤量の違いなど)や、グループ間(非投与群・プラセボ群と投与群など)において遺伝子の発現量が大きく上昇もしくは減少した遺伝子を見つけ出す解析手法です。抽出された遺伝子をもとに、どのようなパスウェイが活性化しているかのパスウェイ解析もあわせて行うことが多く、観察された事象のより深い理解(分子メカニズム解明)につながります。
問い合わせ先
研究に関する問い合わせ
国立研究開発法人国立がん研究センター
研究所 医療AI研究開発分野
分野長 浜本 隆二
理化学研究所 革新知能統合研究センター がん探索医療研究チーム
上級研究員 浅田健
広報窓口
国立研究開発法人国立がん研究センター
企画戦略局 広報企画室
理化学研究所 広報室 報道担当