2025-10-22 産業技術総合研究所
データ拡張によりタンパク質の機能値予測の精度を向上
<関連情報>
- https://www.aist.go.jp/aist_j/press_release/pr2025/pr20251022/pr20251022.html
- https://academic.oup.com/bib/article/26/5/bbaf536/8280447
分子シミュレーションとタンパク質言語モデルによる弱監視によるデータ効率の高いタンパク質変異効果予測 Data-efficient protein mutational effect prediction with weak supervision by molecular simulation and protein language models
Teppei Deguchi, Nur Syatila Ab Ghani, Yoichi Kurumida, Shinji Iida, Kaito Kobayashi, Yutaka Saito
Briefings in Bioinformatics Published:10 October 2025
DOI:https://doi.org/10.1093/bib/bbaf536
Abstract
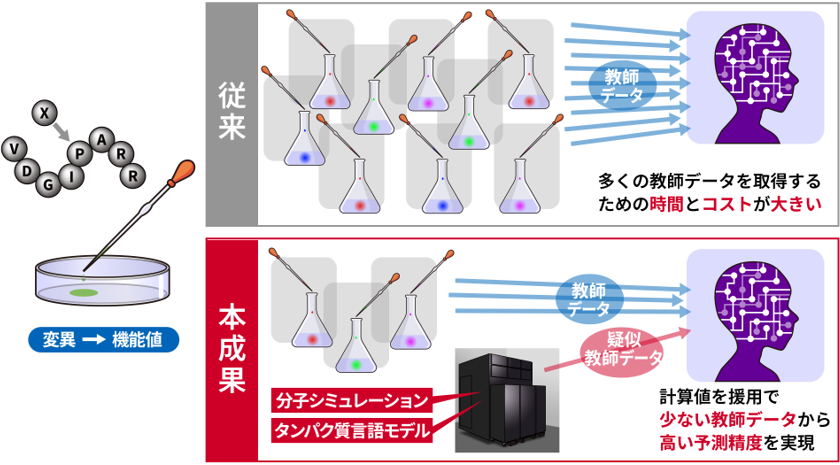
Machine learning-based protein mutational effect prediction is widely used in protein engineering and pathogenicity prediction, but training data scarcity remains a major challenge due to high costs of experimental measurements. A previous study proposed data augmentation using computational estimates by molecular simulation. However, this approach has been limited to predicting mutational effects on thermostability. Here, we present a new data augmentation method that combines molecular simulation with zero-shot prediction computed by protein language models. These computational estimates serve as ‘weak’ training data to supplement experimental training data. Our method dynamically adjusts the weight and inclusion of weak training data based on available experimental training data. This reduces potential negative impacts of weak training data while extending applicability to diverse protein properties such as binding affinity and enzymatic activity. Benchmark tests demonstrate that our method improves prediction accuracy particularly when experimental training data are scarce. These results indicate the capability of our approach to advance protein engineering and pathogenicity prediction in small data regimes.