同法を活用し日本人感染者約100例の網羅的ウイルス情報を取得
2019-10-16 熊本大学,日本医療研究開発機構
ポイント
- ヒトT細胞白血病ウイルス(HTLV-1)は、我が国に約80万人の感染者が存在し、感染者の一部にがんや慢性炎症性疾患を引き起こすことが知られています。
- HTLV-1にはウイルスが感染したヒト細胞のDNAにウイルスDNAを組み込ませる特性があり、それがウイルス病原性の起点となっています。
- 今回、ウイルスに対するDNAプローブ (注1)と次世代シークエンサー (注2)を活用することで、網羅的かつ高精度に感染細胞DNAに存在するウイルス情報を取得する、新しい方法を確立しました。
- さらに本研究では、同法を活用した日本人の感染者約100人の網羅的なウイルス情報を取得しました。
- 本研究によって、今後のHTLV-1研究推進や関連疾患の診断向上に資する情報が得られました。
- 今回の研究方法はHTLV-1感染症に限らず、他のがんに関連するウイルス感染症などへの今後の更なる応用展開が期待されます。
説明
ヒトレトロウイルス学共同研究センター熊本大学キャンパスの佐藤賢文教授は、佐賀大学 勝屋弘雄助教、鹿児島大学 宇都宮與客員教授、聖マリアンナ医科大学 山野嘉久教授らと共同で、ヒトT細胞白血病ウイルス(HTLV-1)に感染した細胞DNAに存在するウイルス情報を網羅的かつ高精度に取得する、新しい解析方法を確立しました。
HTLV-1は、我が国に約80万人の感染者が存在し、感染者の一部にがんや慢性炎症性疾患を引き起こすことが知られています。HTLV-1には感染した細胞DNAにウイルスDNAを組み込ませる特性があります。感染した細胞DNAに組み込まれたウイルスは、病原性発現の起点となることから、組み込まれたウイルスDNAに関する情報は、そのウイルスに関連した病気の診断やメカニズム解明の研究において極めて重要です。
従来、HTLV-1感染者におけるウイルス感染病態を高い精度で把握するためには、ウイルスDNAの全長塩基配列(欠損型ウイルスの有無)、ウイルスDNAの構造、ウイルスDNAの組み込み部位、感染した細胞の増殖度などの情報を取得するために、複数の解析法を組み合わせて行う必要がありました。本研究グループは、ウイルスに対するDNAプローブと次世代シークエンサーを活用した新手法で、それらのウイルス情報を網羅的かつ高精度に解析することが可能になることを見出しました(図1)。
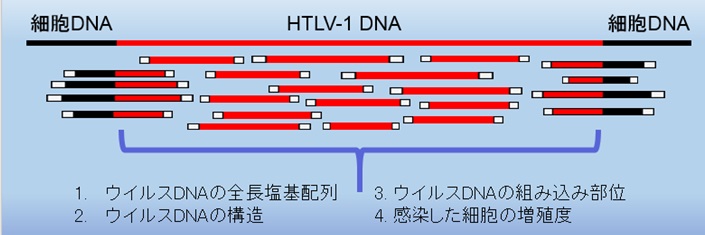
図1:HTLV-1 を網羅的かつ高精度に解析する新手法
さらにこの新手法を用いて、日本人の感染者約100人の網羅的かつ詳細なウイルス情報の取得を行いました(図2)。そのウイルス情報は、論文としてオープンアクセスジャーナルで広く公開されるとともに、公的なデータベースに登録しその情報が広く発信され、今後のHTLV-1研究推進や関連疾患の診断向上に資することが期待されます。
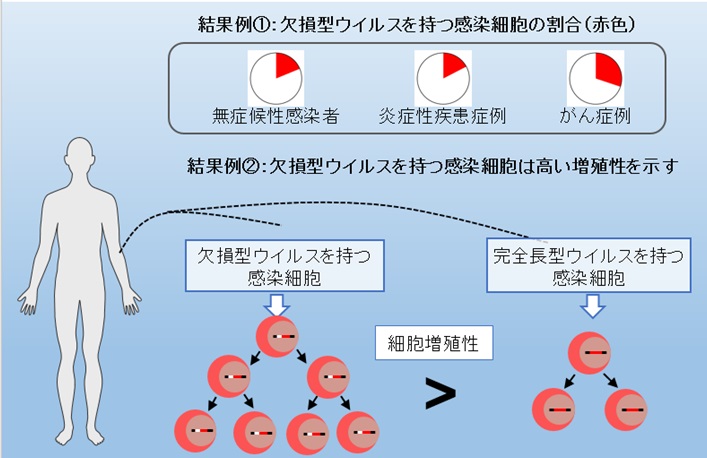
図2:新解析法により取得されたウイルス情報の結果例
また、ウイルスに対するDNAプローブと次世代シークエンサーを活用した本研究手法はHTLV-1感染症に限らず、他のがんに関わるウイルス感染症にも応用できる可能性があり、今後のさらなる展開が期待されます。
本研究は、日本医療研究開発機構(AMED)感染症研究革新イニシアティブ(J-PRIDE)「単一細胞分析データマイニングによるHTLV-1持続潜伏感染機構の解明」(研究開発代表者 佐藤賢文)の研究支援を受けて行ったものです。
本研究成果は、2019年10月15日午前11時(米国東部時間)発行の「Cell Reports」に掲載されます。
用語解説
- 注1:DNA プローブ
- DNAとはデオキシリボ核酸(deoxyribonucleic acid)の略称で、私たちヒトの生命活動の根源である遺伝子の情報が書き込まれています。DNAはA(アデニン)、T(チミン)、G(グアニン)、C(シトシン)という4つの塩基が長い鎖状に連なった分子で、通常、A-T, G-Cが水素結合で相補的に結合した二重らせん構造をしています。そのDNAがもつAとT、GとCが結合しやすいという特性を利用して、特定のDNA配列に結合するように設計されたプローブ(検出子)のこと。
- 注2:次世代シークエンサー
- 2000年代に入り開発されたDNAの塩基配列を高速かつ網羅的に読み出せる装置のことで、それまでの装置に比べDNAの解析速度を飛躍的に向上させたことで知られています。
論文情報
- 論文名:
- The nature of HTLV-1 provirus in naturally infected individuals analyzed by viral DNA-capture-seq approach
- 著者:
- Hiroo Katsuya, Saiful Islam, Benjy Jek Yang Tan, Jumpei Ito, Paola Miyazato, Misaki Matsuo, Yuki Inada, Saori C Iwase, Yoshikazu Uchiyama, Hiroyuki Hata, Tomoo Sato, Naoko Yagishita, Natsumi Araya, Takaharu Ueno, Kisato Nosaka, Masahito Tokunaga, Makoto Yamagishi, Toshiki Watanabe, Kaoru Uchimaru, Jun-ichi Fujisawa, Atae Utsunomiya, Yoshihisa Yamano, Yorifumi Satou
- 掲載誌:
- Cell Reports
お問い合わせ先
研究に関すること
ヒトレトロウイルス学共同研究センター
熊本大学キャンパス
ゲノミクス・トランスクリプトミクス分野
教授 佐藤 賢文(さとう よりふみ)
報道に関すること
熊本大学総務部総務課広報戦略室
AMED事業に関すること
国立研究開発法人日本医療研究開発機構
戦略推進部 感染症研究課

