2021-07-27 国立遺伝学研究所
真核細胞内のクロマチンは、DNA、ヒストンと様々な関連タンパク質からなる、負に帯電した長いポリマーです。クロマチンは強く帯電していて不均一なため、その構造は化学修飾やタンパク質の量などの様々な因子や、陽イオンなどの周囲の環境に応じて、10 nm線維から折りたたまれた30 nm線維、凝集体・液滴まで大きく変化します。近年の一分子ヌクレオソームイメージング(Figure 1A)など、イメージング技術の進歩によって、クロマチンが細胞内でダイナミックな液体のように振る舞い、構造を変化させることがわかってきました(Figure 1B)。また、近年のコンピュータモデリングでは、多数のヌクレオソームを原子レベルのシミュレーションから粗視化したクロマチン繊維モデルに至る幅広いスケールで扱うことによって、細胞内の液体のようなクロマチンの振る舞いを再構築することが可能となっています (Figure 1C)。さらに、イメージング技術のみでは不可能な、観察された振る舞いを引き起こす分子メカニズムを調べるための強力な技術となっています。本総説論文では、イメージングとモデリング研究の両方における新しい知見に基づいて、生細胞内のクロマチンのダイナミックな側面と、その機能との関係性について議論しました。ゲノムダイナミクス研究室の伊藤優志 日本学術振興会特別研究員(元遺伝研博士研究員)、南克彦 総研大生、前島一博 教授、英国・ケンブリッジ大学のEsmae J. Woods 大学院生、グループリーダーRosana Collepardo-Guevara 博士の共同成果です。
日本学術振興会 (JSPS) 及び文部科学省科研費 (19K23735, 20J00572, 20H05936, 21H02453)、武田科学振興財団、上原記念生命科学財団、国立遺伝学研究所博士研究員、JSPS特別研究員(PD)の支援を受けました。
また、7月26日、ゲノムダイナミクス研究室の前島一博 教授、飯田史織 総研大生はeLife誌にInsight 論文を掲載しました。クロマチンが細胞内で様々な機能を発揮するためには、クロマチンループドメインなどのクロマチン高次構造が重要です。このクロマチンループの形成はコヒーシンというリング状の分子複合体が担っていると考えられています (Figure 2 左)。現在、クロマチンループ形成のメカニズムは細胞生物学のとてもホットな話題となっており、コヒーシンがそのリングの中でDNAを押し出すloop extrusionと呼ばれるモデルが脚光を浴びています。また実際に、試験管内でコヒーシンが裸のDNAを押し出す様子も捉えられています。最近、英国フランシス・クリック研究所のFrank Uhlmann 博士らのグループは試験管内でコヒーシンがどのようにloop extrusionを引き起こし、ループを作るのかを発表しました (Figure 2下段)(Higashi et al. “A Brownian ratchet model for DNA loop extrusion by the cohesin complex”. eLife, 2021. DOI:10.7554/eLife.67530)。その論文の内容に基づいて、細胞内でのコヒーシンの振る舞いや、loop extrusionが細胞内で起こる可能性について考察しました。
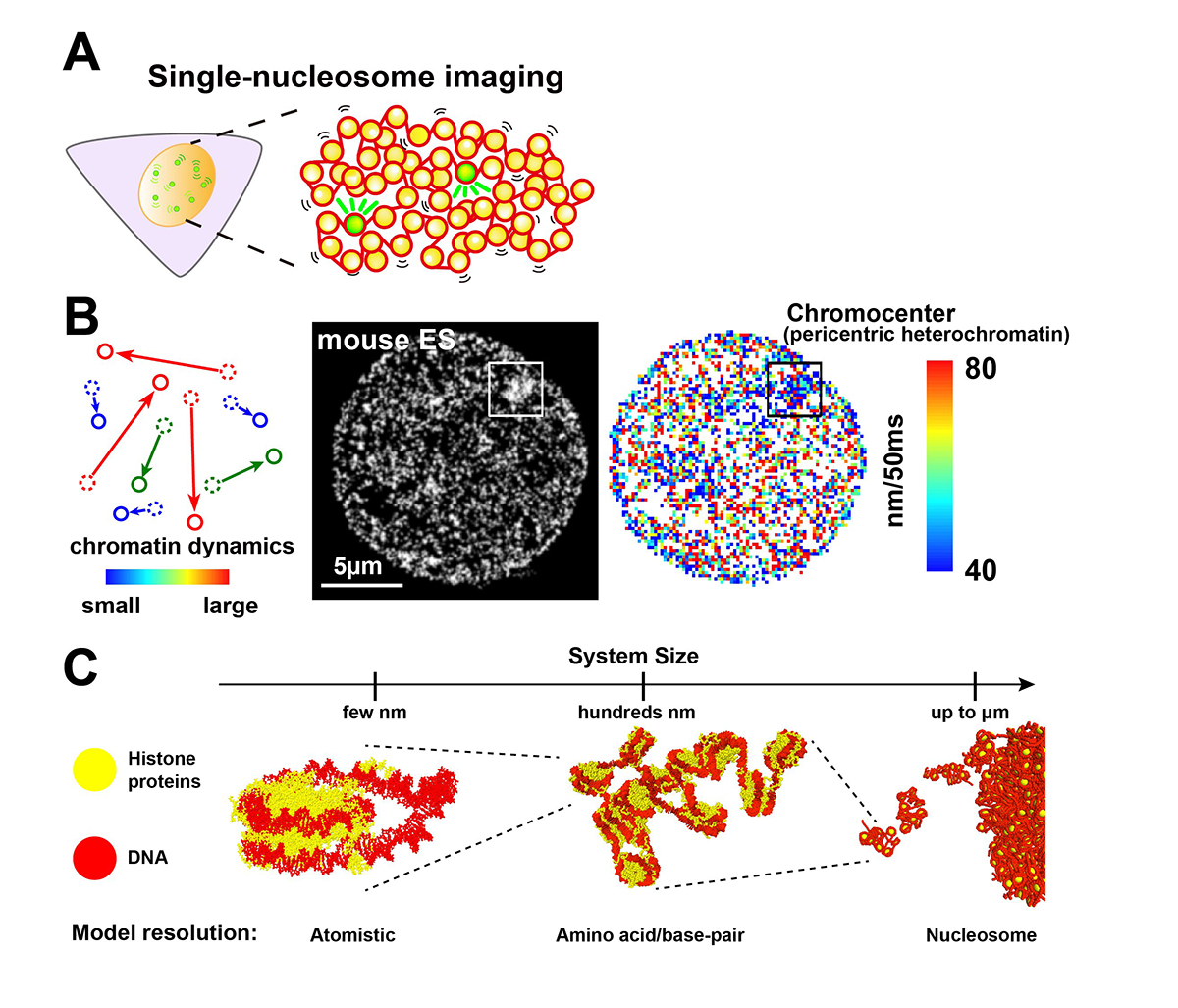
Figure1:(A,B)単一ヌクレオソームイメージング(A)をはじめとしたイメージング技術の進歩により、生きた細胞内において液体のようなクロマチンの振る舞いや、核内でのダイナミクスの多様性(B)を捉えられるようになった。 (C)近年の計算機シミュレーションでは、原子(左)・アミノ酸(中央)・ヌクレオソーム(右)と、幅広い階層を組み合わせたモデリングが可能となり、先端イメージングにより発見されたクロマチンの振る舞いを原子レベルの解像度で検証できる強力な手段となっている。
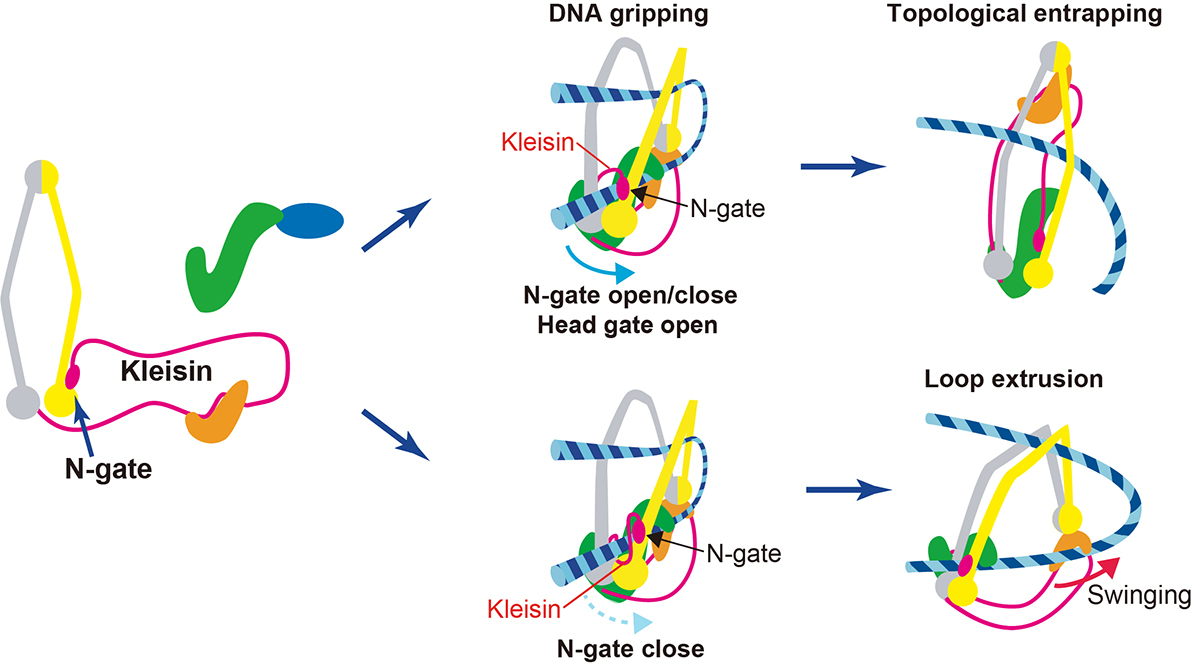
Figure2:Higashi et al. では、loop extrusionのメカニズムを、コヒーシンがDNAをリング構造のなかに入れる反応(図上、DNA捕捉)の“枝分かれ経路”として提唱した。(左)コヒーシンの構造。(中央、右)Kleisin N-gate が開くか開かないかでコヒーシンがDNAを捕捉するか、loop extrusionにはたらくかが決まる。(上)N-gateが開くときはコヒーシンはDNAを捕捉できる。(下)N-gateが開かないとコヒーシンはDNAを捕捉できず、ブラウン運動をつかったラチェットによってDNAループを押し出す。しかしながら、このメカニズムでは、高等真核細胞ではヌクレオソームの塊が立体障害となり、ループを作れない。
Liquid-like chromatin in the cell: What can we learn from imaging and computational modeling?
Yuji Itoh, Esmae J. Woods, Katsuhiko Minami, Kazuhiro Maeshima, and Rosana Collepardo-Guevara
Current Opinion in Structural Biology 71, 123-135 (2021) DOI:10.1016/j.sbi.2021.06.004
The loopy world of cohesin.
Kazuhiro Maeshima and Shiori Iida
eLife 10, e71585 (2021) DOI:10.7554/eLife.71585