「疎」なデータから遺伝子群に内包される正負の相関を推定する
2021-07-22 東京大学
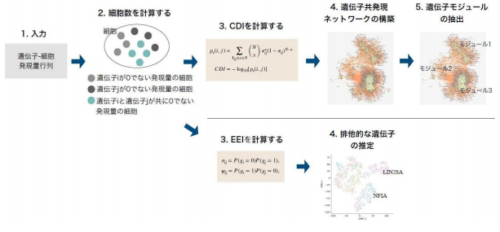
シングルセル遺伝子発現量解析(scRNA-seq)は、生体組織や腫瘍組織に含まれる細胞不均一性を同定するための強力な手法です。一方でデータ感度は限られており、ばらつきが多い「疎」なデータであるため、従来の遺伝子間相関・ネットワーク推定手法をそのまま適用することが困難でした。東京大学定量生命科学研究所の仲嶋 なつ 特任研究員、中戸 隆一郎 講師らの研究グループは、そのようなscRNA-seqデータに対して頑健に遺伝子共発現ネットワークを構築・比較する新規手法を開発しました。さらに、相互排他的な遺伝子の推定を行うための指標、Exclusively Expressed Index(EEI)を提案し、相互排他的な遺伝子ペア群を細胞クラスタリングのための特徴量として応用する手法を開発しました。
本手法を同研究所の林 寛敦 特任助教、秋山 徹 特任教授らの研究グループによって樹立された膠芽腫幹細胞データに適用し、血清刺激前後において共発現・排他発現パターンが変動する遺伝子群を網羅的に同定しました。これにより、従来法では見つけられなかった新規の膠芽腫幹細胞マーカー遺伝子候補を複数同定しました。本手法を活用することで、種々の組織細胞における新規マーカー遺伝子の同定や、細胞不均一性への理解の向上につながることが期待されます。
本研究の成果は、日本学術振興会 科研費 新学術領域研究「細胞社会ダイバーシティーの統合的解明と制御」(計画班代表者:中戸隆一郎、秋山徹、分担者:林寛敦)などの支援によって得られたものです。