2022-01-17 横浜市立大学,日本医療研究開発機構
概要
横浜市立大学大学院生命医科学研究科 池口 満徳教授、田辺三菱製薬株式会社、三井情報株式会社、理化学研究所、京都大学の共同研究グループは、機械学習を用いた新規のタンパク質結晶構造評価AI技術であるQAEmap(Quality Assessment based on Electron density map)を確立しました。
本研究では、公共データベース*1に登録されている高解像度の構造データを3D-CNN*2と呼ばれる3次元情報を扱う方法で機械学習することにより、データの解像度に依存しない構造評価ができることを示しました。この成果は、タンパク質構造を用いる創薬研究の加速化に貢献することが期待されます。
本研究成果は『Scientific Reports』に掲載されました。(2021年12月8日)
研究成果のポイント
- タンパク質の結晶構造をアミノ酸単位で評価できる新規のAIを確立(アミノ酸単位で機械学習することで、局所構造評価ができる。)。
- 低解像度の構造解析で、特に構造決定が難しいループ領域の構造決定に利用できる。
- 創薬研究での課題をアカデミア、IT企業、製薬会社が一体となって取り組んだ産学連携の成果。
研究背景
タンパク質の立体構造は、X線結晶構造解析あるいはクライオ電子顕微鏡法によって原子レベルの構造決定がなされますが、データの解像度が構造決定の確度に大きな影響を与えます。また、タンパク質の立体構造は、薬剤候補化合物と標的タンパク質との結合様式を見る創薬研究や分子シミュレーション研究の基盤となるため、特に低解像度での構造決定は長年の課題となっています。
研究内容
タンパク質の構造を構築するには、X線結晶構造解析用の電子密度マップやクライオ電子顕微鏡用のCoulombポテンシャルマップを実験データから計算し、そのマップに従ってタンパク質の原子を配置します。従って、タンパク質分子内のマップが明瞭な部分は構造決定が容易ですが、周囲の領域やループ領域、あるいは実験データの質が悪くマップの解像度が低い場合、原子の配置は実験者の主観や経験に大きく依存します。
本研究では、X線結晶構造解析の電子密度マップに基づく新しいタンパク質結晶構造評価の方法を確立し、QAEmapと名付けました。この方法では、Protein Data Bankに登録されている高解像度構造から作成した電子密度マップとタンパク質構造を入力データとし、タンパク質の正しい構造の電子密度マップとの相関を新たな評価指標(bCC)として定義し、これらを3D-CNNを用いて機械学習させることで、X線結晶構造解析の低解像度の課題の解決を目指しました。
QAEmapは、評価したいタンパク質構造の電子密度マップとそれに対応する座標を入力するとbCCを予測でき、より正しい構造を選ぶことができます(図1)。さらに、この方法はリガンド結合の評価に適用できる可能性があることから、低解像度で困難な化合物結合様式の評価にも適用できると考えられます。
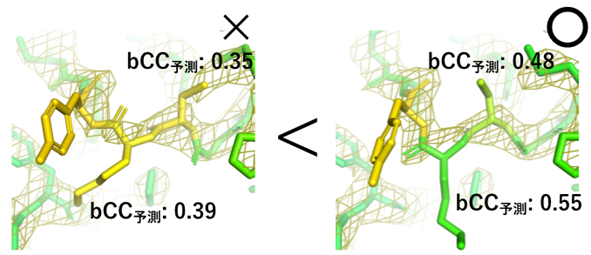
図1 ループ領域の構造決定において、各アミノ酸の構造の評価スコア(bCC:box Correlation Coefficient)をQAEmapにより予測することで、複数のモデルからより正しい構造を選択できる。
今後の展開
QAEmapは、タンパク質に結合する化合物の結合様式評価や、近年、創薬分野での利用が急速に広がっているクライオ電子顕微鏡を用いたタンパク質構造解析に応用可能で、タンパク質構造を用いる創薬研究の加速化に貢献すると期待しています。
従来の実験者の経験や技術に頼って行われてきたタンパク質の構造決定に、AIを取り入れた方法として今後の発展も期待されます。
QAEmapは以下で公開しています。
研究費
本研究は、LINCコンソーシアム*3の活動の一環として実施され、日本医療研究開発機構(AMED)の創薬等ライフサイエンス研究支援基盤事業(BINDS)「分子動力学計算による構造生物学データを活用した構造ダイナミクス研究」の支援を受けています。
用語説明
- *1 公共データベース
- Protein Data Bank(通称PDB。PDB: Homepage )
- *2 3D-CNN
- Three dimensional Convolutional Neural Network(3次元畳み込みニューラルネットワーク)の略。近年のAIの発展を促した技術の一つ。3D-CNNは、画像や動画の中から特徴パターンを見つける等、画像認識や動作検出、音声、信号データにも利用されている。本研究では3D-CNNをタンパク質の立体構造及び電子密度データをそのまま入力として深層学習するために用いている。
- *3 LINCコンソーシアム
- ライフインテリジェンスコンソーシアム(Life Intelligence Consortium; LINC)
ライフ系企業、IT企業、大学や研究機関など約70の企業・団体が参画し、医薬品開発などライフ系の課題解決やAI技術の開発に取り組む。2016年11月に発足し、2021年4月からは一般社団法人。
論文情報
- タイトル
- Machine learning to estimate the local quality of protein crystal structures
- 著者
- Ikuko Miyaguchi, Miwa Sato, Akiko Kashima, Hiroyuki Nakagawa, Yuichi Kokabu, Biao Ma, Shigeyuki Matsumoto, Atsushi Tokuhisa, Masateru Ohta, Mitsunori Ikeguchi
- 掲載雑誌
- Scientific Reports
- DOI
- 10.1038/s41598-021-02948-y
- URL
- www.nature.com/articles/s41598-021-02948-y
- 横浜市立大学のプレスリリース
- https://www.yokohama-cu.ac.jp/news/2021/202112ikeguchi_scirep.html
お問い合わせ先
研究に関するお問い合わせ先
池口 満徳(いけぐち みつのり)
横浜市立大学大学院生命医科学研究科 教授
取材に関するお問い合わせ先
横浜市立大学 広報課
AMED事業に関するお問い合わせ先
日本医療研究開発機構 創薬事業部 医薬品研究開発課
創薬等ライフサイエンス研究支援基盤事業(BINDS)