2022-06-28 理化学研究所
理化学研究所(理研)バイオリソース研究センター疾患ゲノム動態解析技術開発チームの杉本道彦開発研究員、阿部訓也チームリーダーらの共同研究グループは、1細胞遺伝子発現解析[1]において、複数の細胞サンプルをまとめた後、1回の実験操作で解析する「サンプル多重化」が、あらゆる細胞に対して効率よく行える新しい細胞標識手法を開発しました。
この技術を用いることで、1細胞遺伝子発現解析のコスト低減と高精度化につながるサンプル多重化を簡便かつ確実に行えるようになるため、本研究成果は1細胞遺伝子発現解析のさらなる普及に大きく貢献すると期待できます。
1細胞遺伝子発現解析では、サンプルを多重化することでバッチ効果[2]などのエラーの低減や実験コストの削減が可能になります。この際、あらかじめ各サンプルを固有のバーコードDNA[3]で標識し、各細胞がどのサンプル由来かを追跡できるようにします。しかし既存の手法では、細胞表面抗原に対する抗体を利用するため、発現している表面抗原の種類に依存することになり、細胞の種類によってはうまく標識できないという問題がありました。
今回、共同研究グループは、細胞表面タンパク質をビオチン[4]化することにより、ビオチン結合タンパク質であるストレプトアビジン[5]を介してバーコードDNAをどのような細胞にも結合させることのできる手法を開発し、「Universal Surface Biotinylation(USB)法」と名付けました。
本研究は、科学雑誌『DNA Research』オンライン版(6月16日付)に掲載されました。
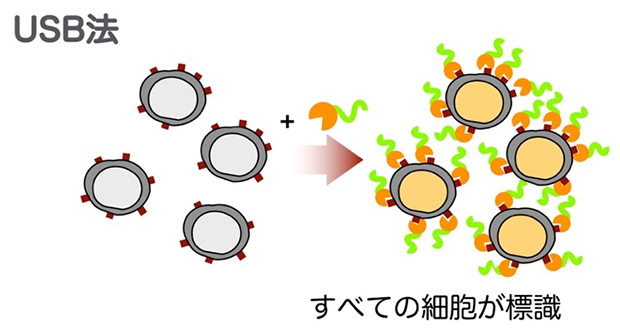
本研究で開発したサンプル多重化のための新しい細胞標識手法(USB法)の模式図
背景
複数種類の細胞から構成される組織や細胞集団の遺伝子発現解析をする上で、1細胞解析は極めて有効な手法です。1細胞での網羅的遺伝子発現解析を行うことで、より的確かつ詳細に集団内の細胞の状態を捉えることが可能になります。
近年、1細胞サンプル調製装置の進歩により、1検体当たり数万個単位の細胞を一挙に解析できるようになり、1細胞当たりの解析コストは大幅に削減することが可能になりました。しかし、多数の検体(例えば、複数の患者からの臨床検体や時系列に沿ったサンプル)を用いた比較解析を行う場合、それぞれの検体を異なる実験操作で処理するために生じるエラー(バッチ効果)の問題や、当然ながら検体の数に応じて解析費用が増大するコスト面の問題が生じます。
これらを解決するための手法の一つとして、「サンプル多重化」があります。この手法では、あらかじめ各細胞サンプルに固有のバーコードDNAを付加して標識した後、それらをひとまとめにして1細胞解析を行います。すると、各固有バーコードの配列を指標にして、それぞれの遺伝子発現データがどの検体由来なのかを追跡することができます(図1)。
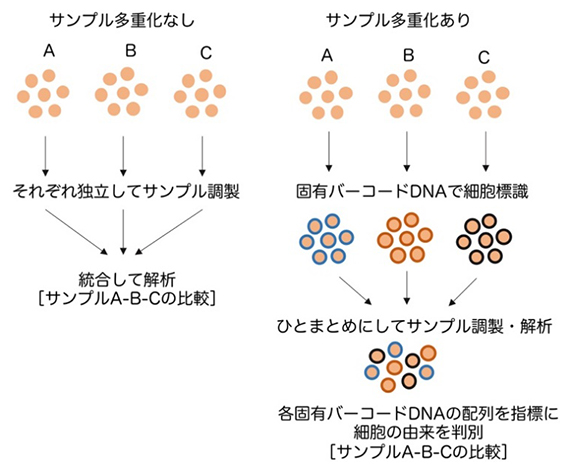
図1 1細胞遺伝子発現解析におけるサンプル多重化
複数サンプルを用いて1細胞遺伝子発現解析を行う場合、サンプル多重化を行うことで、サンプルをまとめて1サンプルとして調製できるようになる。
このように、複数の細胞集団サンプルを単一のサンプルとして解析できるため、サンプルごとの解析細胞数は減るものの、エラーやコストの低減が可能になります。しかし、既存のサンプル多重化法(細胞表面タンパク質に対する抗体を用いた手法:ここではAb法と呼ぶ)では、発現している表面タンパク質の種類に依存することになり、細胞の種類によっては適用できないという問題がありました。
研究手法と成果
共同研究グループは、あらゆる細胞に適用可能な、特定の細胞表面タンパク質に依存しない、サンプル多重化のための細胞標識法として「Universal Surface Biotinylation(USB)法」を新たに開発しました。この方法では、スルホ-NHS-ビオチン[6]という試薬を用いて、表面タンパク質のアミノ基にビオチンを付加します。この処理により、あらゆる表面タンパク質がビオチン化されるため、バーコードDNAを結合させたストレプトアビジン(ビオチンと特異的に結合する)を介して、全ての細胞にバーコードDNAを付加することができます(図2)。
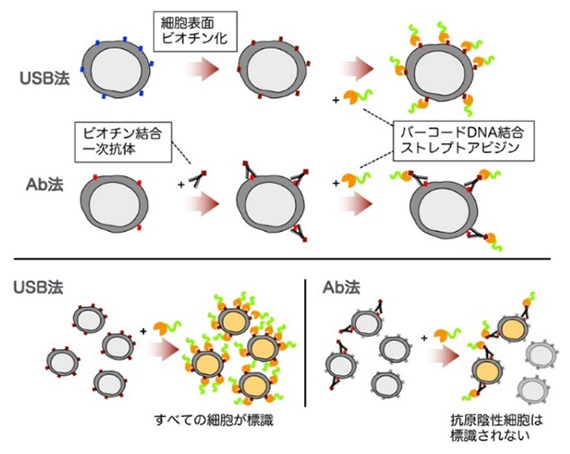
図2 既存のAb法と新規開発USB法によるサンプル標識の模式図
USB法では、細胞表面タンパク質を網羅的にビオチン化することができる。Ab法では抗体が認識する抗原タンパク質のみが標識に利用されるため、抗原タンパク質を発現していない細胞は解析することができない。
USB法の有効性を検証するために、分化とともに、表面タンパク質の種類が変動するマウスES細胞[7]を用いて、モデル実験を行いました。未分化状態にあるマウスES細胞では、細胞接着に関与するタンパク質CDH1が恒常的に発現していましたが、分化とともに減少し、CDH1はごく少数の分化ES細胞でしか発現しなくなりました(図3)。
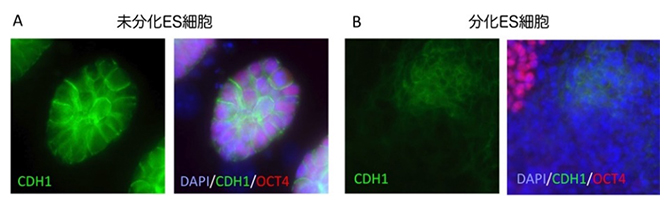
図3 マウスES細胞におけるCDH1タンパク質の発現
(A)未分化ES細胞におけるCDH1の発現画像、(B)分化ES細胞におけるCDH1の発現画像
(左:CDH1の発現画像、右:CDH1、OCT4、DAPIの発現の重ね合わせ画像、緑=CDH1、赤=OCT4、青=核)未分化ES細胞では全ての細胞でCDH1タンパク質が発現しているが、分化ES細胞では一部の細胞でのみ発現する。
Ab法(CDH1に対する抗体を使用)を用いて、未分化ES細胞と分化ES細胞で標識効率を調べてみると、未分化ES細胞では約90%の細胞がCDH1陽性でしたが、分化細胞では逆に90%が陰性でした。一方、同じサンプルをUSB法で標識すると、期待どおり未分化ES細胞でも分化ES細胞でもほぼ全ての細胞が標識されることが確認できました(図4)。また、USB法の実験操作により、細胞生存率や増殖への短期的あるいは長期的影響は見られませんでした(図5)。
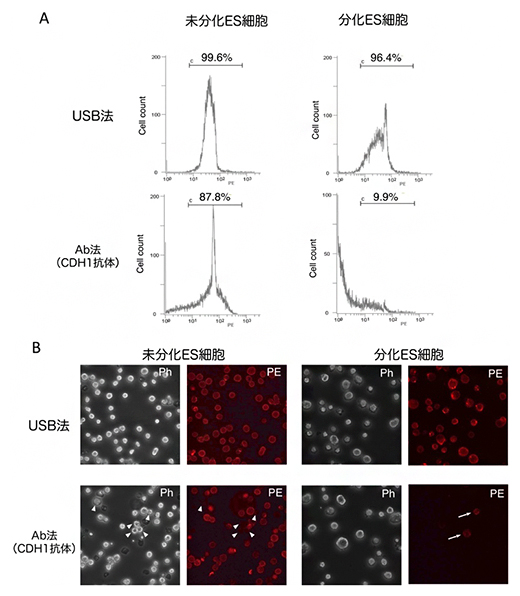
図4 CDH1抗体を用いたAb法とUSB法の標識効率の比較
(A)フローサイトメトリーによる標識細胞の計測結果、(B)蛍光顕微鏡による標識細胞の確認。
USB法ではほぼ全ての細胞が標識されているのに対し、Ab法では分化ES細胞サンプルにおいては約90%の細胞が標識されなかった。Ph:位相差画像(細胞の可視光イメージ)、PE:赤色蛍光画像(細胞が標識されると赤色蛍光を発する)、矢頭:Ab法で標識されなかった未分化ES細胞、矢印:Ab法で標識された分化ES細胞。
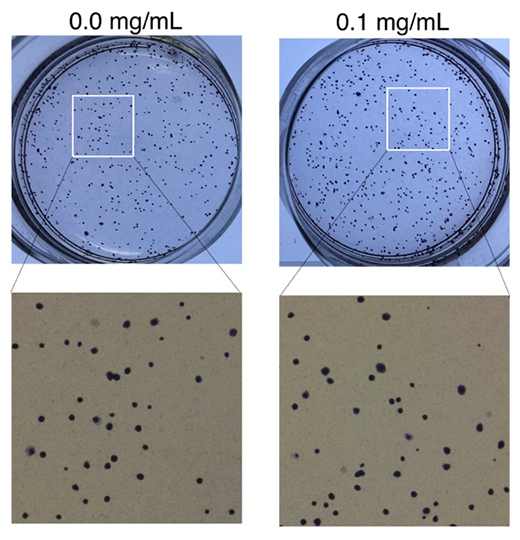
図5 USB法による細胞の生存や増殖への影響の確認
通常の10倍濃度のUSB法試薬で未分化ES細胞(右)を処理した後に培養を継続し、生じた未分化ES細胞のコロニー形成数を確認したところ、未処理細胞(左)との間に差は見られなかった。
次に、実際にUSB法で多重化した細胞を用いて、1細胞遺伝子発現解析が本当に可能か、さらにこの処理によって遺伝子発現に影響が現れないかを示すことが必要です。そこで、USB法、あるいは既存のAb法で標識した8種類のサンプルを用いて得られた1細胞遺伝子発現データを比較しました(図6上段)。次元削減法の一つであるUMAP法[8]などにより、1細胞ごとの遺伝子発現パターンの類似度に応じて細胞をクラスタリング(分類)することができます。つまり、発現パターンが似ている細胞同士は、UMAP上では近接した場所に表示されます。
比較の結果、未分化ES細胞では、USB法で標識した細胞とAb法で標識した細胞の分布がほぼ同じであり、各細胞の発現プロファイルが類似していることが分かりました(図6中段)。実際に、これらの細胞間で統計的に有意な発現の差を示す遺伝子はありませんでした。すなわち、USB法で用いた表面タンパク質のビオチン化などの実験操作が、細胞内の遺伝子発現に与える影響はほぼ無視できることが確認されました。
一方、分化細胞の解析結果を見ると、実際に多様な分化細胞が検出され、多数のクラスターが同定されました(図6下段)。重要なのは、USB法で多重化後、検出された細胞は全てのクラスターで見つかるのに対し、Ab法による細胞はいくつかのクラスターではごく少数しか検出されないことです。これらの細胞群では、CDH1が発現していないため、Ab法では検出されなかったものと考えられます。

図6 サンプル多重化1細胞遺伝子発現解析によるUSB法とAb法の比較
0~7の8種類の細胞について(上段)、UMAP法により、どのような種類の細胞が検出されたかを分類(クラスタリング)した。Cdh1陽性未分化ES細胞では、USB法とAb法で同様に細胞集団が検出された(中段)。分化ES細胞では、Cdh1陰性のクラスター2と4の細胞がAb法ではほとんど検出されなかったが、USB法ではCdh1の有無に関わらずすべての細胞が検出された(下段)。
このマウスES細胞を用いた実験のほかにも、既存のAb法では困難だった細胞の標識・検出がUSB法では可能だった例もあり、USB法が細胞の種類にとらわれることなく、あらゆる細胞で1サンプル多重化を行える汎用性の高い技術であることが明らかになりました。また、USB法は市販の試薬を組み合わせて比較的簡便に実施できる実験技術であり、市販の実験キットなどを使用するよりも低コスト化が可能です。
今後の期待
本研究では、細胞によらない(厳密には、特異的な細胞表面タンパク質に依存しない)サンプル多重化のための細胞標識手法を開発しました。サンプル多重化はエラー低減、コスト削減でのメリットが大きいため、いまだにハードルが高いと思われがちな1細胞遺伝子発現解析の普及に大きく貢献することが期待できます。
補足説明
1.1細胞遺伝子発現解析
網羅的遺伝子発現解析を1細胞ごとに行う手法。さまざまな種類の細胞から構成される集団における細胞の種類・構成比の同定、各細胞で発現する遺伝子の網羅的解析が行える。
2.バッチ効果
実験手技間で生じる結果のゆらぎに起因する誤差。1細胞遺伝子発現解析では、同じ試薬、同じ細胞を用いたとしても、サンプル調製を行ったタイミングや人によって結果に影響が現れてしまう。サンプル多重化により、ひとまとめにして1サンプルとして調整すれば、バッチ効果をなくすことができる。
3.バーコードDNA
サンプルを特定するための、固有の配列を持つ15塩基のDNA配列。バーコードDNAを検出するために必要な配列を加えた69塩基の1本鎖DNAを細胞表面に付加したサンプルを用いて解析することで、得られた1細胞遺伝子発現データがもともとどのサンプル由来であったかをたどることができる。
4.ビオチン
水溶性ビタミンの1種。研究では、DNA、RNA、タンパク質などを標識するためによく利用される。
5.ストレプトアビジン
ビオチンと特異的にかつ強固に結合するタンパク質。あらかじめストレプトアビジンに検出用の物質(本研究ではバーコードDNA)を結合させておくことで、ビオチンで標識された標的(タンパク質など)に検出用の物質を結合させることができる。
6.スルホ-NHS-ビオチン
1級アミンと共有結合する性質を持つNHS-エステルに、スルホ基とビオチンが付加された試薬。スルホ基があることで細胞膜を透過できなくなるため、細胞に対する毒性が低減されている。細胞表面には多くのタンパク質が結合しており、通常タンパク質は少なくとも一つの1級アミンを持つため、この試薬を用いることであらゆる細胞の表面にビオチンを付加できる。
7.ES細胞
胚性多能性幹細胞のこと。着床前胚から樹立される未分化細胞で、体を構成するあらゆる細胞に分化する能力を持つ。本研究ではマウスES細胞を使用した。
8.UMAP法
高次元データとして排出される1細胞遺伝子発現解析結果の次元を削減して、遺伝子発現パターンが類似している細胞同士が近接して表示されるように可視化する手法。同じ種類の細胞同士は塊(クラスター)となって表示される。UMAPはUniform Manifold Approximation and Projectionの略。
共同研究グループ
理化学研究所 バイオリソース研究センター
疾患ゲノム動態解析技術開発チーム
開発研究員 杉本 道彦(スギモト・ミチヒコ)
チームリーダー 阿部 訓也(アベ・クニヤ)
(筑波大学大学院 ライフイノベーション学位プログラム 教授)
開発研究員 田夛 祐喜(タダ・ユウキ)
東京理科大学研究推進機構 生命医科学研究所 炎症・免疫難病制御部門
助教 七野 成之(シチノ・シゲユキ)
京都大学 iPS細胞研究所臨床応用研究部門
教授 妻木 範行(ツマキ・ノリユキ)
(大阪大学 大学院医学系研究科 組織生化学)
テクニカルスタッフ 小屋松 冴子(コヤマツ・サエコ)
(大阪大学 大学院医学系研究科 組織生化学)
研究支援
本研究は、理化学研究所2021年度理事長裁量経費「バイオリソースに対する大規模解析のためのシングルセルトランスクリプトーム基盤構築と検証(申請者:林洋平、阿部訓也)」、日本学術振興会(JSPS)科学研究費助成事業基盤研究(C)「多機能因子として哺乳類胚発生を制御するGARP complexの役割(研究代表者:杉本道彦)」、同挑戦的研究(萌芽)「マイクロパターン培養系を用いたヒト初期発生の統合的理解と疾患研究への展開(研究代表者:阿部訓也)」の支援を受けて行われました。
原論文情報
Michihiko Sugimoto, Yuhki Tada, Shigeyuki Shichino, Saeko Koyamatsu, Noriyuki Tsumaki, and Kuniya Abe, “Universal Surface Biotinylation: a simple, versatile and cost-effective sample multiplexing method for single-cell RNA-seq analysis”, DNA Research, 10.1093/dnares/dsac017
発表者
理化学研究所
バイオリソース研究センター 疾患ゲノム動態解析技術開発チーム
開発研究員 杉本 道彦(スギモト・ミチヒコ)
チームリーダー 阿部 訓也(アベ・クニヤ)
報道担当
理化学研究所 広報室 報道担当