2024-04-03 京都大学iPS細胞研究所
ポイント
- 新型コロナウイルス(SARS-CoV-2)オミクロンBF.5株に持続感染した患者さんから分離したウイルスゲノムを解析した。
- 2カ月以上の持続感染により、ウイルスゲノムに変異が蓄積していた。
- 分離ウイルスのレムデシビルおよびソトロビマブに対する感受性に変化はみられなかった。
- 分離ウイルスのゲノムにおいて、BF.5株以降に誕生した変異株(BJ.1株など)に特徴的な変異が確認された。
1. 要旨
二ツ迫拓樹 学部生(CiRA増殖分化機構研究部門)、高山和雄 講師(CiRA同部門)、長尾美紀 教授(京都大学大学院医学研究科)らは、免疫不全により新型コロナウイルス(SARS-CoV-2)注1)に持続感染した患者さんの検体を用いて経時的なウイルスの変化を解析しました。その結果、抗ウイルス薬や抗体医薬の使用よりも、2か月以上にわたる持続感染によるウイルス複製によってSARS-CoV-2の変異が生じた可能性があること、持続感染した患者さんの検体を用いたウイルスゲノム解析は将来の変異株の予測に役立つ可能性を示しました。
本研究では、約4か月にわたってSARS-CoV-2に持続感染した免疫不全の患者さんから定期的に採取された鼻咽頭スワブ検体より各時点のウイルスを分離し、ゲノムの変化の過程を解析しました。本研究に検体を提供した患者さんは、SARS-CoV-2に対する治療薬としてレムデシビル注2)およびソトロビマブ注3)を投与されていましたが、その期間にウイルスの増殖能や薬剤抵抗性に関わる変異が出現したかを、ゲノム解析に加えてヒトiPS細胞由来肺オルガノイドを用いて調べました。その結果、薬剤の投与によって薬剤耐性ウイルスが出現した可能性は低いことが示唆される一方で、2カ月以上の持続感染によりウイルスがゲノム変異を獲得した可能性が高いことが示されました。これらの解析により、SARS-CoV-2が変異を獲得する過程の一端を明らかにすることができました。
この研究成果は2024年3月29日(米国時間)に「iScience」でオンライン公開されました。
2. 研究の背景
SARS-CoV-2は、2019年12月中国武漢で初めて確認されて以降、世界的な流行が続いています。その要因の一つとして、変異株が高頻度に出現したことが考えられます。SARS-CoV-2変異株ごとに病原性や感染性に加えて、抗体に対する抵抗性も大きく異なります。変異株が生じる機序を理解し、今後流行する変異株を予測することは、新たな変異株の出現に迅速に対応するために役立ちます。しかし、SARS-CoV-2の変異株が出現する要因は十分に明らかになっていません。新型コロナウイルス感染症(COVID-19注4))は通常、発症後1週間程度で患者さんの体からウイルスが排除されるため、ウイルスゲノムの変異の過程を観察することが困難です。一方で、免疫不全の患者さんの場合、SARS-CoV-2が持続的に感染しやすいことから、SARS-CoV-2が変異を獲得する過程を観察できることが知られています。また、SARS-CoV-2に持続感染し、抗ウイルス薬や抗体医薬の投与を受けた患者さんから分離したウイルスゲノムを解析することで、抗ウイルス薬や抗体医薬が変異獲得に及ぼす影響を明らかにできます。本研究では、持続感染患者さんから経時的に採取された検体に含まれるウイルスを分離し、変異株が生じる機序を明らかにすることを試みました。
3. 研究結果
1)SARS-CoV-2持続感染患者さんのプロファイル
本研究は、自己免疫疾患および低ガンマグロブリン血症に罹患し、SARS-CoV-2に持続感染した59歳の女性の患者さん(図1)から採取した検体を用いて解析を行いました。この患者さんは、自己免疫疾患の治療のために、免疫抑制剤のプレドニゾロンを約8年間服用しており、SARS-CoV-2オミクロンBF.5株に感染した後、肺炎などのCOVID-19症状の発症と寛解を繰り返していました。発症後83日目(Day 83)には急性膵炎や重度の肺炎が認められ、人工呼吸器管理やトシリズマブ(TCZ)の投与などが行われましたが、125日目(Day 125)に亡くなりました。発症時(Day 0)と発症後17、56、119日目(Day 17、56、119)に実施したPCR検査ではいずれも多量のウイルスゲノムが検出された(図1表右列; Ct値 = 12.2、14.05、17.05、16.7)ことから、患者さんがSARS-CoV-2に持続感染していたと推測されます。
SARS-CoV-2に対する治療薬としては、レムデシビル(RDV)およびソトロビマブ(Sotrovimab)が投与されていました。患者さんからは発症時(Day 0)以降の鼻咽頭スワブと血清が検体として回収されており、経時的なウイルスの変化を観察することが可能でした。そのため、本症例は変異株が生じる原因を調べるために適していると考え、本研究の解析に使用しました。
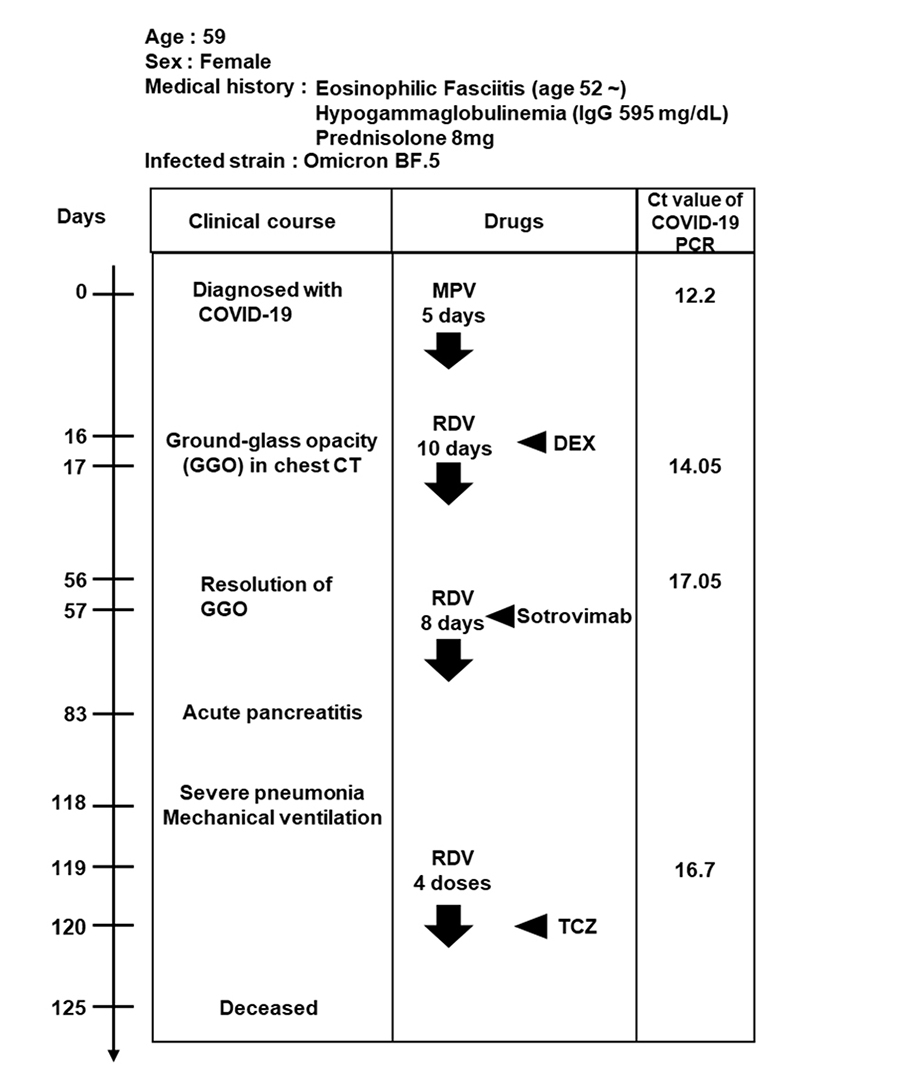
図1:SARS-CoV-2持続感染患者さんの臨床経過
2)分離ウイルスのゲノム解析
はじめに、Day 0、17、56、119における鼻咽頭スワブ検体からウイルスを分離し、ウイルスゲノムを解析しました(図2A)。その結果、各時点で分離されたウイルスはゲノム上にそれぞれ異なる変異をもっていることがわかりました(図2B)。このことは、本症例の患者さんの検体を用いて、SARS-CoV-2のゲノムの変化を追跡できる可能性を示唆しています。また、Day 0からDay 17の期間に10カ所のゲノムが変異していましたが、Day 17からDay 56の期間には新たに27カ所、Day 56からDay 119の期間にはさらに37カ所のゲノムが変異していました。このことから、感染期間が2カ月以上続くことでゲノム変異を獲得しやすくなる可能性が示唆されました。
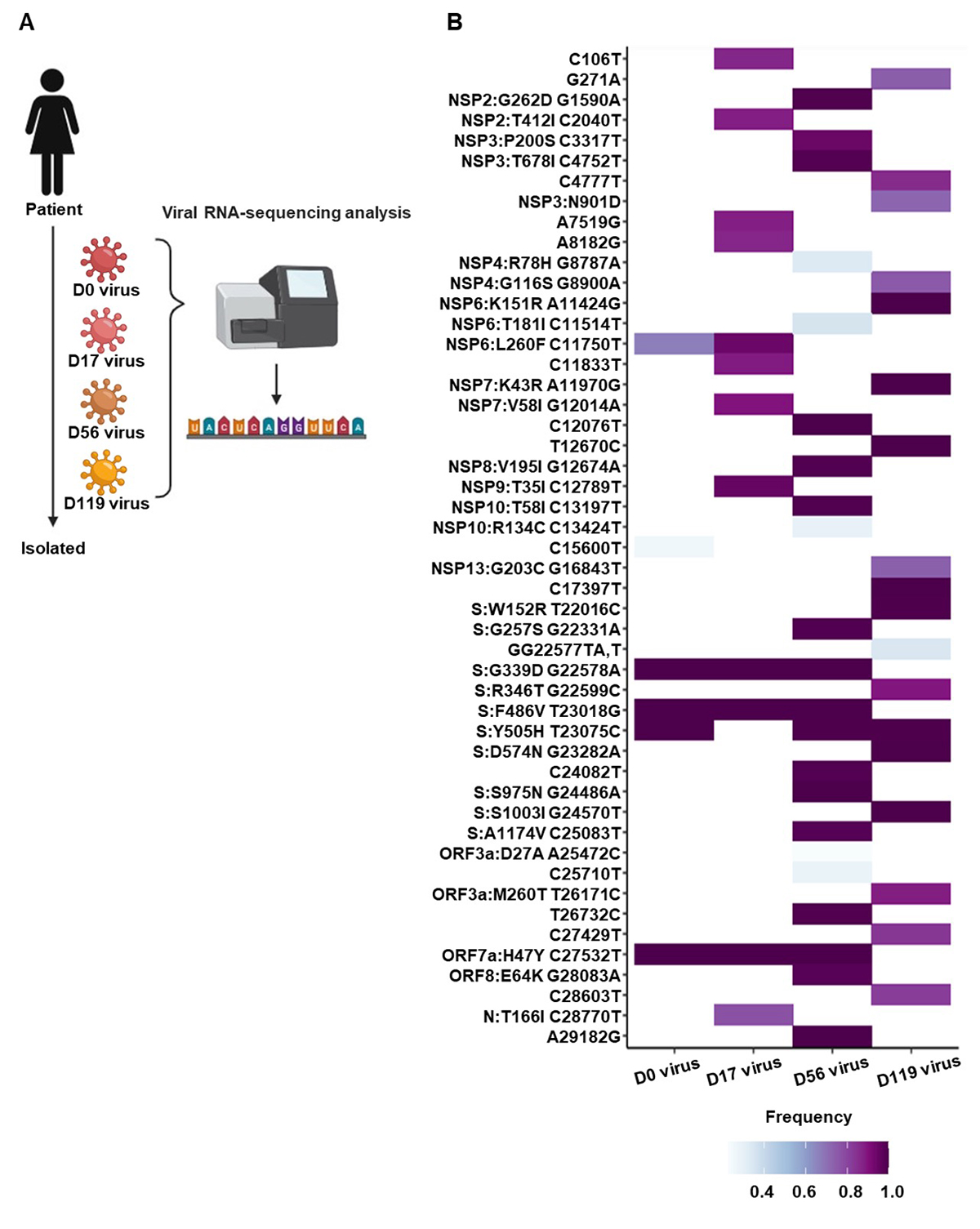
図2:分離ウイルスのゲノム解析
(A) Day 0, 17, 56, 119における患者さんの鼻咽頭スワブ検体からウイルスを分離し、次世代シーケンサーによりゲノム配列を特定した。図はBioRender.comにて作成。(B) ゲノム変異の検出頻度を示すヒートマップ(SARS-CoV-2武漢株の配列との比較)。
3)SARS-CoV-2の持続感染における薬剤耐性ウイルスの出現の検証
研究グループは、各時点の検体から分離されたウイルスの複製能を調べるために、ヒトiPS細胞から作製した肺オルガノイド注5)に感染させ、感染4日後の培養上清におけるウイルスRNAコピー数を測定しました。その結果、いずれの時点の分離ウイルスもRNAのコピー数に大きな差は認められず(図3A)、患者さんの肺細胞への感染能は持続感染を経てもほとんど変化していなかったことが示唆されました。
次に、ウイルスの持続感染と治療薬に対する抵抗性の関係について検討するため、肺オルガノイドと各分離ウイルスを用いて、レムデシビルおよびソトロビマブの感受性を調べました(図3B)。その結果、どの分離ウイルスもレムデシビルに対する感受性は高い一方で(EC50注6) = 0.6-9 nM)、ソトロビマブに対する感受性は低いことがわかりました(EC50 > 10 mg/mL)。また、ソトロビマブの作用標的であるSタンパク質の配列を解析したところ、4種の分離ウイルスはソトロビマブに対して抵抗性を示すことが知られるゲノム変異(G339D、R346T)をもっていました(図3C)。分離ウイルスの肺オルガノイド感染においてソトロビマブの感受性が低かったことは、各ウイルスが共通してもつソトロビマブ抵抗性のゲノム変異を反映していると考えられます。一方で、分離ウイルスにおいて、レムデシビルに対する抵抗性の獲得に関与するNSP12注7)の変異は確認されませんでした(図2B)。以上の結果より、抗ウイルス薬や抗体医薬への感受性は持続感染を経ても変化していなかったと考えられます。

図3:分離ウイルスの抗ウイルス薬および中和抗体に対する感受性
(A)ヒトiPS細胞由来肺オルガノイドに各分離ウイルスを感染させて4日後の培養上清中のウイルスゲノムコピー数。(B)レムデシビル(RDV)またはソトロビマブ(Sotrovimab)の存在下、非存在下のヒトiPS細胞由来肺オルガノイドに各分離ウイルスで感染させ、4日後の培養上清中のウイルスゲノムコピー数から、RDVとソトロビマブのEC50を算出した。(C)Sタンパク質におけるゲノム変異の検出頻度を示すヒートマップ(武漢株の配列との比較)。*p < 0.05, **p < 0.01の有意差を示す。
4)SARS-CoV-2持続感染におけるウイルスゲノムの変化の予測可能性
最後に、SARS-CoV-2に持続感染した患者さんから分離されたウイルスの解析により、ウイルスゲノムの変化を予測できるかを、Sタンパク質のアミノ酸配列に着目して検討しました。患者さんの検体から分離されたウイルスを比較し、持続感染を経て獲得したことが確認された4つのゲノム変異D574N、S975N、S1003I、A1174V(図3C)は、過去にほとんど報告がない変異でした。これらの4つのゲノム変異について、オミクロンBF.5株以降に出現した変異株における出現頻度を調べたところ、興味深いことに、D574N、S1003IはBF.5株と同じBA.5株の子孫株(BA.5.24株、BA.5.2.36株、CG.1株、BF.7.26株、BQ.1.1.21株)のゲノムにおいて1%以上の頻度で出現していました。この2つの変異D574N、S1003Iは、AY.127(デルタ株)やBM.2株、BJ.1株のようなBF.5株と系統発生学的に異なる変異株のゲノムにおいても確認されました(図4)。すなわち、BF.5株は、約4か月間の持続感染の期間に、BF.5株以降に出現する変異を獲得していました。これらの結果より、SARS-CoV-2に持続感染した患者さんのウイルスを今回のように解析することで、将来的なウイルスゲノムの変化を予測できる可能性が示唆されました。一方で、S975N、A1174Vは他の変異株においてほとんど確認されなかったことから、一部のゲノム変異は、実世界におけるウイルスゲノムの変化とは関係がない場合もあると考えられます。
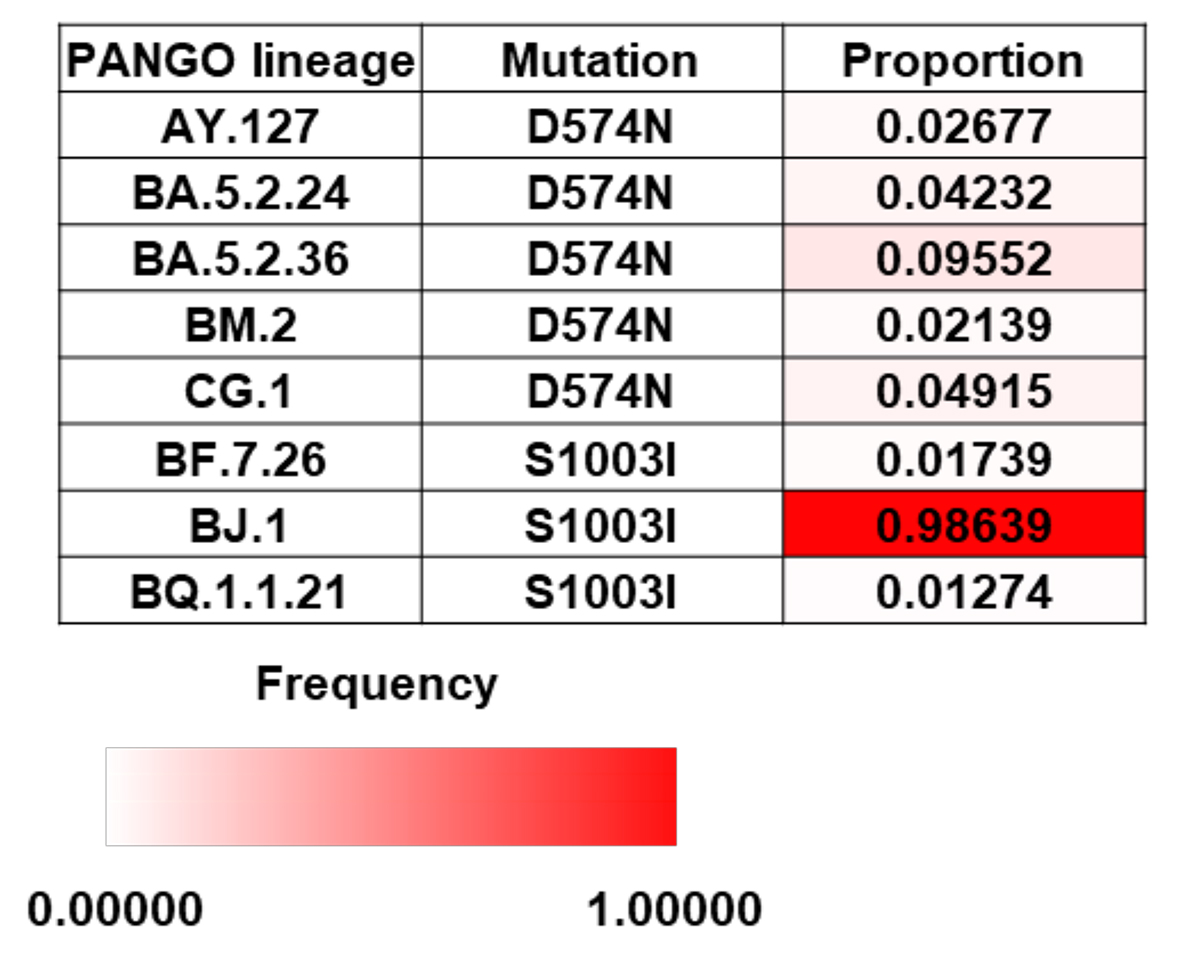
図4:D574NあるいはS1003Iを有する変異株(BF.5株以降に出現したものに限定)
とこれらの変異株におけるゲノム変異の検出頻度
4. まとめと展望
本研究では、SARS-CoV-2に持続感染した患者さんの検体を用いて、ウイルスが変異を獲得する過程を解析しました。ゲノム変異を獲得したウイルスの性状を解析することで、患者さんへの薬剤投与により薬剤耐性ウイルスが出現した可能性は低く、2か月以上の持続感染によりウイルスが変異を獲得した可能性が高いことを明らかにしました。また、SARS-CoV-2持続感染患者さんにおけるウイルス変異蓄積パターンの解析は、ウイルスの変異獲得機序の解明および変化の予測に活用できる可能性があることを見出しました。
5. 論文名と著者
- 論文名
Longitudinal analysis of genomic mutations in SARS-CoV-2 isolates from persistent COVID-19 patient - ジャーナル名
iScience - 著者
Hiroki Futatsusako1, Rina Hashimoto1, Masaki Yamamoto2, Jumpei Ito3, Yasufumi Matsumura2, Hajime Yoshifuji4, Kotaro Shirakawa5, Akifumi Takaori-Kondo5, Genotype to Phenotype Japan (G2P-Japan) Consortium, Kei Sato3, Miki Nagao2,*, Kazuo Takayama1,6,*
*:責任著者 - 著者の所属機関
- 京都大学iPS細胞研究所(CiRA)
- 京都大学大学院医学研究科 臨床病態検査学
- 東京大学医科学研究所
- 京都大学大学院医学研究科 臨床免疫学
- 京都大学大学院医学研究科 血液・腫瘍内科学
- 日本医療研究開発機構革新的先端研究開発支援事業(AMED-CREST)
6. 本研究への支援
本研究は、下記機関より支援を受けて実施されました。
- iPS細胞研究基金
- 京都大学iPS細胞研究所山中伸弥研究室への新型コロナウイルス特別研究助成
- 国立研究開発法人日本医療研究開発機構(JP21gm1610005, JP22fk0108511, JP23jf0126002)
7. 用語説明
注1)新型コロナウイルス(SARS-CoV-2)
新型コロナウイルス感染症(COVID-19)の原因ウイルス。さまざまな変異型が存在している。SARS-CoV-2は、severe acute respiratory syndrome coronavirus 2の略。
注2)レムデシビル
ウイルスタンパク質のうち、ウイルスの複製時に必須な酵素RdRpに作用する抗ウイルス薬。英語ではRemdesivir。図中ではRDVと略している。
注3)ソトロビマブ
生体内への侵入に必要なウイルスの構造タンパク質の一つであるスパイク(S)タンパク質に対する抗体医薬。英語ではSotrovimab。
注4)COVID-19
新型コロナウイルス感染症の正式名称。CO=「corona」、VI=「virus」、D=「disease」。
注5)肺オルガノイド
ヒトiPS細胞より分化誘導した肺オルガノイド。細胞株よりも生体肺組織に類似したモデルとして汎用されている。
注6)EC50
50%有効濃度。薬剤のウイルス阻害効果が50%を示す濃度のこと。
注7)NSP12
RdRpをコードする遺伝子の一つ。