2020-05-06 理化学研究所
理化学研究所(理研)開拓研究本部岩崎RNAシステム生化学研究室の岩崎信太郎主任研究員、韓佩恂国際プログラム・アソシエイト、七野悠一基礎科学特別研究員、水戸麻理テクニカルスタッフI、環境資源科学研究センターケミカルゲノミクス研究グループの吉田稔グループディレクター、ティルマン・シュナイダー・ポッチュ研究員らの共同研究グループは、「ダイソームプロファイリング法」を確立し、タンパク質の翻訳中に起きる「リボソーム渋滞[1]」の網羅的な探索を行いました。
本研究成果は、翻訳そのものに関する基礎研究はもとより、リボソーム渋滞が原因で発症する疾患(神経変性疾患など)の理解に貢献すると期待できます。
翻訳中のリボソームは、一定速度でコドン[2]を読み取るのでなく、一時停止し渋滞することがあります。しかし、どのメッセンジャーRNA(mRNA)[3]のどのコドン上で渋滞するのかなど、根本的なことは全く分かっていませんでした。
今回、共同研究グループは、リボソームの渋滞時に生じるダイソーム(衝突した2個のリボソーム)から得られるmRNA断片を次世代シークエンサー[4]を用いて読み取るダイソームプロファイリング法を開発し、渋滞の位置をヒト細胞の全ゲノムで特定しました。さらに、渋滞は複数の特定アミノ酸配列や終止コドン[2]などで起きること、渋滞したリボソームから合成される新生ペプチドの一部はリボソーム品質管理(RQC)[5]により分解されることなどを明らかにしました。
本研究は、オンライン科学雑誌『Cell Reports』(5月5日付:日本時間5月6日)に掲載されます。
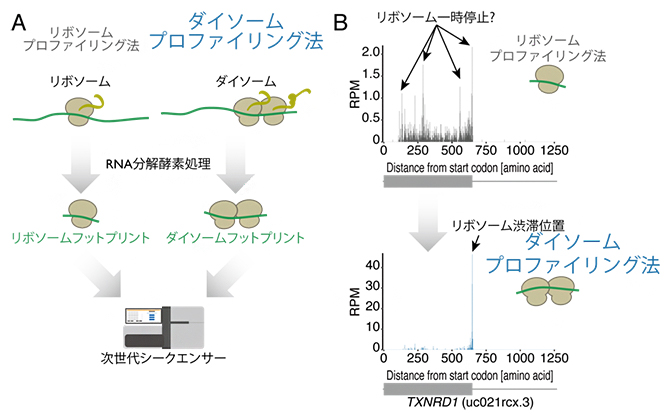
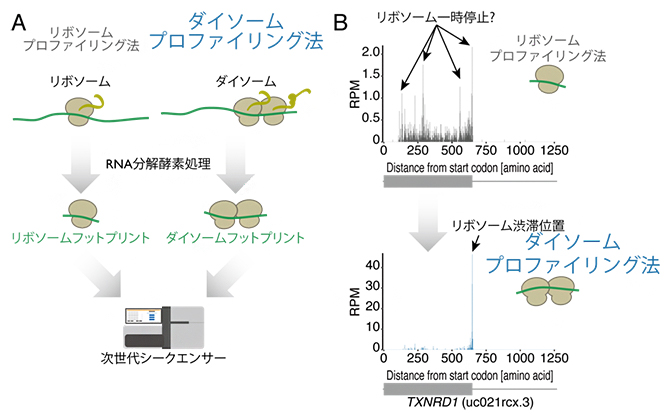
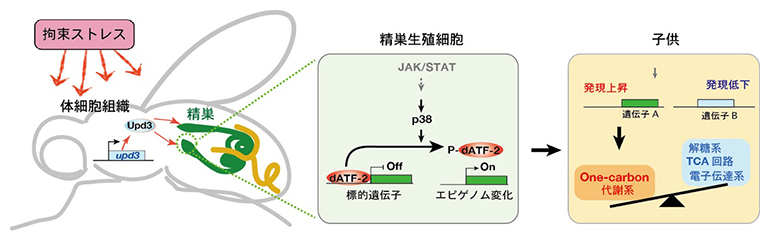
ダイソームプロファイリング法(A)による、リボソーム渋滞位置の高精度同定(B)
背景
「セントラルドグマ[6]」における最後のステップである翻訳は、メッセンジャーRNA(mRNA)の遺伝情報を最終産物のタンパク質に転換するプロセスであり、さまざまな因子によって制御されています。それら制御因子の円滑な働きにより、細胞は恒常性(ホメオスタシス)を保っています。
翻訳の主役を担うリボソームは一定の速度でコドンを読み取るのでなく、その進行は常に多様な因子(例えばmRNAの二次構造、転移RNA[tRNA[3]]の濃度など)に影響されます。そのため、リボソームは一時停止し、「リボソーム渋滞」を引き起こすことがあります。一時停止しても翻訳を再開するならば、細胞のホメオスタシスに影響を与えませんが、翻訳が完全に止まると、リボソームは不完全なペプチドを生産して細胞に毒性をもたらす恐れがあります。これを防ぐために、細胞は複数の経路でリボソームの翻訳速度を監視しています。これまで、適切な技術が存在しなかったために、細胞中のどのmRNAのどの位置でリボソーム渋滞が起きるのか、またその原因などについてはよく分かっていませんでした。
近年、翻訳中のリボソームのmRNA上の位置を網羅的に特定する手法として、「リボソームプロファイリング[7]」という手法が広く用いられています。リボソームプロファイリングでは、リボソームとmRNAの複合体をRNA分解酵素で処理した後、リボソームによって保護されるmRNA断片(リボソームフットプリント)を次世代シーケンサーで読み取ることで、翻訳中のリボソームの位置が特定されます。しかし、従来のリボソームプロファイリングでは、通常通り翻訳しているリボソームと渋滞しているリボソームを見分けることが難しいため、その渋滞位置を正しく知ることができませんでした。
そこで、共同研究グループは、は新しい研究手法を開発することによって、この問題の解決を試みました。
研究手法と成果
共同研究グループは、既存のリボソームプロファイリング法を改良し、渋滞時に生じるリボソーム二つが衝突した複合体ダイソーム(di-ribosome = disome)から得られるmRNA断片を次世代シーケンサーにより解析する手法を開発し、「ダイソームプロファイリング法」と名付けました。そして、この新手法を用いて、リボソーム渋滞の位置を網羅的に同定することに成功しました(図1)。
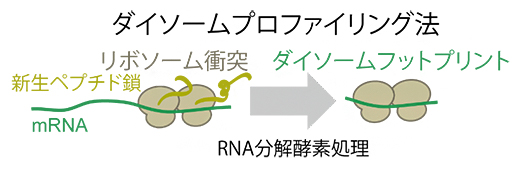
図1 ダイソームプロファイリング法の概略
リボソーム渋滞が生じると、二つのリボソームが衝突する。RNA分解酵素処理後、この二つ分のリボソーム(ダイソーム)によって保護されるmRNA断片(ダイソームフットプリント)を回収し、次世代シークエンサーで解析する。
ヒト細胞を解析したところ、全てのmRNAのうち約10%の遺伝子に少なくとも一つのリボソーム渋滞位置が同定されたことから、従来の想定よりも多くの箇所で渋滞が起きていることが分かりました。また、渋滞は2個のリボソームに限らず、5~6個のリボソームが数珠つなぎになっている様子も観察されました。
次に、渋滞位置を解析した結果、渋滞が生じやすい場所の特徴が明らかになりました。例えば、リボソームに存在する三つのtRNA結合部位であるE-P-A部位[8]に、Pro-Pro-X(P-P-X)もしくはArg-X-Lys(R-X-K)といったアミノ酸配列が多く見つかりました。これらのアミノ酸配列は、リボソーム中で特殊な立体配置をとってしまうことがあります。そのためリボソームのアミノ酸合成に直接関わるペプチジル転移反応[9]が起きにくくなり、渋滞が起きると考えられます(図2上段)。
また、渋滞はmRNAのコーディング領域[10]上だけでなく、一部の終止コドンでも起きることが分かりました。特に、終止コドン上の下流[11]に塩基シトシン(C)がある場合に多く見られ、リボソームが終止コドンで直ちに解離できずに渋滞が起きると考えられます(図2中段)。また、終止コドンの直下流の非翻訳領域3′UTR[10]でも渋滞が起きることが分かりました。通常、リボソームは終止コドン上でmRNAから解離しますが、一部のリボソームは解離されないまま3′UTRに蓄積し、リボソーム渋滞が起きると考えられます(図2下段)。
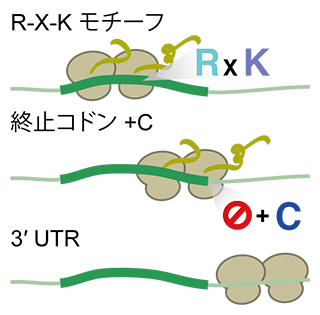
図2 リボソーム渋滞が見つかる位置の特徴
上段: コーディング領域中にArg-X-Lys(R-X-K)といったアミノ酸配列が見られる。
中段: 終止コドン、特に下流に塩基シトシン(C)を持つ場合が多く観察される。
下段: 終止コドンの直下流で、通常ではリボソームが存在しないはずの非翻訳領域3′UTRでもリボソーム渋滞が観察される。
また、リボソーム渋滞のメカニズムの進化的保存性を調べるために、モデル生物のゼブラフィッシュの細胞をダイソームプロファイリング法で解析しました。その結果、ゼブラフィッシュにおいてもヒトと同様のアミノ酸配列で渋滞が生じ、さらに共通祖先に由来するホモログ遺伝子では同じコドンで渋滞が起きることが分かりました。これらの結果から、渋滞のメカニズムが脊椎動物で広く保存されている可能性が示されました。
次に、リボソーム渋滞を解消するメカニズムを理解するために、リボソームがコドンを読み解くために必要な翻訳伸長因子であるeIF5Aに焦点を当てて研究しました。eIF5AはPro-Pro(P-P)配列の間のペプチジル転位反応を促進し、渋滞を防ぐ役割を果たしています。ヒトのeIF5Aノックダウン細胞[12]を作製し、ダイソームプロファイリング法で解析した結果、本来の渋滞位置では著しい変化は見られなかったものの、通常条件下で渋滞が起きなかった部位に新たな渋滞が現れました。そこで、eIF5Aが影響しない渋滞位置をより詳しく解析したところ、渋滞部位の上流[11]に正に荷電したアミノ酸残基が連続していることが分かりました。このことから、正電荷の新生ペプチドと負電荷のリボソームが相互作用することで、eIF5Aが作用できなくなっていることが示されました(図3)。
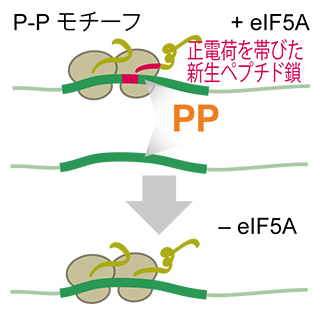
図3 翻訳伸長因子eIF5Aによるリボソーム渋滞を防ぐメカニズム
Pro-Pro-Xモチーフ(P-Pモチーフ)上ではリボソーム渋滞が生じてしまうが、eIF5Aの作用によって解消される。eIF5Aをノックダウンするとそのような渋滞位置が特定できる(下矢印)。P-P モチーフを持っていても、上流にさらに正電荷を持ったアミノ酸配列があると、eIF5Aが作用できなくなり、リボソーム渋滞が生じてしまう。
リボソーム渋滞は、リボソーム品質管理(RQC)と呼ばれる機構を引き起こすことが明らかになっています。RQCは完全に止まったリボソーム渋滞を解消したり、ペプチドを分解したりして、細胞に悪影響が及ばないように働く仕組みだと考えられています。しかし、RQCの内在の標的mRNAはこれまで見つかっていませんでした。そこで、リボソーム渋滞を持つmRNAの中からRQCの標的となるmRNAの特定を試みました。その結果、細胞ストレス依存的な転写因子[13]をコードするXBP1u mRNA[14]がRQCの標的であることが分かりました(図4)。
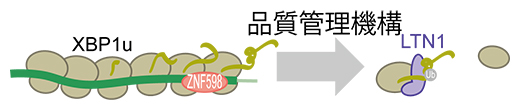
図4 リボソーム渋滞の品質管理
XBP1u mRNA上に生じるリボソーム渋滞は、リボソーム品質管理(RQC)経路による品質管理を受けることが本研究から明らかになった。リボソーム渋滞はZNF598と呼ばれるタンパク質によって認識され、リボソームの解離が誘導される。さらに、新生ペプチド鎖はLTN1と呼ばれるタンパク質因子によってユビキチン化(Ub)され、分解へと導かれる。
今後の期待
ダイソームプロファイリング法は、リボソーム渋滞を網羅的に調べることに特化した高精度の手法です。この手法を活用することで、多種多様な生物種におけるリボソームの翻訳制御をより一層詳しく理解できると期待できます。また、細胞ストレス条件下で生じるリボソーム渋滞が細胞内品質管理を駆動させる機構の理解に応用することができます。
さらに、リボソーム渋滞が原因で発症する疾患の理解に貢献できると考えています。
補足説明
1.リボソーム渋滞
リボソームは、メッセンジャーRNA(mRNA)の翻訳を行なう(遺伝情報からタンパク質を合成する)場所。真核生物では、40Sの小サブユニットと60Sの大サブユニットからなる。リボソームの渋滞は、翻訳を停止したリボソームに次のリボソームが衝突して起こる現象。
2.コドン、終止コドン
タンパク質の中のアミノ酸の並び方は、そのタンパク質の遺伝子(DNA)の中の塩基配列に対応している。3つの塩基がひとまとまりになって一つのアミノ酸に対応しており、この3塩基の並びを「コドン」と呼ぶ。64通りのコドンが存在するが、そのうちの61種類のコドンがアミノ酸を意味しており、例えばTTTはフェニルアラニンである。残り三つのコドン(TAA、TGA、TAG)はタンパク質合成終了を意味する「終止コドン」であり、遺伝子の最後に現れる。
3.メッセンジャーRNA(mRNA)、転移RNA(tRNA)
メッセンジャーRNAは、タンパク質のアミノ酸の並び方の情報(コドン)を持つRNAである。リボソームによってそのコドンが読み取られ、タンパク質が合成される。転移RNAは80塩基程度の小さなRNAであり、アミノ酸に結合している。tRNAはmRNA上のコドンを認識し、対応するアミノ酸をリボソーム上へと運搬する。
4.次世代シーケンサー
DNAの塩基配列を決定するための装置で、DNA断片の塩基配列を同時並行的に、より高速高精度に決定することができる。
5.リボソーム品質管理(RQC)
一時停止したリボソームは細胞に、「異常」として認識され、品質管理の対象として解消される。この機構をリボソーム品質管理機構と呼ぶ。この機構により合成途中のペプチド鎖およびmRNAの分解が誘導されることが分かっている。RQCはRibosome-associated Quality Control の略。
6.セントラルドグマ
細胞内の遺伝情報が、ゲノムDNAの中の遺伝子配列からmRNAに「転写」され、さらに、そのコーディング配列からタンパク質へと「翻訳」されるという、生命に普遍的な決まり(ドグマ)のこと。
7.リボソームプロファイリング
組織からリボソームを抽出し、リボソームと結合しているRNA配列を同定することにより、どの遺伝子がどの程度の効率で翻訳されているかを知る解析法。リボソームは大きな複合体であるため、一定のmRNA領域を覆うように結合する。これらのリボソームとmRNAの複合体に対してRNA分解酵素で処理すれば、リボソームが保護するmRNA断片だけが分解されず回収できる。
8.E-P-A部位
リボソームには三つのtRNA結合部位が存在する。A部位はアミノアシルtRNA結合部位、P部位はペプチジルtRNA結合部位、E部位はtRNAがリボソームから解離する部位である。
9.ペプチジル転移反応
tRNAと合成途中のペプチド鎖が連結した分子をペプチジルtRNAと呼ぶ。リボソームはtRNAとペプチド鎖の結合を切断しつつ、次のアミノアシルtRNAのアミノ酸にペプチド鎖を転移し、アミノ酸が一つ長いペプチジルtRNAを合成する。この反応をペプチジル転移反応と呼ぶ。
10.コーディング領域、3′UTR
mRNAはタンパク質情報を備えているコーディング領域を持つ。その下流のmRNA領域は3′UTR(un-translated region)と呼ばれる非翻訳領域である。
11.上流、下流
RNAは塩基の結合に方向性があり、5′末端側を上流、3′末端側を下流と呼ぶ。リボソームは5′末端側から3′末端側へとコドンを読み解く。
12.ノックダウン細胞
小分子RNAなどの手法により、遺伝子の発現が抑制されることを指す。
13.転写因子
DNAの特定の塩基配列に結合するタンパク質で、プロモーターやエンハンサーといった転写制御領域に結合し、RNAポリメラーゼによる遺伝子の転写を活性化あるいは不活性化する。
14.XBP1u mRNA
XBP1はストレス誘導性の転写因子をコードする遺伝子である。XBP1遺伝子からはXBP1u (un-spliced form)と呼ばれる前駆体mRNAとして転写される。小胞体ストレスにより細胞内でスプライシングを受け、XBP1s (spliced form) mRNAが産生され、適切な転写因子タンパク質が合成される。
共同研究グループ
理化学研究所 開拓研究本部 岩崎RNAシステム生化学研究室
主任研究員 岩崎 信太郎(いわさき しんたろう)
国際プログラム・アソシエイト 韓 佩恂(はん ぺいしゅん)
基礎科学特別研究員 七野 悠一(しちの ゆういち)
テクニカルスタッフI水戸麻理(みとまり)
環境資源科学研究所センター ケミカルゲノミクス研究グループ
グループディレクター 吉田 稔(よしだ みのる)
研究員 ティルマン・シュナイダー・ポッチュ(Tilman Schneider-Poetsch)
東北大学大学院 薬学研究科
教授 稲田 利文(いなだ としふみ)
助教 宇田 川剛(うだがわ つよし)
大学院生 橋本 怜史(はしもと さとし)
奈良先端科学技術大学 動物細胞工学研究室
特任教授 河野 憲二(こうの けんじ)
京都産業大学生命科学部
准教授 三嶋 雄一郎(みしま ゆういちろう)
研究支援
本研究は、本研究は、日本学術振興会(JSPS)科学研究費補助金若手研究(A)「翻訳開始因子パラログによる選択的翻訳の網羅的解析(研究代表者:岩崎信太郎)」、同基盤研究(S)「革新的化学遺伝学による内在性代謝物の新機能の解明と応用(研究代表者:吉田稔)」、文部科学省科学研究費補助金新学術領域研究「Chasing translation along the time by novel ribosome profiling(研究代表者:岩崎信太郎)」、同新学術領域研究「ユビキチン機能制御のためのケミカルバイオロジー(研究代表者: 吉田稔)」、武田科学振興財団ライフサイエンス研究奨励および継続助成「抗がん作用をもつ翻訳阻害剤Rocaglamide Aの作用機序の解明(研究代表者:岩崎信太郎)」による支援を受けて行われました。
原論文情報
Peixun Han, Yuichi Shichino, Tilman Schneider-Poetsch, Mari Mito, Satoshi Hashimoto, Tsuyoshi Udagawa, Kenji Kohno, Minoru Yoshida, Yuichiro Mishima, Toshifumi Inada, and Shintaro Iwasaki, “Genome-wide survey of ribosome collision”, Cell Reports
発表者
理化学研究所
開拓研究本部 岩崎RNAシステム生化学研究室
主任研究員 岩崎 信太郎(いわさき しんたろう)
国際プログラム・アソシエイト 韓 佩恂(はん ぺいしゅん)
基礎科学特別研究員 七野 悠一(しちの ゆういち)
テクニカルスタッフI 水戸 麻理(みと まり)
環境資源科学研究所センター ケミカルゲノミクス研究グループ
グループディレクター 吉田 稔(よしだ みのる)
研究員 ティルマン・シュナイダー・ポッチュ(Tilman Schneider-Poetsch)
報道担当
理化学研究所 広報室 報道担当