2024-09-02 理化学研究所,大阪大学
理化学研究所(理研)開拓研究本部 岩崎RNAシステム生化学研究室の岩崎 信太郎 主任研究員、斉藤 大寛 大学院生リサーチ・アソシエイト(研究当時)、陈 明明 国際プログラムアソシエイト(研究当時)、七野 悠一 研究員、環境資源科学研究センター ケミカルゲノミクス研究グループの吉田 稔 グループディレクター、ティルマン・シュナイダー・ポッチュ 専任研究員、生命機能科学研究センター 翻訳構造解析研究チームの伊藤 拓宏 チームリーダー、髙橋 真梨 テクニカルスタッフⅠ、大阪大学大学院 薬学研究科 量子生命情報薬学分野の福澤 薫 教授、半田 佑磨 特別研究学生(研究当時)らの国際共同研究グループは、抗がん活性を示す小分子化合物「desmethyl, desamino pateamine A(DMDA-PatA)」の作用メカニズムを解明しました。その結果、DMDA-PatAは標的であるRNA結合タンパク質[1] eIF4A[2]およびDDX3[2]に新たなRNA配列特異性[1]を与える化合物であることが分かりました。
本研究成果は、今後DMDA-PatAを基にした抗がん剤設計を支援すると期待されます。
がん治療の手法として抗がん剤の使用は主要な方法の一つですが、重い副作用が問題点として挙げられます。DMDA-PatAは、一部のがん細胞に高い毒性を示す反面、通常細胞には毒性が低いことが報告されており、この細胞特異的な毒性は抗がん剤の重い副作用を回避できる可能性を秘めています。
DMDA-PatAは遺伝子発現のうち翻訳[3]のプロセスを阻害することが報告されていましたが、その分子的な作用機序は未解明でした。今回の研究で、DMDA-PatAは翻訳に関わるタンパク質eIF4AとDDX3をメッセンジャーRNA(mRNA)[4]上の特定の配列(GNGモチーフ。Gはグアニン塩基、Nは任意の塩基)に強固に結合させ、翻訳開始点を探す走査リボソーム[5]進行の立体障害物となることによってmRNAの翻訳を選択的に抑制することを発見しました。
本研究は、科学雑誌『Nature Communications』オンライン版(9月2日付:日本時間9月2日)に掲載されました。
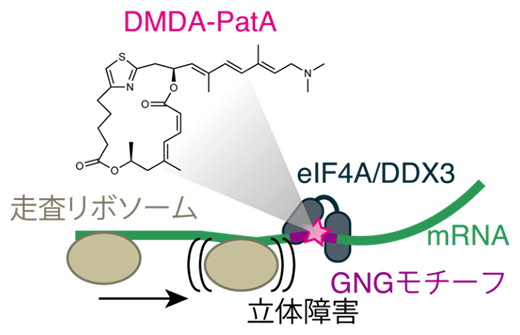
DMDA-PatAの翻訳抑制機構
背景
タンパク質は生体の構成要素であるとともに、生体機能の調節に必須の物質です。そのため、異常なタンパク質の合成は生体の機能を損なう要因となり、多くの疾患の引き金となります。実際、がん細胞ではタンパク質の過剰な合成が観察されています。この過剰なタンパク質合成を抑制する手法の確立は医療分野において非常に重要であり、その手法の一つとして小分子化合物を用いる方法が着目されています。
タンパク質の合成はmRNAからタンパク質を作り出す翻訳という段階を経ます。mRNAはアデニン(A)、ウラシル(U)、グアニン(G)、シトシン(C)の4種類の塩基の組み合わせで構成されています。翻訳ではリボソームと呼ばれる分子がmRNA上を移動しながらその塩基の配列を読み取り、読み取った配列を基にタンパク質を合成します。このリボソームはさまざまな分子と共に機能しますが、代表的なものにRNA結合タンパク質であるeIF4Aが挙げられます。eIF4Aはリボソームが効率的に翻訳を始めることができるようにサポートします(図1左)。
これまで小分子化合物のDMDA-PatAはeIF4Aを標的として翻訳を阻害することが示唆されてきましたが、その詳細なメカニズムは不明でした(図1右)。
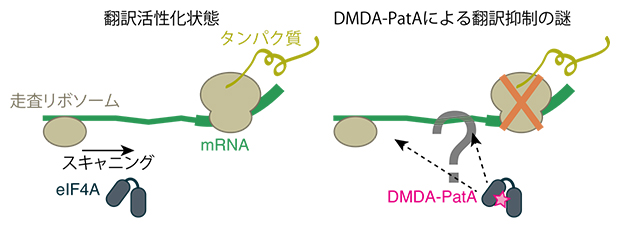
図1 タンパク質合成におけるmRNA翻訳活性化とDMDA-PatAによる翻訳抑制
タンパク質はmRNAのリボソームによる開始コドン走査(スキャニング)、ペプチド伸長という素過程を経てつくられる。eIF4Aは走査リボソームによるスキャニングをサポートする。一方、eIF4Aを標的とするDMDA-PatAがどのように翻訳を抑制しているかという機構は分かっていなかった。
研究手法と成果
本研究ではDMDA-PatAがどのように翻訳を抑制するかを解明するため、次世代シークエンサー[6]技術、古典分子動力学計算[7]・量子化学計算[8]、生化学実験などを組み合わせた多角的なアプローチを行いました。
まず、細胞中のmRNA翻訳状況を網羅的に解析するため、次世代シークエンサー技術であるリボソームプロファイリング法[9]を用いてDMDA-PatAが翻訳に与える影響を評価しました。その結果、DMDA-PatAは一部のmRNAのみを強力に翻訳抑制すること(mRNA選択性)が明らかになりました(図2A)。さらに別の実験では、より強く抑制されるmRNAに対してDMDA-PatAは標的であるeIF4Aをより強固に結合させることも発見しました(図2B)。これらの結果から、DMDA-PatAはeIF4Aを一部のmRNAに強固に結合させ、eIF4が結合したmRNAの翻訳を抑制することが示唆されました。
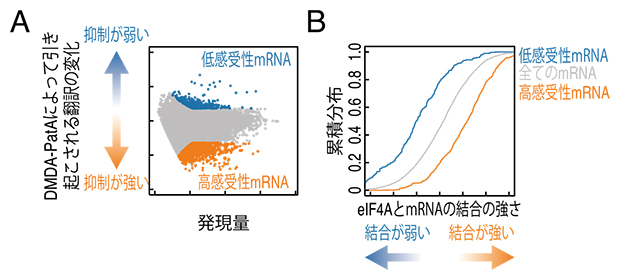
図2 eIF4Aを介したDMDA-PatAの翻訳抑制の実験結果
(A)DMDA-PatAは特定のmRNAの翻訳を強く抑制する。
(B)DMDA-PatAはeIF4Aを高感受性mRNAに対してより強く結合させる。
次に、なぜDMDA-PatAがeIF4Aを一部のmRNAに結合させることができるのかを調べました。DMDA-PatAはmRNA上の特定の配列にeIF4Aを結合させているかどうかを次世代シークエンサー技術のRNA Bind-n-seq法[10]で解析しました。その結果、DMDA-PatAはeIF4AをGNGモチーフ(NはA、U、G、Cのいずれかの塩基)に特に強固に結合させることが明らかになりました(図3A)。これらの結果からGNGモチーフ上に結合したeIF4Aが走査リボソームの障害物として働き、翻訳が抑制されているのではないかと考えられます。実際に試験管内で、GNGモチーフを多く持つmRNAを翻訳させると、DMDA-PatAに高感受性を示し、翻訳が顕著に抑制されました(図3B)。
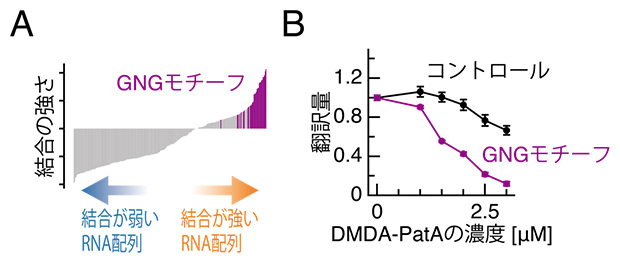
図3 DMDA-PatAを介してGNGモチーフに結合したeIF4Aによるリボソーム停滞と翻訳抑制
(A)DMDA-PatAはeIF4AをGNGモチーフに強固に結合させる。
(B)GNGモチーフを持つmRNAはDMDA-PatAによってより強く翻訳抑制を受ける。濃度単位μMはマイクロモーラ(μmol/L)。
また、DMDA-PatAがどのようにeIF4AをGNGモチーフに結合させるのかを調査するために、eIF4A、RNA、DMDA-PatAの誘導体の複合体の結晶構造を基に、分子動力学計算とフラグメント分子軌道法[11]に基づく量子化学計算を行いました。その結果、DMDA-PatAの先端に位置する三級アミン[12](図4A)とGAGの持つ二つのグアニン塩基(G)上の酸素原子との静電的な相互作用[13]がGNGモチーフ選択性の原因であることが明らかになりました(図4B、C)。
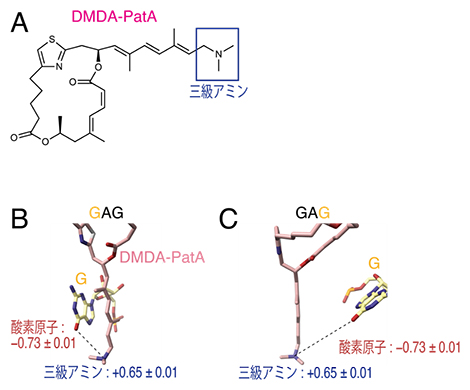
図4 DMDA-PatAとGAGとの相互作用
(A)DMDA-PatAの構造式と三級アミンの位置。
(B、C)DMDA-PatAの三級アミンとグアニン塩基の酸素原子との相互作用。数値はマイナスで負の電荷、プラスで正の電荷(単位はe)を表す。(B)はRNA上のGAG配列のうち1番目のGについて、(C)は3番目のGについて表した。
さらに、本研究の過程で、DMDA-PatAがeIF4Aだけでなく、同じくRNA結合タンパク質であるDDX3も標的とすることが発見されました。RNA Bind-n-seq法で解析した結果、DMDA-PatAはDDX3もGNGモチーフに結合させることが分かりました。
以上の結果から、DMDA-PatAがeIF4AおよびDDX3をmRNA上のGNGモチーフに強固に結合させ、mRNA上で走査リボソームの障害物となることによって、翻訳反応を抑制していることが明らかになりました。同時に、翻訳に対して促進的に働くと考えられてきたeIF4Aが、DMDA-PatAという化合物によってGNGモチーフ選択的な翻訳抑制因子として働くという新しい機能を有していることも分かりました。
今後の期待
本研究により、DMDA-PatAがRNA選択的に翻訳を抑制するメカニズムが明らかになりました。またDDX3という新規標的の発見は、DMDA-PatAがどのようながん細胞に効果的であるのかを調査する上で有用な情報となります。さらに、RNA配列特異性を持たせる上で三級アミンの重要性が明確になったことは、より効力の高いDMDA-PatA誘導体を合成する足掛かりとなります。今後の研究によって、DMDA-PatAを抗がん剤として用いる上での最適化と、従来の治療法よりも副作用の少ない新たな治療薬の開発が期待されます。
補足説明
1.RNA結合タンパク質、RNA配列特異性
RNAに結合するタンパク質は大きく分けて、決まったRNAの配列に結合するもの(RNA配列特異性)とRNAの配列によらず結合するもの(RNA配列非特異性)が存在する。RNA配列特異性を持つRNA結合タンパク質は、決まった種類のRNAのみを制御できる。
2.eIF4A、DDX3
どちらも翻訳開始因子の一つであるDEAD-box型RNA結合タンパク質。mRNAの5非翻訳領域に結合し、RNAの二次構造を解きほぐすなどの機能により、翻訳開始を促進すると考えられている。eIFはeukaryotic translation initiation factorの略。DDXはDEAD-box helicaseの意。
3.翻訳
mRNA(下記[4]参照)へコピーされた塩基配列をアミノ酸配列へ変換して、リボソームでアミノ酸を順番につなげてタンパク質を合成すること。
4.メッセンジャーRNA(mRNA)
タンパク質のアミノ酸の並び方の情報(コドン)を持つRNAのこと。リボソームによってそのコドンが読み取られ、タンパク質が合成される。
5.リボソーム
リボソームRNA(rRNA)とリボソームタンパク質から構成される超巨大複合体。リボソームはmRNAにコードされているコドンを読み取り、タンパク質を合成する。
6.次世代シークエンサー
DNAの塩基配列を決定するための装置。複数のDNA断片の塩基配列を同時並行で、高速・高精度に決定できる。
7.古典分子動力学計算
ニュートンの運動方程式に基づいて、コンピュータを用いて原子や分子の動きをシミュレーションする計算。力場ポテンシャル関数を用いる。
8.量子化学計算
原子や分子の挙動を理解するために、量子力学に基づいて分子の電子状態を明らかにする計算。
9.リボソームプロファイリング法
組織から翻訳装置であるリボソームを抽出し、リボソームと結合しているRNA配列を同定することで、どの遺伝子がどの程度の効率で翻訳されているかを知る解析法。リボソームは大きな複合体であるため、一定のmRNA領域を覆うように結合する。これらのリボソームとmRNAの複合体をRNA分解酵素で処理すると、リボソームが保護するmRNA断片だけが分解されずに回収される。
10.RNA Bind-n-seq法
精製タンパク質とランダムな配列を持つRNAを混合し、タンパク質上に結合したRNAを精製し、RNA配列を次世代シークエンサーにより同定する解析法。
11.フラグメント分子軌道法
タンパク質やRNAをアミノ酸や塩基などのフラグメント単位に分割し、フラグメントとフラグメントペアの電子状態を計算して全体を再構築する量子化学計算法。フラグメント単位の相互作用エネルギー解析が可能である。
12.三級アミン
アンモニアの三つの水素原子がそれぞれ炭化水素基に置換された化合物の総称。
13.静電的な相互作用
電荷を持ったもの同士が引き合ったり、反発したりする力に基づく相互作用。電荷にはプラスとマイナスがあり、反対の電荷同士は引き合い、同一の電荷同士は反発する。
国際共同研究グループ
理化学研究所
開拓研究本部 岩崎RNAシステム生化学研究室
主任研究員 岩崎 信太郎(イワサキ・シンタロウ)
研究員 七野 悠一(シチノ・ユウイチ)
大学院生リサーチ・アソシエイト(研究当時)斉藤 大寛(サイトウ・ヒロノリ)
国際プログラムアソシエイト(研究当時)陈 明明(Chen Mingming)
環境資源科学研究センター ケミカルゲノミクス研究グループ
グループディレクター 吉田 稔(ヨシダ・ミノル)
専任研究員 ティルマン・シュナイダー・ポッチュ(Tilman Schneider-Poetsch)
生命機能科学研究センター 翻訳構造解析研究チーム
チームリーダー 伊藤 拓宏(イトウ・タクヒロ)
テクニカルスタッフⅠ 髙橋 真梨(タカハシ・マリ)
大阪大学大学院 薬学研究科 量子生命情報薬学分野
教授 福澤 薫(フクザワ・カオリ)
特別研究学生(研究当時)半田 佑磨(ハンダ・ユウマ)
ベイラー大学(米国)
教授 ダニエル・ロモ(Daniel Romo)
マックスプランク研究所(ドイツ)
教授 アロイス・ファストナー(Alois Fürstner)
研究支援
本研究は、RIKEN Pioneering Projects「Biology of Intracellular Environments(研究分担者:岩崎信太郎、伊藤拓宏、七野悠一)」、奨励課題(ティルマン・シュナイダー・ポッチュ)で実施し、日本学術振興会(JSPS)科学研究費助成事業基盤研究(B)「ミトコンドリア特異的リボソームプロファイリング法によるオルガネラ翻訳網羅解析(研究代表者:岩崎信太郎)」、同基盤研究(A)「細胞内構造を支えるヘテロポリマー間相互作用の網羅的解析(研究分担者:岩崎信太郎)」、同基盤研究(S)「革新的化学遺伝学による内在性代謝物の新機能の解明と応用(研究代表者:吉田稔)」、同基盤研究(C)「細胞内顆粒P-bodyによるmRNAの空間的制御を介した細胞周期調節機構(研究代表者:七野悠一)」、同学術変革領域研究(B)「新規Disome-Seq法:パラメトリックなリボソーム渋滞の網羅的探索(研究代表者:岩崎信太郎)」、同学術変革領域研究(A)「時間タンパク質学:翻訳速度の大規模並列網羅解析(研究代表者:岩崎信太郎)」「非ドメイン型バイオポリマーの立体構造・相互作用解析(研究代表者:伊藤拓宏)」「APEX-Ribo-Seq:近傍標識による非典型局所翻訳の網羅解析(研究代表者:七野悠一)」「非典型局所翻訳を包括的に解明するAPEX-Ribo-Seq法の確立(研究代表者:七野悠一)」、同新学術領域研究「ユビキチン機能制御のためのケミカルバイオロジー(研究代表者:吉田稔)」、日本医療研究開発機構(AMED)生命科学・創薬研究支援基盤事業(BINDS)「AIとFMO法を融合したインシリコスクリーニングと分子間相互作用解析支援(研究分担者:福澤薫)」による助成を受けて行われました。
原論文情報
Hironori Saito, Yuma Handa, Mingming Chen, Tilman Schneider-Poetsch, Yuichi Shichino, Mari Takahashi, Daniel Romo, Minoru Yoshida, Alois Fürstner, Takuhiro Ito, Kaori Fukuzawa, and Shintaro Iwasaki, “DMDA-PatA mediates RNA sequence-selective translation repression by anchoring eIF4A and DDX3 to GNG motifs”, Nature communications, 10.1038/s41467-024-51635-9
発表者
理化学研究所
開拓研究本部 岩崎RNAシステム生化学研究室
主任研究員 岩崎 信太郎(イワサキ・シンタロウ)
研究員 七野 悠一(シチノ・ユウイチ)
大学院生リサーチ・アソシエイト(研究当時)斉藤 大寛(サイトウ・ヒロノリ)
国際プログラムアソシエイト(研究当時)陈 明明(Chen Mingming)
環境資源科学研究センター ケミカルゲノミクス研究グループ
グループディレクター 吉田 稔(ヨシダ・ミノル)
専任研究員 ティルマン・シュナイダー・ポッチュ(Tilman Schneider-Poetsch)
生命機能科学研究センター 翻訳構造解析研究チーム
チームリーダー 伊藤 拓宏(イトウ・タクヒロ)
テクニカルスタッフⅠ 髙橋 真梨(タカハシ・マリ)
大阪大学大学院 薬学研究科 量子生命情報薬学分野
教授 福澤 薫(フクザワ・カオリ)
特別研究学生(研究当時)半田 佑磨(ハンダ・ユウマ)
報道担当
理化学研究所 広報室 報道担当
大阪大学 薬学研究科 庶務係