2026-01-15 ゲーテ大学
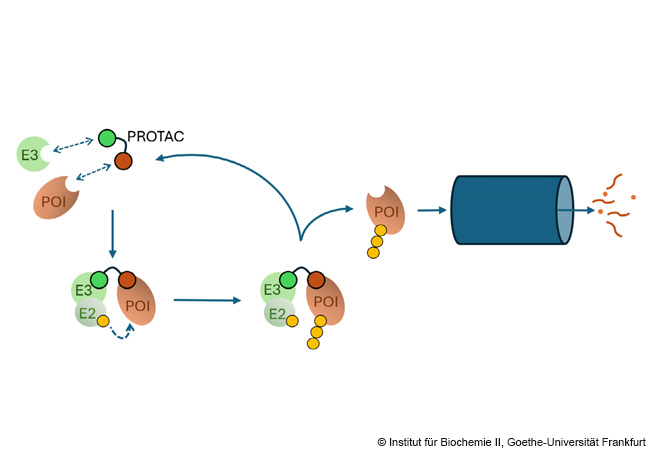
PROTACs link a target protein (protein of interest, POI) with an E3 ligase, which mediates the ubiquitin labeling (yellow) of the POI via an E2 enzyme. The POI is then degraded in the proteasome shredder (blue). © Institute of Biochemistry II, Goethe University Frankfurt
<関連情報>
- https://aktuelles.uni-frankfurt.de/english/the-broker-family-helps-tidy-up-the-cell/
- https://www.nature.com/articles/s41467-025-67450-9
マルチスケール分類によりヒトE3リゴームの複雑さを解明 Multi-scale classification decodes the complexity of the human E3 ligome
Arghya Dutta,Alberto Cristiani,Siddhanta V. Nikte,Jonathan Eisert,Yves Matthess,Borna Markusic,Cosmin Tudose,Chiara Becht,Varun Jayeshkumar Shah,Thorsten Mosler,Koraljka Husnjak,Ivan Dikic,Manuel Kaulich & Ramachandra M. Bhaskara
Nature Communications Published:25 December 2025
DOI:https://doi.org/10.1038/s41467-025-67450-9
Abstract
E3 ubiquitin ligases are vital enzymes that define the ubiquitin code in cells. Beyond promoting protein degradation to maintain cellular health, they also mediate non-degradative processes like DNA repair, signaling, and immunity. Despite their therapeutic potential, a comprehensive framework for understanding the relationships among diverse E3 ligases is lacking. Here, we classify the “human E3 ligome”—an extensive set of catalytic human E3s—by integrating multi-layered data, including protein sequences, domain architectures, 3D structures, functions, and expression patterns. Our classification is based on a metric-learning paradigm and uses a weakly supervised hierarchical framework to capture authentic relationships across E3 families and subfamilies. It extends the categorization of E3s into RING, HECT, and RBR classes, including non-canonical mechanisms, successfully explains their functional segregation, distinguishes between multi-subunit complexes and standalone enzymes, and maps E3s to substrates and potential drug interactions. Our analysis provides a global view of E3 biology, opening strategies for drugging E3-substrate networks, including drug repurposing and designing specific E3 handles.


