2022-11-03 大阪大学,科学技術振興機構
ポイント
- 酵素の基質・補酵素特異性をつかさどるアミノ酸残基を人工知能により推定する手法を開発
- 進化過程で保存されてきたアミノ酸配列をひも解くことで機能に関わるアミノ酸残基の推定が可能に
- 酵素が高い基質特異性を達成できている仕組みの解明と酵素工学技術の高精度化に期待
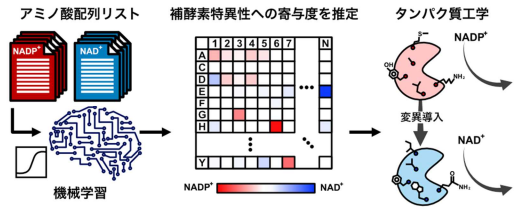
大阪大学 大学院情報科学研究科の二井手 哲平 助教、清水 浩 教授らの研究グループは、データベース上の酵素のアミノ酸配列を人工知能により解析することで酵素の分子認識能力の発現をつかさどるアミノ酸残基を高い精度で可視化できることを世界で初めて明らかにしました。この技術は基質特異性の発現に関わるアミノ酸残基の寄与度をアミノ酸残基レベルで数値表現できるため、たんぱく質工学などで基質特異性改変に応用できます。
反応基質や補酵素に対する酵素の選択性を変えるこれまでの取り組みは、基質・補酵素と酵素の共結晶構造から研究者が知見や経験に依存した変異候補残基の決定や、完全ランダムな変異導入により試みられてきました。しかし、酵素は反応の過程でダイナミックに構造が変化することや、対象とする変異位置の数に応じて変異体候補が指数的に増加することから、基質・補酵素選択性をつかさどるアミノ酸残基の予測や選択性の設計は困難な課題でした。
今回、同研究グループは補酵素特異性ごとに分類したアミノ酸配列データセットをもとに機械学習を実施することで、進化過程で保存されてきた酵素中のどのアミノ酸残基が分子の特異性に寄与するかといった情報を抽出し、さらにどのアミノ酸に変異させると分子選択性が切り替わるかを推論する手法を開発しました。この手法はさまざまな生物に見られる共通した触媒機能を持つ酵素のアミノ酸配列の配列保存度に着目したものであり、データベースの拡充により今後ますますの発展が期待されます。
本研究成果は、米国科学誌「ACS Synthetic Biology」に、11月2日(水)(アメリカ東部時間)に公開されます。
本研究は、JST 戦略的創造研究推進事業 ACT-X「環境とバイオテクノロジー」、JSPS 科学研究費補助金 基盤研究S、基盤研究Cの一環として行われました。
<論文タイトル>
- “Logistic Regression-Guided Identification of Cofactor Specificity-Contributing Residues in Enzyme with Sequence Datasets Partitioned by Catalytic Properties”
DOI:10.1021/acssynbio.2c00315
<お問い合わせ先>
<研究に関すること>
二井手 哲平(ニイデ テッペイ)
大阪大学 大学院情報科学研究科 助教
清水 浩(シミズ ヒロシ)
大阪大学 大学院情報科学研究科 教授
<JST事業に関すること>
宇佐見 健(ウサミ タケシ)
科学技術振興機構 戦略研究推進部 先進融合研究グループ
<報道担当>
科学技術振興機構 広報課