スーパーコンピュータ「京」で巨大分子機械の動きを計算
2018-03-12 理化学研究所,横浜市立大学,東京工業大学
要旨
理化学研究所(理研)計算科学研究機構粒子系生物物理研究チームの松永康佑研究員(JSTさきがけ研究員)、横浜市立大学大学院の山根努特任助教、池口満徳教授、木寺詔紀教授、東京工業大学の村上聡教授らの共同研究グループ※は、社会問題となっている多剤耐性細菌の原因の一つとされる多剤排出トランスポーター[1]「AcrB」の薬剤排出機構を、スーパーコンピュータ「京」[2]を用いたシミュレーションによって解明しました。
病原菌やがん細胞に対して薬が作用しなくなる「薬剤耐性化」は、現代の医療現場で大きな問題となっています。特に、院内感染を起こす緑膿菌などの薬剤耐性化は、細菌の膜に存在する多剤排出トランスポーターと呼ばれるタンパク質が薬剤を細胞外へ排出することが主な原因と考えられています。そのため、多剤排出トランスポーターの薬剤排出機構の解明が課題となっています。大腸菌由来の多剤排出トランスポーターであるAcrBは、村上教授らが2002年および2006年にX線結晶構造解析[3]によって構造を解明し、それに基づいた作動原理として「機能的回転機構[4]」仮説を提唱しました注1)。2010年には、この仮説は京都大学の高田彰二教授らによる粗視化シミュレーション技法[5]などによって実証されました注2)。しかし、膜を介したプロトン(水素イオン)移動によってどのようにAcrBの構造変化が起き、機能的回転が起るのか、その動的な機構は分かっていませんでした。
今回、共同研究グループは、AcrBとその周りの膜や水分子を「丸ごと」模した全原子モデルを計算機上で再現し、「京」の高並列性を生かすアルゴリズムを用いて薬剤排出過程を計算しました。その結果、AcrBの膜内にあるアスパラギン酸にプロトンが結合すると、薬剤排出へつながる構造変化が引き起こされること、また50オングストローム(Å、1Åは100億分の1メートル)も離れた薬剤排出部位の構造変化へ至る過程を明らかにしました。
本成果は、排出を阻害する薬剤開発の基礎に貢献するとともに、生体分子で見られる化学エネルギー・力学エネルギー変換機構の一例を提供するものです。
本研究は、英国のオンライン科学雑誌『eLife』(3月6日付け)に掲載されました。
本研究の一部は、文部科学省「次世代生命体統合シミュレーションソフトウエアの研究開発」プロジェクトによる支援を受けました。また、本研究はHPCI「京」一般利用課題「最小自由エネルギー経路探索法による多剤排出トランスポーターの薬剤排出機構の解明(課題番号:hp120027)」、ポスト「京」重点課題1研究開発枠(課題番号:hp150269, hp160223, hp170255)として「京」の計算資源を用いて実施しました。
注1) S. Murakami, R. Nakashima, E. Yamashita, T. Matsumoto, and A. Yamaguchi, “Crystal structures of a multidrug transporter reveal a functionally rotating mechanism” Nature 443, 173-179 (2006). doi:10.1038/nature05076
注2)2010年11月17日プレスリリース「多剤排出トランスポーターの機能を分子シミュレーションで初解明」
※共同研究グループ
理化学研究所 計算科学研究機構 粒子系生物物理研究チーム
研究員 松永 康佑 (まつなが やすひろ)
横浜市立大学大学院 生命医科学研究科
特任助教 山根 努 (やまね つとむ)
特任准教授 森次 圭 (もりつぐ けい)
教授 池口 満徳 (いけぐち みつのり)
教授 木寺 詔紀 (きでら あきのり)
東京工業大学 生命理工学院
教授 村上 聡 (むらかみ さとし)
東京大学 大学院農学生命科学研究科
特任准教授 寺田 透 (てらだ とおる)
日本医科大学 物理学教室
准教授 藤崎 弘士 (ふじさき ひろし)
背景
効くはずの薬が効かなくなるという「薬剤耐性化」の問題は、現代の医療現場で大きな問題となっています。病原菌やがん細胞がこのような薬剤耐性を持つメカニズムはいくつかありますが、細胞膜に埋まっている多剤排出トランスポーターと呼ばれるタンパク質が原因の一つであることが分かっています。このタンパク質は巨大な分子機械で、抗生物質や毒物などの異物を取り込んでは細胞外に排出します。これにより、病原菌などに作用するはずの薬剤が効かなくなる現象が起こります。特に、院内感染で社会問題となっている緑膿菌などの薬剤耐性化は、RND型[6]と呼ばれる多剤排出トランスポーターが主な要因になっています。
RND型の多剤排出トランスポーターは、細胞膜内外の酸性度(pH)の違いによりプロトン(水素イオン)が移動することを利用して薬剤を排出します。大腸菌由来のRND型多剤排出トランスポーター「AcrB」の構造は、2002年および2006年に村上教授らがX線結晶構造解析によって解明しました。AcrBは約1,000個のアミノ酸からなる巨大な分子が三つ集合した3量体で、三つの分子はそれぞれ異なる構造をとり、取込状態→結合状態→排出状態と順に構造変化して薬剤を排出していると考え、「機能的回転機構」仮説を提唱しました。
AcrBの構造で興味深いことは、薬剤排出に関わっている部位が、細胞膜内のプロトン結合部位と約50オングストローム(Å、1Åは100億分の1メートル)も離れている点です。プロトン移動という非常に小さな動きが、どのように増幅されて機能的回転につながる大きな構造変化と関わっているのか、機能的回転やプロトンを実験で観測することは難しく、その詳細は未解明でした(図1)。
2010年に京都大学の高田彰二教授らは、AcrBを構成する原子を粗視化したモデルのシミュレーションを行って、機能的回転機構仮説を実証しました。その粗視化モデルでは、プロトン移動による駆動力を抽象的に扱い、機能的回転が起こると薬剤を排出することを示しました。さらに2013年には、山根特任助教と池口教授らが、AcrBの全原子モデルのシミュレーションを行い、プロトン移動の際に膜内のどこの部位にプロトンが一時的に結合するのかを解明しました。プロトンをいくつかのアミノ酸に結合させ、シミュレーションを多数行うことで、排出型分子の膜内のアスパラギン酸(Asp408)というアミノ酸にプロトンが結合していると安定であることを示しました注3)。
しかし、Asp408にプロトンが一時的に結合・解離することが、どのように機能的回転へとつながる構造変化を引き起こすのかは解明されていません。プロトンと他の原子の相互作用を観察するには、全原子モデルを使う必要がありますが、機能的回転はおよそミリ秒(1,000分の1秒)で起こる非常に遅いプロセスのため、全原子モデルでシミュレーションすることは困難でした。そこで、共同研究グループはスーパーコンピュータ「京」とその高並列性を生かすアルゴリズムを用いて、解明に取り組みました。
注3) T. Yamane, S. Murakami, and M. Ikeguchi, “Functional rotation induced by alternating protonation states in the multidrug transporter AcrB: all-atom molecular dynamics simulations” Biochemistry 52, 7648-7658 (2013). doi:10.1021/bi400119v
研究手法と成果
共同研究グループは、AcrB 3量体の全原子モデル(周りの水分子や脂質、薬剤を全て含めて約50万原子系)について、「京」を用いて機能的回転へとつながる構造変化を分子動力学法[7]でシミュレーションしました。分子動力学法では、1フェムト秒(1,000兆分の1秒)の1ステップごとに各原子に働く力を計算する必要があります。機能的回転は約1ミリ秒(1,000分の1秒)で起こるので、シミュレーションするには1兆回もの計算が必要となります。これを達成するには、例え1ステップを実時間で1ミリ秒で計算したとしても、数十年かかってしまいます。
そこで、計算を時間方向に分割するストリング法[8]というアルゴリズムを組み合わせました。ストリング法では、一つの系をシミュレーションする代わりに、構造変化の経路を表した複数の系の分子動力学計算をまとめて行い、最も起こりやすい構造変化経路を探します。複数の系をまとめてシミュレーションするため、「京」の高並列環境があって初めて可能となった計算です(図2)。
ストリング法を使ったシミュレーションの結果、AcrB 3量体の内で結合状態にある分子のAsp408にプロトンが結合すると、機能的回転へとつながる構造変化が引き起こされることが分かりました。実際、構造変化経路に沿ったエネルギー変化を計算すると、結合状態が不安定化する一方で、排出状態が安定化することが示されました。対照実験として、排出状態にある分子のAsp408にプロトンを結合させて計算すると、排出状態が安定化したままで機能的回転は誘起されませんでした。
また、得られた構造変化経路を解析することで、離れた部位が連携して動いていることが明らかになりました。まず、膜に埋まっているAsp408の周りで何が起こっているのかを構造解析したところ、プロトン結合により膜に埋まっている部位で特定のαヘリックス[9]が下がる運動が生じていることが分かりました。興味深いことに、この下がる運動はプロトン供給源である水分子の出入りを制御しており、結合状態においてプロトンの取り込み、排出状態においてプロトンの放出を起こしやすくしていました。これは結合状態においてプロトンが結合し機能的回転を起こすという結果とつじつまが合います。
さらに、膜に埋まっている部位と薬剤排出部位の動きの網羅的な相関解析を行ったところ、プロトン結合から薬剤排出へと至る相互作用の連鎖が明らかになりました。具体的には、αヘリックスが下がる運動が、膜に埋まっている部位と薬剤排出部位をつなぐ部位のコイル・ヘリックス転移[10]を制御しており、その転移が薬剤排出部位の大規模な構造変化を引き起こしていました(図3)。
今後の期待
本成果は、多くの生体分子にみられるプロトン結合による化学エネルギーと薬剤輸送という力学的エネルギーの間の変換という基礎的問題に対して、一つの機構を提供します。それと同時に、多剤排出トランスポーターによって排出されない薬剤や、排出を阻害する薬剤の開発に資すると期待できます。
また、「京」などのスーパーコンピュータを用いた高性能計算により、実験では観察することが難しい分子機械の動く様子を明らかにできることを示しています。今後、ポスト「京」[11]により網羅的な計算が可能になると、さまざまな薬剤の排出過程を観測して、排出される薬剤とされない薬剤の違いを解明したり、AcrBと結合するAcrZなどの他のタンパク質の影響を明らかにできると期待されます。
原論文情報
Yasuhiro Matsunaga, Tsutomu Yamane, Tohru Terada, Kei Moritsugu, Hiroshi Fujisaki, Satoshi Murakami, Mitsunori Ikeguchi, and Akinori Kidera, “Energetics and conformational pathways of functional rotation in the multidrug transporter AcrB”, eLife, doi: 10.7554/eLife.31715
発表者
理化学研究所
計算科学研究センター 研究部門 粒子系生物物理研究チーム
研究員 松永 康佑 (まつなが やすひろ)
横浜市立大学大学院 生命医科学研究科
特任助教 山根 努 (やまね つとむ)
教授 池口 満徳 (いけぐち みつのり)
教授 木寺 詔紀 (きでら あきのり)
東京工業大学 生命理工学院
教授 村上 聡 (むらかみ さとし)
報道担当
理化学研究所 広報室 報道担当
横浜市立大学 研究企画・産学連携推進課長 渡邊 誠
東京工業大学 広報・社会連携本部 広報・地域連携部門
産業利用に関するお問い合わせ
理化学研究所 産業連携本部 連携推進部
補足説明
-
- 多剤排出トランスポーター
- 細胞膜を介して分子を輸送する膜タンパク質を総称してトランスポーターと呼ぶが、抗生物質などいくつかの薬剤を排出するトランスポーターを特に多剤排出トランスポーターという。
-
- スーパーコンピュータ「京」
- 文部科学省が推進する「革新的ハイパフォーマンス・コンピューティング・インフラ(HPCI)の構築」プログラムの中核システムとして、理研と富士通が共同で開発を行い、2012年9月に共用を開始した計算速度10ペタFLOPS級のスーパーコンピュータ。
-
- X線結晶構造解析
- タンパク質の結晶を作製し、その結晶にX線を照射して得られる回折データを解析することにより、タンパク質の内部の原子の立体的な配置を調べる方法。この方法によって、タンパク質の立体構造や内部構造を知ることができる。
-
- 機能的回転機構
- X線結晶構造解析で得たAcrB3量体の非対称構造をもとに、2006年に村上教授らが提案したAcrBの作動原理を説明するモデル。非対称構造では3量体の各分子は三つの異なる状態をとる。一つ目の分子は薬剤待ちの「取込状態」、二つ目の分子は薬剤に結合する「結合状態」、三つ目の分子は薬剤排出する「排出状態」と呼ばれる。薬剤1分子を排出すると3量体の構造状態がちょうど1段階ずつ変化し、一つ目の分子が結合型、二つ目の分子が排出型、三つ目の分子が取込型になると考えた。図に示したように、細胞外側からみると、3量体の構造状態が120度回転していることに対応するので、機能的回転機構と呼ばれる。この変化によって薬剤1分子が外側に運ばれる。同じ分子でできた3量体が非対称な構造状態をとる様子は、ATP合成酵素のF1-ATPaseと類似していることから、F1-ATPaseの作動原理との類推によって考案された。
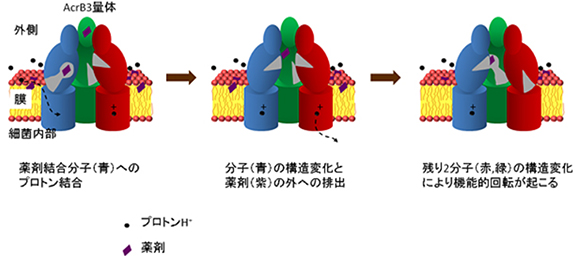
プロトン結合に起因したAcrBの薬剤排出と機能的回転
-
- 粗視化シミュレーション技法
- 広義には「もとの問題の重要な側面だけを残してより簡単な表現にする」ことを粗視化というが、ここでは原子レベルによるタンパク質表現から、アミノ酸1個を1粒子として近似する粗視化による分子動力学シミュレーションの技法を表す。
-
- RND型
- RNDスーパーファミリーは、大腸菌、緑膿菌などのグラム陰性菌にみられるトランスポーター群であり、主に、pHの差に起因するプロトン輸送を駆動力としてリガンドを輸送する機能を持つ。RNDはResistance-nodulation-cell divisionの略。
-
- 分子動力学法
- 計算機の中でモデルの原子間に働く力を計算し、ニュートンの運動方程式を繰り返し解くことで、分子の動きを追跡する方法。
-
- ストリング法
- ニュートンの運動方程式を解く代わりに、最初に始状態と終状態を設定し、その間をつなぐ構造変化経路が物理的にもっともらしくなるように最適化する手法。始状態と終状態の間には、複数の少しずつ構造の異なるシミュレーション系を配置して、構造変化経路を表現する。それらを連携させながらまとめてシミュレーションを行うことで、構造変化経路を最適化する。
-
- αへリックス
- タンパク質の二次構造の共通モチーフの一つで、バネのような右巻きらせんの形をしている。
-
- コイル・ヘリックス転移
- タンパク質やポリペプチド鎖の一部または全部が、特定の構造をとらないランダムコイル構造からαヘリックスへと構造転移する現象。
-
- ポスト「京」
- 「京」の後継機として、2021年から2022年の運用開始を目標に、理化学研究所が主体となって開発を進めている次世代フラッグシップスーパーコンピュータ。
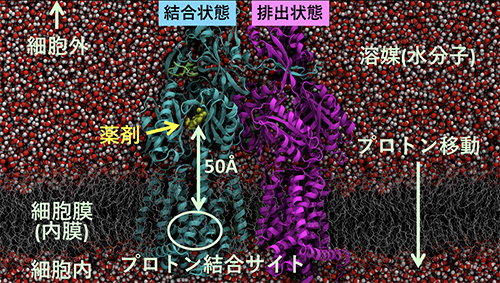
図1 多剤排出トランスポーターAcrBの構造
多剤排出トランスポーターAcrBの構造を横から見た図。上が細胞外、下が細胞内に対応する。結合状態にある分子に薬剤が結合している。プロトン結合サイトは膜内に存在する。
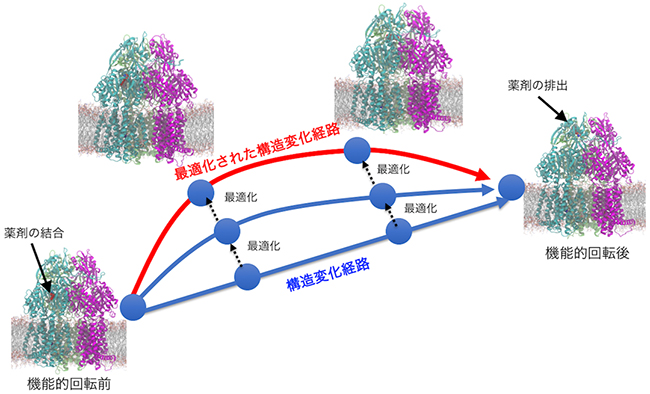
図2 ストリング法の概念図
最初に始状態と終状態を設定し、機能的回転へつながる構造変化経路を複数のシミュレーション系(青の丸)で表現する。それらを連携させながらまとめてシミュレーションを行うことで、赤の線のように構造変化経路を最適化する。
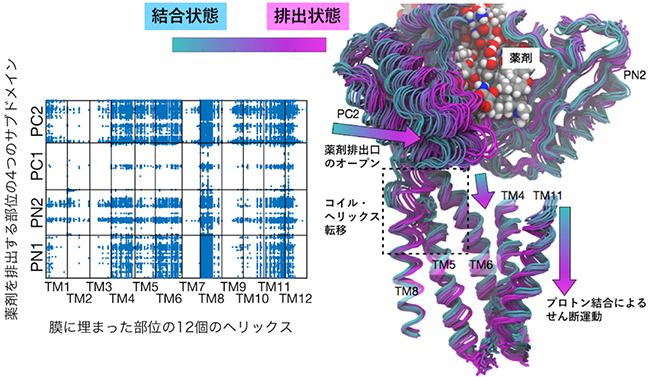
図3 膜に埋まった部位と薬剤排出部位の動きの網羅的な相関解析の結果
(左) 相関のあるアミノ酸残基のペアをプロットしたもの。特定のαヘリックスとサブドメイン間に相関があることを示す。
(右) 相関のある部位の動いている構造を重ねて描いたもの。図の右側から、プロトン結合による並進運動(αへリックスが下がる運動)→ヘリックス・コイル転移→ドメイン運動による薬剤排出口のオープンを示す。