誰にでも使える創薬支援解析ツールを開発・公開
2020-10-02 大阪大学,科学技術振興機構
ポイント
- 遺伝子発現情報のみから実験をせずに細胞中の薬剤標的分子の活性を定量的に予測する計算手法の開発
- 計算手法の高度化によるがんゲノムデータを用いた細胞シミュレーション解析ツールの構築・公開
- 個人の遺伝子特性を反映した患者固有モデルの構築を実現することで創薬の加速や個別化医療の進展に期待
大阪大学 蛋白質研究所 細胞システム研究室(岡田 眞里子 教授)の研究グループは、同大学 大学院理学研究科の大学院生 井元 宏明さん(博士後期課程)を中心に、個別医療を目標とした患者固有モデルの構築に向けた計算手法を開発し、それに基づき、がんゲノム情報と細胞シミュレーション技術を組み合わせた創薬支援解析ツールを構築・公開しました。
これまでがん治療においては、がんの大きさ、浸潤の程度、疾患マーカーの有無などのいくつかの指標に基づき治療法が選択されてきました。しかし、これまでの分類法では患者の予後に大きな違いが生じることから、個々の患者の遺伝子情報に基づいた新たな分類法の開発が急務となっています。本研究では、がんの層別化(たくさんのがんゲノムデータを遺伝子の特徴や薬剤応答によってグループ分けすること)や個別化医療を目標とした患者固有モデルの構築を目的としました。
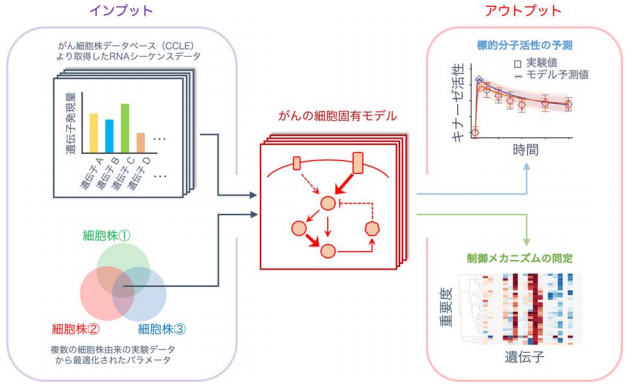
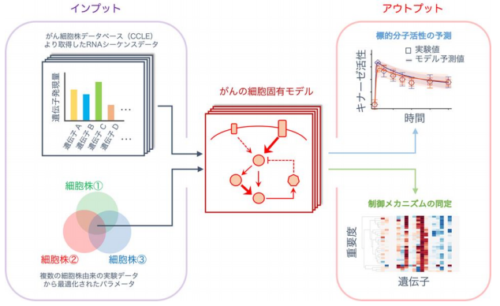
そのために、公共データベースから取得したがん細胞株のRNAシーケンス(網羅的な遺伝子発現)データと細胞シミュレーションを組み合わせ、がん細胞増殖の標的であり、薬剤探索の指標となるキナーゼ(リン酸化酵素)の活性を定量的に予測する計算手法を開発しました。
この方法を用いると、複数の細胞株の実験データを数理モデルに学習させることにより、RNAシーケンスデータのみを入力として、学習していない別の細胞株のキナーゼの活性値を実験を行わずにシミュレーションのみで予測できるようになるため、創薬研究現場の大幅な効率向上が可能となります。
さらに、この手法をもとに、細胞実験データの種類や量を増やし、数理モデルや解析法の精緻化と拡張を行っていけば、細胞株データを用いて学習させた数理モデルに患者検体由来のRNAシーケンスデータを入力することにより、患者固有モデルの構築が可能になると考えられます。また、そのことにより、がんの層別化や患者ごとに適した薬剤の探索が可能になると考えられます。
本研究では、多くのがんで重要な働きを担うErbB(HER)受容体シグナル伝達系のネットワーク(313パラメータ、39遺伝子)について、分子間の結合解離・酵素反応・分子局在・分解などを多数の文献をもとに常微分方程式により再現した数理モデルを構築し、乳がん細胞株を対象に研究を進めました。数理モデルは、ネットワークを構成する分子、分子の量などの初期値、分子間の結合強度などの動的なパラメータから構成されており、これらの要素、量、パラメータを変えることにより、薬剤探索や薬剤評価に資するシミュレーションが可能となります。本解析では、感度解析という手法により、多くのがんにおいて薬剤耐性時の活性化上昇が認められ薬剤標的としても研究が進められるAktキナーゼ(プロテインキナーゼB)の分子活性を指標に、Akt活性の制御に適した分子および適さない分子を同定しました。また、本解析からは細胞ごとの遺伝子発現量の差異が、遺伝子変異などよりも、それぞれの細胞内での分子の実活性や細胞の性質を決める上で、重要な指標となり得る可能性が示唆されました。
このような数理モデルを用いた細胞シミュレーション技術は、疾患メカニズムの同定や定量的解析において優れており、ビッグデータに基づく分類に優れた人工知能(AI)と相補的に創薬研究に用いられることが期待されています。本研究における細胞シミュレーションの解析基盤は、学習実験データをもとにモデルの動的パラメータを最適化するパラメータ最適化法、遺伝子ネットワークの重要な要素を同定する感度解析、公共データベースから遺伝子発現情報を取得するツール、シミュレーション・解析結果を簡単に可視化する解析ツールを含み、BioMASS(Modeling and Analysis of Signaling Systems)は、パッケージ化し、オープンソースソフトウェアとして公開しています(https://github.com/okadalabipr/biomass)。この解析ツールは、細胞モデリングに必要な一連の技術を集積し、モデリングに馴染みのない人でも約一週間で解析ができるようになります。
本研究成果は、近日中に国際科学誌「Cancers」に公開されます。
本研究は、科学技術振興機構(JST) 未来社会創造事業 探索加速型「共通基盤」領域の研究開発課題「創薬を加速する細胞モデリング基盤の構築(研究開発代表者:岡田 眞里子)」および日本学術振興会科学研究費補助金 基盤研究(A)「疾病機序理解のための遺伝子ネットワーク数理モデル基盤の構築」などの支援を得て行われました。
<論文タイトル>
- “A computational framework for prediction and analysis of cancer signaling dynamics from RNA sequencing data—Application to the ErbB Receptor Signaling Pathway”
<お問い合わせ先>
<研究に関すること>
岡田 眞里子(オカダ マリコ)
大阪大学 蛋白質研究所 細胞システム研究室 教授
医薬基盤・健康・栄養研究所(NIBIOHN) 細胞ネットワーク制御プロジェクト プロジェクトリーダー
京都大学 化学研究所 バイオインフォマティクスセンター 客員教授
<JST事業に関すること>
水田 寿雄(ミズタ ヒサオ)
科学技術振興機構 未来創造研究開発推進部
<報道担当>
科学技術振興機構 広報課