2021-04-28 理化学研究所
理化学研究所(理研)生命医科学研究センターメタボローム研究チームの有田誠チームリーダー、岡橋伸幸客員研究員、安田柊研修生(研究当時)らの研究チームは、未知代謝物を含めた網羅的な解析が可能なノンターゲット質量分析法[1]と、未知分子の構造推定を支援するMolecular spectrum networking[2]技術を組み合わせることで、腸内細菌叢が作り出す複雑な脂質多様性を解明する新しいリピドミクス解析[3]手法を開発しました。
本研究成果は、腸内細菌叢と宿主との相互作用から形成される複雑な脂質代謝ネットワークの解明や、宿主の生体恒常性や疾患の発症に関わる新しい機能性代謝物の発見に貢献すると期待できます。
今回、研究チームは、マウスの糞便を試料に、未知分子も含めて網羅的な計測ができるノンターゲット質量分析法を適用しました。約6,100個に及ぶ大規模スペクトルデータに対し、構造類似分子をクラスタリングするMolecular spectrum networking技術を適用することで、互いに似た構造を持つ分子群を特定し、構造解析するべき対象を迅速に可視化することに成功しました。さらに、その中から既存のデータベースに収載されていない新しい腸内細菌脂質Acyl Alpha-Hydroxy Fatty Acid(AAHFA)を見いだし、その産生に関わる細菌を推定しました。
本研究は、科学雑誌『iScience』(2020年12月18日付掲載)、および『STAR Protocols』オンライン版(2021年4月23日付)に掲載されました。
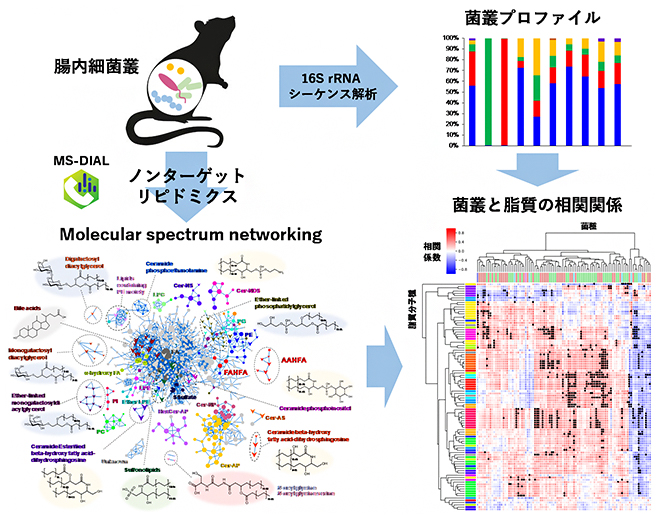
ノンターゲットリピドミクス技術を用い、マウス腸内細菌叢と脂質の関連を明らかにした
背景
哺乳類の腸管内には、数千種にも及ぶ共生細菌が生息しています。腸内細菌は哺乳類とは異なる代謝経路を持ち、それらが産生するユニークな代謝物が宿主の生体恒常性の維持や疾患の発症に関わっていると考えられています。特に、菌体表層を覆う脂質成分には、宿主のパターン認識受容体[4]を介して炎症を引き起こす分子が数多く知られています。これらの脂質には、炭素鎖の長さや二重結合の位置、立体構造などの違いによって数万から数十万に上る多様な分子種が存在し、その構造的な差異が受容体との親和性を左右します。この構造の特殊性と多様性ゆえに、これまで腸内細菌叢が形成する脂質の全体像を捉えることはできませんでした。
研究手法と成果
研究チームは、マウスの腸内細菌を対象に、質量分析によるノンターゲットリピドミクス注1)を実施しました。従来は特定の分子の検出に特化したターゲット解析[5]が汎用されてきたのに対し、ノンターゲット解析[5]では、あらかじめ分析対象を限定する必要がなく、腸内細菌叢が産生する未知の分子を捉えるのに適しています。
まず、マウスの糞便から抽出した総脂質を液体クロマトグラフィー四重極飛行時間型質量分析装置(LC-QTOF/MS)[6]で分析したところ、未知分子も含めて約6,100個に及ぶ包括的なMS/MSスペクトル[7]データを取得できました。既存のデータベースを用いて、これらの分子の同定を試みたところ、そのうちの実に90%が同定できませんでした。これは、多くの未同定分子が腸内細菌によって産生されていることを意味します。
これらの中から腸内細菌が産生する脂質を効率良く同定するために、似た構造の脂質分子は似たMS/MSスペクトルを示すという性質に注目し、従来天然物の構造解析に利用されてきたMolecular spectrum networking技術をリピドミクスデータに応用しました。この方法は、LC-QTOF/MSで取得した任意の二つのMS/MSスペクトル間の類似度を計算し、高い類似性を示すイオン同士を線でつないだネットワークを作成することで、脂質分子群の全体像を可視化します(図1)。
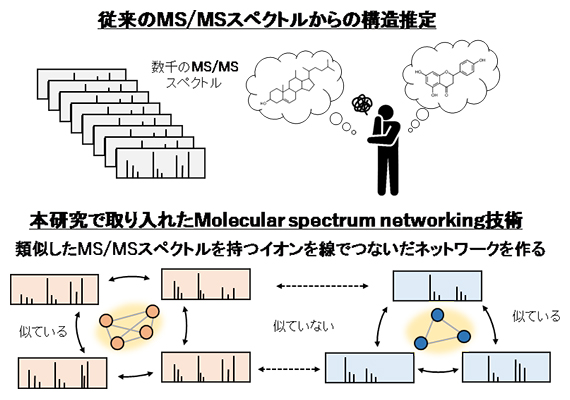
図1 MS/MSスペクトルからの化学構造推定
(上)従来のMS/MS解読作業。これまでは数千個に及ぶMS/MSスペクトルを一つずつ人の目で見て構造推定してきたが、これには熟練の経験と膨大な時間を要する。
(下)Molecular spectrum networking技術の概要。この方法では構造推定をする前に、あらかじめ似たMS/MSスペクトルを持つイオンをまとめることができるため、構造類似体を一度に釣り上げることができ、効率的な構造推定を可能にした。
これによって、構造未知のままでも、似たMS/MSスペクトルを示すイオンを分類することが可能になります。この方法を適用することで、構造類似分子からなると推定されるクラスターが複数発見されました(図2)。
次に、このデータに、抗生物質カクテル投与で腸内細菌を死滅させたマウスの糞便の分析結果を統合することで、腸内細菌が産生に関わる脂質を炙り出しました。抗生物質投与で量が減少したイオンは、腸内細菌由来の代謝物であることが強く示唆されるため、優先的に構造解析するべき対象として絞り込むことができます。これらのMS/MSスペクトルを読み解くと、エーテル結合含有グリセロ脂質[8]、極性頭部にホスホエタノールアミンやホスホイノシトールなどの修飾を持つスフィンゴ脂質[9]、炭素-硫黄原子が直接結合したスルホノリピド[10]、脂肪酸がアミノ酸で抱合されたN -アシルアミド[11]など、腸内細菌依存的に変動する多様な構造の脂質分子群であることが明らかになりました(図2)。
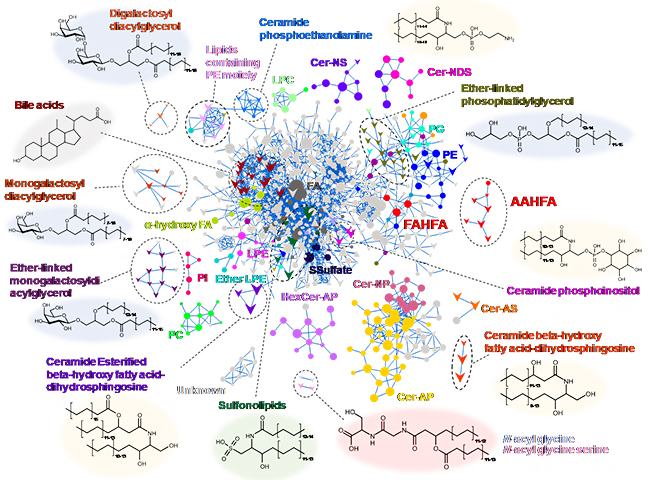
図2 マウス糞便中脂質のMolecular spectrum networkingの結果
それぞれの丸が検出された脂質分子種を表し、MS/MSスペクトルが類似しているもの同士を線でつないだ。さらに、抗生物質投与で検出量が10分の1以下に減少したものを下三角印で表記したところ、それらが独立したクラスター(黒点線で囲んだ部分)を形成していることが分かった。これらの構造(楕円内)を推定した結果、宿主の代謝酵素では産生されないユニークな脂質が含まれていることが明らかになった。
また、これらの中から、酢酸、プロピオン酸、酪酸などの短鎖脂肪酸[12]がα位にヒドロキシ基のある極長鎖脂肪酸[12]とエステル結合した化合物と推定された非常にユニークな新しい脂質Acyl Alpha-Hydroxy Fatty Acid(AAHFA)を見いだしました。そして、実際に有機合成し、その推定構造が正しいことを実証しました(図3)。
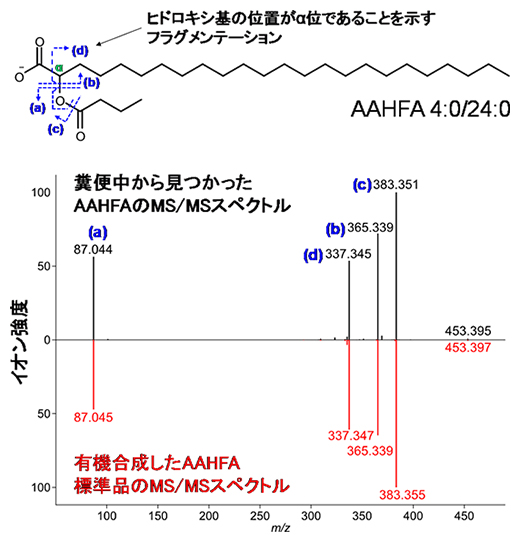
図3 新しい腸内細菌脂質AAHFAのMS/MSスペクトルと分子構造
(上)AAHFAの推定構造。糞便試料中から検出されたMS/MSスペクトルを読み解いた結果、炭素数24の極長鎖脂肪酸のα位のヒドロキシ基に炭素数4の短鎖脂肪酸がエステル結合したユニークな構造をしていると推定された。
(下)糞便試料中のAAHFAと有機合成AAHFAのMS/MSスペクトルの比較。糞便試料中のサンプル中のAAHFAと化学的に合成したAAHFAのMS/MSスペクトルが非常によく一致しており、AAHFAの構造が推定通りであることを実証できた。
さらに、今回見いだした腸内細菌依存的に変動する脂質の産生に関わる細菌種を推定するために、特定の菌群だけを死滅させるさまざまな抗生物質を別のマウスに与え、腸内細菌叢にバリエーションのあるマウスを作製しました。このマウスの糞便に、ノンターゲット質量分析と16S rRNAシーケンス解析[13]を適用し、脂質分子と細菌種の相関解析を行うことで、今回見いだした脂質群がそれぞれ特定の菌種群と正の相関を示すことが明らかになりました(図4)。
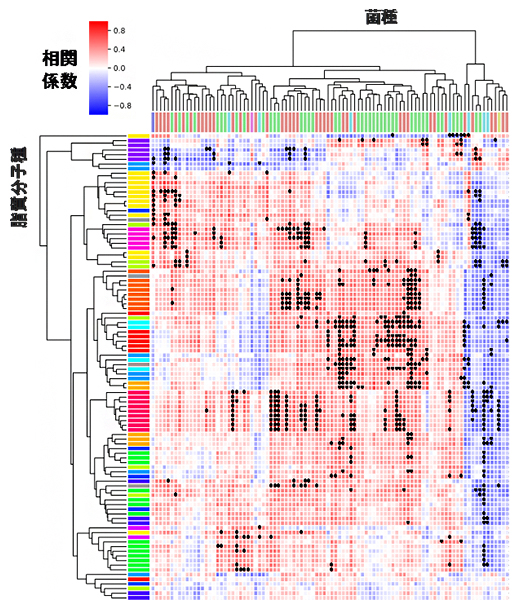
図4 腸内細菌依存性を示す脂質分子種と菌種の相関
さまざまな抗生物質を投与することで、腸内細菌叢にバリエーションを持たせた複数のマウスの糞便試料を分析し、脂質分子種と菌種の量の相関解析を実施した。異なる色のカラーバーはそれぞれ異なる脂質クラス(縦)および菌群(横)を、●(黒点)は特に有意な相関(p値<0.0001)を表す。腸内細菌依存的に産生される脂質分子種が特定の菌群と有意な正の相関を示すことが明らかとなり、それらの脂質の産生に関わる菌種の推定に成功した。
注1)2020年6月16日プレスリリース「生命の脂質多様性を解明」
今後の期待
今回の研究では、ノンターゲット質量分析法とMolecular spectrum networking技術を組み合わせることで、複雑な構造の脂質が腸内細菌によって産生されていることを明らかにしました。このような従来のターゲット解析では見落としていた分子群を正しく捉えることは、がん、肥満、アレルギーなどの種々の疾患で変動する細菌種の意義を理解することにつながると考えられます。
本研究で開発した手法は、そのような菌叢の変化を脂質の量および質の変化として俯瞰的に捉えることを可能にする技術であり、腸内細菌の関与する生命現象を支配する機能性の実行因子を特定することに寄与すると期待できます。
補足説明
1.質量分析法
物質をイオン化し、そのイオンを検出することで分析対象の質量を計測する分析法。イオンの量が試料中の測定対象の量に対応する。分析対象をイオン化後、エネルギーを加えてイオンを断片化し、それらを検出するタンデム型質量分析装置では、分析対象の化学構造も推定できる。
2.Molecular spectrum networking
二つのMS/MSスペクトル間の類似度を計算し、一定以上の類似性を示すイオン同士をつなぎ合わせたネットワークを作成する質量分析データの情報科学的な解析手法。
3.リピドミクス解析
脂質分子種の総体(リピドーム)を網羅的に計測する解析手法のこと。
4.パターン認識受容体
生体に侵入した外的をいち早く感知し排除する自然免疫系を活性化させる受容体で、微生物の持つ共通した分子構造を認識する。
5.ターゲット解析、ノンターゲット解析
ターゲット解析は、あらかじめ質量分析装置で測定したいイオンを指定し、それを高感度かつハイスループットに測定する計測法であり、広く用いられている。一方、注目する対象以外を計測していないため、想定していない分子を見落とすリスクをはらんでいることから、あらかじめ対象を限定しないノンターゲット解析が注目されている。ノンターゲット解析は包括的な計測が可能だが、大規模データの解析が煩雑となる。
6.液体クロマトグラフィー四重極飛行時間型質量分析装置(LC-QTOF/MS)
タンデム型質量分析装置の一種である四重極飛行時間型質量分析装置を液体クロマトグラフィーと組み合わせた分析装置。クロマトグラフィーで物性の違いに応じて分子が分離された後、それらは質量分析装置に導入され、イオン化される。分析対象のイオンの量、およびその断片情報を同時取得する分析モードに長けており、ノンターゲット解析を行う際に汎用されている。
7.MS/MSスペクトル
タンデム型質量分析装置で特定のイオンを選択後、断片化して生じたイオン群を計測したスペクトル。厳密にはプロダクトイオンスペクトルという。
8.グリセロ脂質
生体で見られる代表的な脂質クラス。通常グリセロール分子に脂肪酸がエステル結合した構造をとる。
9.スフィンゴ脂質
グリセロ脂質と並ぶ代表的な脂質クラス。スフィンゴイド塩基と脂肪酸がアミド結合した構造をとり、通常哺乳類では極性頭部に糖やホスホコリンが結合している。
10.スルホノリピド
スフィンゴ脂質と似ているが、スフィンゴイド塩基部分が、硫黄原子が直接炭素原子と結合したカプノイド塩基に置換された構造の分子群。
11.N -アシルアミド
脂肪酸のカルボキシ基にグリシンやセリン、オルニチンなどがアミド結合した分子群。
12.短鎖脂肪酸、極長鎖脂肪酸
脂肪酸の鎖長による分類で、おおむね炭素数6以下を短鎖脂肪酸、20以上を極長鎖脂肪酸と呼ぶ。
13.16S rRNAシーケンス解析
生物の系統分類に使われる16S rRNA遺伝子の配列を解読し、データベース上の既知の細菌の配列と比較することで、試料中に存在する細菌を調べる解析手法。
研究チーム
理化学研究所生命医科学研究センター
メタボローム研究チーム
チームリーダー 有田 誠(ありた まこと)
(慶應義塾大学 薬学部 代謝生理化学講座 教授)
(横浜市立大学 大学院生命医科学研究科 大学院客員教授)
客員研究員 岡橋 伸幸(おかはし のぶゆき)
(大阪大学 大学院情報科学研究科 准教授)
研修生(研究当時) 安田 柊(やすだ しゅう)
(北里大学 薬学部 衛生化学教室 助教)
研究員(研究当時) 津川 裕司(つがわ ひろし)
(理研 環境資源科学研究センター メタボローム情報研究チーム 研究員(研究当時))
(東京農工大学大学院グローバルイノベーション研究院 准教授)
客員研究員 池田 和貴(いけだ かずたか)
(かずさDNA研究所)
客員研究員 上田 政宏(うえだ まさひろ)
(JSR株式会社)
マイクロバイオーム研究チーム
チームリーダー(研究当時) 服部 正平(はっとり まさひら)
副チームリーダー 須田 亙(すだ わたる)
研究員 緒方 勇亮(おがた ゆうすけ)
研究支援
本研究は、日本学術振興会(JSPS)科学研究費補助金新学術領域研究(研究領域提案型)「脂質クオリティが解き明かす生命現象(領域代表者:有田誠)」、同基盤研究(B)「統合オミクス研究に資する質量分析インフォマティクスによる新規代謝制御機構の解明(研究代表者:津川裕司)」、日本医療研究開発機構革新的先端研究開発支援事業インキュベートタイプ(AMED-LEAP)「腸内細菌株カクテルを用いた新規医薬品の創出(研究開発代表者:本田賢也)」による支援を受けて行われました。
原論文情報
Shu Yasuda, Nobuyuki Okahashi, Hiroshi Tsugawa, Yusuke Ogata, Kazutaka Ikeda, Wataru Suda, Hiroyuki Arai, Masahira Hattori & Makoto Arita, “Elucidation of gut microbiota-associated lipids using LC-MS/MS and 16S rRNA sequence analyses”, iScience, 10.1016/j.isci.2020.101841
Nobuyuki Okahashi, Masahiro Ueda, Shu Yasuda, Hiroshi Tsugawa & Makoto Arita, “Global profiling of gut microbiota-associated lipid metabolites in antibiotic-treated mice by LC-MS/MS-based analyses”, STAR Protocols, 10.1016/j.xpro.2021.100492
発表者
理化学研究所
生命医科学研究センター メタボローム研究チーム
チームリーダー 有田 誠(ありた まこと)
客員研究員 岡橋 伸幸(おかはし のぶゆき)
研修生(研究当時) 安田 柊(やすだ しゅう)
報道担当
理化学研究所 広報室 報道担当