「環境」から微生物を検索し、微生物から「環境」を予測するウェブツール「LEA」を開発
2018-06-19 国立遺伝学研究所
Latent environment allocation of microbial community data
Koichi Higashi, Shinya Suzuki, Shin Kurosawa, Hiroshi Mori and Ken Kurokawa
PLOS Computational Biology Published: June 6, 2018 DOI:10.1371/journal.pcbi.1006143
微生物は様々な環境に存在し、物質循環や宿主の健康などに影響しています。したがって「どのような微生物がどのような環境に生息するのか?」という問いは、環境問題や健康問題の解決の糸口となり得るので世界中で探求されました。その結果、特定の「環境」に生息する微生物の群集についてのデータが大量に蓄積される一方で、その「環境」を弁別する記述が研究者間でまちまちとなっていました。この問題によって、研究者間のデータを横断的に解析することが困難となり、微生物の群集と相互作用する環境因子を包括的に理解することができませんでした。
情報・システム研究機構 国立遺伝学研究所の東光一特任研究員および黒川顕教授らのグループは、微生物群集構造の大規模データから様々な環境と微生物とのつながりを明らかにし、その結果を利用して環境と微生物のつながりを可視化するウェブツール「LEA」(http://leamicrobe.jp/)を開発しました。

本研究では、数万サンプルの微生物群集データと各サンプルの由来環境について研究者が記述した文書(自然言語)を確率モデルで表現し、微生物群集の形成に影響を及ぼす環境を機械学習技術によって抽出しました。抽出された「環境」はそれぞれ固有の「微生物」群集構造と結びついています。この結果をもとに、「環境」に対応する「微生物」を検索する、あるいは逆に「微生物」から「環境」を予測するウェブツール「LEA」を開発しました。LEAは、ヒト共生微生物による疾患の診断や、環境汚染の検出などへの応用が期待できます。
本研究成果は、「PLOS Computational Biology」に平成30年6月6日午後2時(米国東部標準時間)に掲載されました。
本研究は、文部科学省科研費「新学術領域研究『学術研究支援基盤形成』」先進ゲノム解析研究推進プラットフォームの助成を受けて行われました。
本研究成果は、国立研究開発法人科学技術振興機構バイオサイエンスデータベースセンター(JST-NBDC)のライフサイエンスデータベース統合推進事業(統合化推進プログラム)における研究開発課題「データサイエンスを加速させる微生物統合データベースの高度実用化開発」の一環として開発されました。
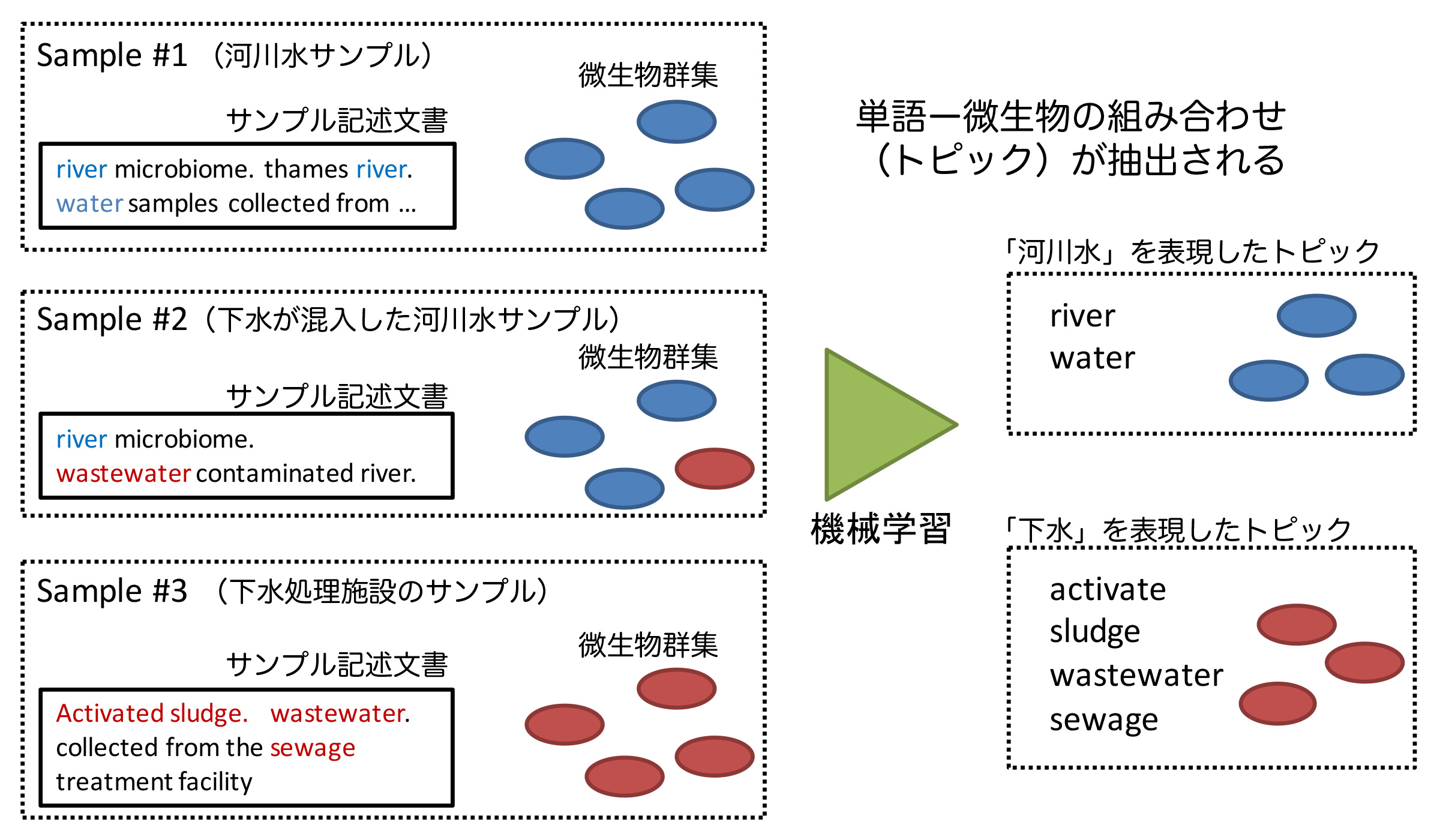
図1: 微生物と自然言語の対応関係を学習
DDBJなどの公共データベース中のすべてのメタゲノムデータには、「サンプルについて研究者が記述した文書(自然言語)」が付随する。
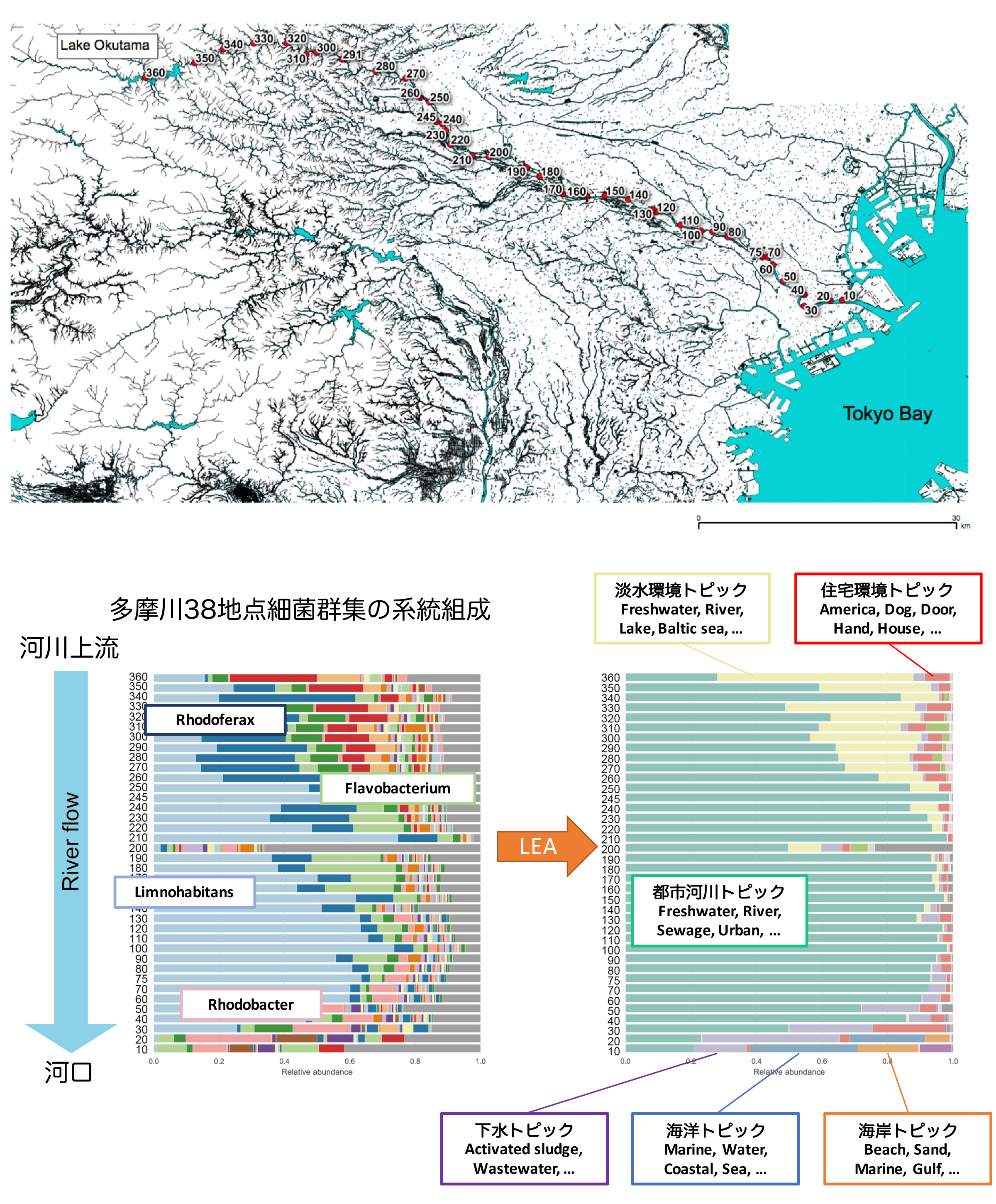
図2:微生物群集構造データからの環境予測
テスト対象:多摩川細菌群集
上流(奥多摩湖)から河口(羽田空港付近)まで38サンプルを解析