2022-03-22 量子科学技術研究開発機構
発表のポイント
- [新しい技術]生きた細胞の中にある60億塩基対ものDNA鎖上にごくわずかに生じた一つひとつの損傷を見つけ出し、原子間力顕微鏡で直接観察する技術を確立。従来の蛍光顕微鏡レベルでは不可能であった、小さな損傷の分布を観察して傷の構造を見分けることを可能にした。
- [新しい発見]放射線によって生じたさまざまな傷が、自然な細胞の働きで修復される速度を調べた結果、特に修復されにくいタイプの傷の構造を明らかにした。
- [今後の期待]一般にDNA損傷の修復エラーが原因とされる、がんや細胞老化のメカニズム解明や、より効果的にがん細胞を殺す治療の確立に、本技術と発見が貢献するものと期待。
国立研究開発法人量子科学技術研究開発機構(理事長 平野俊夫)量子生命・医学部門 量子生命科学研究所 DNA損傷化学研究グループの中野敏彰主幹研究員、赤松憲グループリーダー、鹿園直哉統括グループリーダーらは、広島大学(学長 越智光夫)の井出博名誉教授と共同で、細胞中に生じたDNA損傷部分を極めて長いDNA鎖から新規に開発した特殊技術で取り出し、原子間力顕微鏡(AFM)1)を用いて直接観察することに世界で初めて成功しました。
老化やがんのメカニズムの解明や、効果的ながんの治療法の開発にとって、細胞内のDNA損傷とその修復は本質的なテーマです。細胞内のDNAは環境中のさまざまな要因により日々傷ついており、そのほとんどは細胞の働きによって修復されますが、ごく一部の傷は元通りになりません。これが細胞死、老化やがんの原因と考えられています。また、重粒子線治療などの放射線がん治療においては、がん細胞を効果的に死滅させるべく、修復されにくいDNA損傷をがん細胞内に起こすことが望まれます。こうした研究を進めるためには、DNAに生じた損傷を一つずつ観察して、それぞれの構造的な特徴を明らかにし、その各々が細胞内でどのくらい修復されやすいのか、あるいは修復されにくいのかを調べる必要があります。しかし、従来用いられてきたマイクロメートル3)レベルの解像度しかない蛍光顕微鏡による可視化技術では、原理的に不可能でした。
そこで研究グループは、ナノメートル3)レベルの解像度を持つAFMで直接DNAの損傷個所を観察するべく、この課題に取り組みました。生きた細胞中にある60億塩基対ものDNA鎖上にごくわずかに生じた損傷部位を見つけ出すため、取り出した長いDNAをAFMで観察可能なサイズまで細分し、得られた膨大な量のDNA断片から損傷を含んだDNA断片のみを集める手法を新たに開発しました。これにより、損傷部分のみを探し当てAFMで直接観察することに初めて成功しました。
DNAに生じた個々の損傷をAFMで可視化できたことで、これまで不可能だったDNA損傷の種類分け、例えば、孤立塩基損傷(周辺に別の損傷のない塩基損傷)、クラスター損傷4)(複数の集中した損傷)などを区別できるようになりました。また、直接観察に成功したことで、細胞中に生じた多様なDNA損傷がそれぞれどのくらいの速さで修復されていくのかを解析することも可能になりました。実際に重粒子線によって生じたDNA損傷の修復性を調べた結果、DNAの二本鎖が切断された末端付近にさらに別の塩基損傷が存在するタイプでは極めて修復が困難、つまり細胞にとって致命的な損傷となることが明らかになりました。
細胞内のDNA損傷を一つひとつ調べることを可能とした本成果は、これまで不可能であったDNA損傷の構造それぞれに対応したDNA修復過程や発がんメカニズムの解明、老化を引き起こす原因の解明、効果的にがん細胞を殺す放射線治療の確立等に役立つと考えられます。
本研究は、科学研究費助成事業の基盤研究(C)、新学術領域などの支援を受けて実施されたもので、米国科学アカデミー発行の「Proceedings of the National Academy of Sciences of the United States of America」のオンライン版に令和4年3月22日(日)5:00(日本時間)に掲載されました。
研究開発の背景と目的
私たちの細胞中のDNAは、放射線等、環境中のさまざまな要因によって常に傷ついています。それらの多くは元通りに修復されますが、中には元通りに修復できない損傷のタイプがあり、それが細胞死、突然変異、がん化を引き起こします。しかしながら、細胞中の極めて長いDNA鎖からごくわずかしか存在しない損傷部位を見つけ出すことは困難で、損傷個所を直接観察することはもちろん、実際にどのような損傷が修復されにくいのか、具体的な情報を得る術はこれまでありませんでした。DNA損傷の一形態である二本鎖切断が生成した時に起こるとされる生化学的な現象を、蛍光顕微鏡によってマイクロメートルオーダーで観察する方法は既にありますが、損傷そのものを直接観察するにはナノメートルオーダーの解像度が必要で、蛍光顕微鏡では原理的に不可能です。より具体的な情報を得るためには、DNAにできた損傷一つひとつを目で見てナノメートルオーダーで観察することによって、多様な損傷を種類分けし、それぞれの損傷が細胞中でどのように修復されていくか、あるいは修復できないのかを個別に追跡する必要があります。そこで研究グループは、生きた細胞中のDNAに生じた損傷を直接観察するための新しい技術の開発に取り組みました。
研究開発の内容
放射線照射したヒトリンパ芽球(TK6)細胞5)からDNAを取り出し、塩基に生じた損傷を特殊な酵素DNAグリコシラーゼ6)で切り出します。塩基が切り出された部分(塩基欠損部位:AP)を、APに特異的に化学結合する薬剤ARP7)で標識した後、そのDNA試料を制限酵素8)で処理することによってAFMで観察可能なサイズに切断します。この段階で、損傷を含むDNA断片と、損傷を含まないDNA断片が生じます。両者を含んだ溶液を、ARPと強固に結合するアビジン9)を担持させたビーズ(磁性粒子)を用いて、損傷をもつDNA断片のみを集めます。このアビジンで標識された断片をAFMで観察します(図1参照)。図2に実際に観察された代表的なAFM画像を示しました。
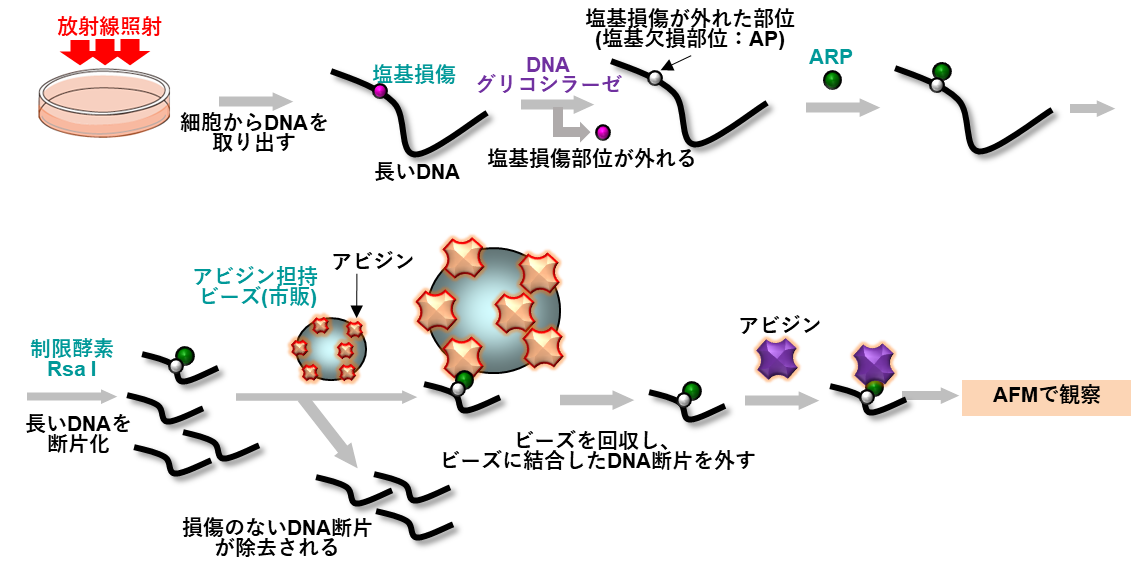
図1.細胞中の長いDNAから損傷を含むDNA領域のみを集めてAFM観察する方法
図では、長いDNA鎖一本の処理過程しか描いていませんが、実際は多くの細胞から長いDNAを取り出し、まとめて上記の処理を行います。
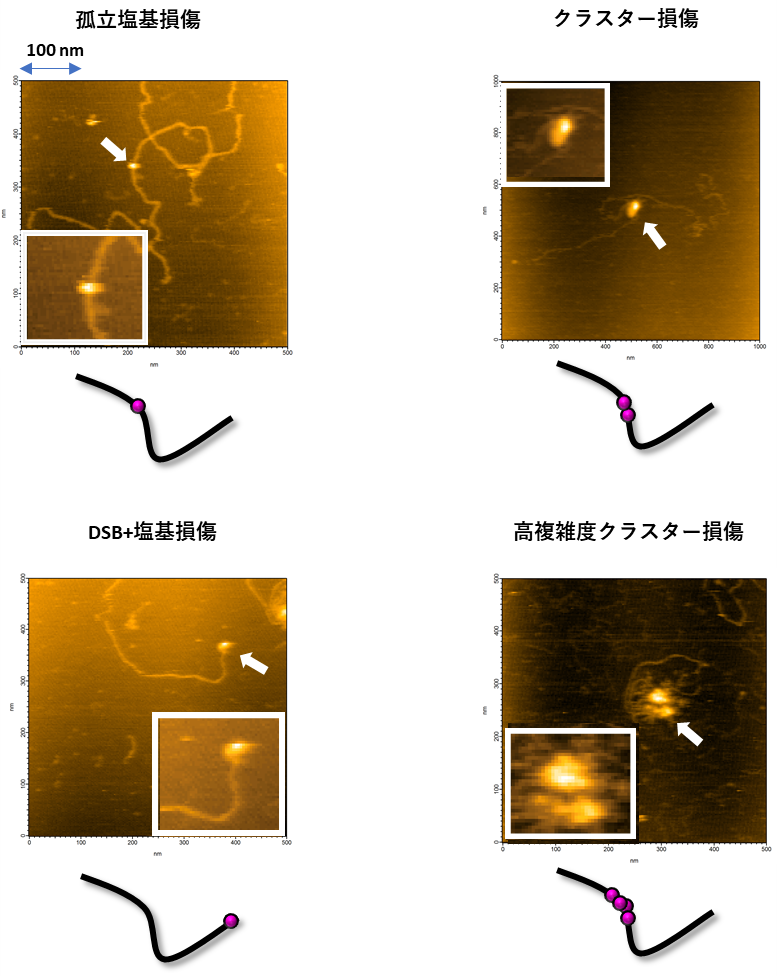
図2.本技術を用いて撮像したさまざまなDNA損傷形態のAFM画像
糸状に見えるDNA鎖に、明るいドット(損傷に結合しているアビジン)が確認できます。これを観察することで、DNA損傷の位置を可視化することができました。観察の結果、通常の孤立した塩基損傷以外に、塩基損傷が集中して生じた領域であるクラスター損傷、DNAの末端に塩基損傷があるタイプの損傷、塩基損傷が複数個固まったような高複雑度クラスター損傷など、多彩なDNA損傷を見ることができ、損傷の「種類分け」に成功しました。
次に個々のDNA損傷の修復性を評価しました。生きた細胞に放射線を照射した後、さらに所定時間培養を行い修復の速さを調べました。重粒子線の一種である鉄イオン線10)の場合、クラスター損傷は孤立塩基損傷と同程度の速度でゆっくりと修復され、6時間で8割程度が修復されました。しかし、塩基損傷を持つ二本鎖切断(DSB)(以下、複雑DSB)は、なかなか修復されないことがわかりました(図3参照)。一方、同様の実験をエックス線10)でも行いましたが、いずれの形態のDNA損傷も、修復時間の増加と共に損傷が減少し、1時間で約半数が修復され、6時間後では約8割が修復されることがわかりました。複雑DSBの質が鉄イオン線とエックス線で異なると考えられますが、実際鉄イオン線で生じる複雑DSBには塩基損傷が最大7つ結合したタイプが観察されています。複数個の塩基損傷をもつDSBのような、いわば「高複雑度DSB」が、細胞にとって致命的なDNA損傷といえます。
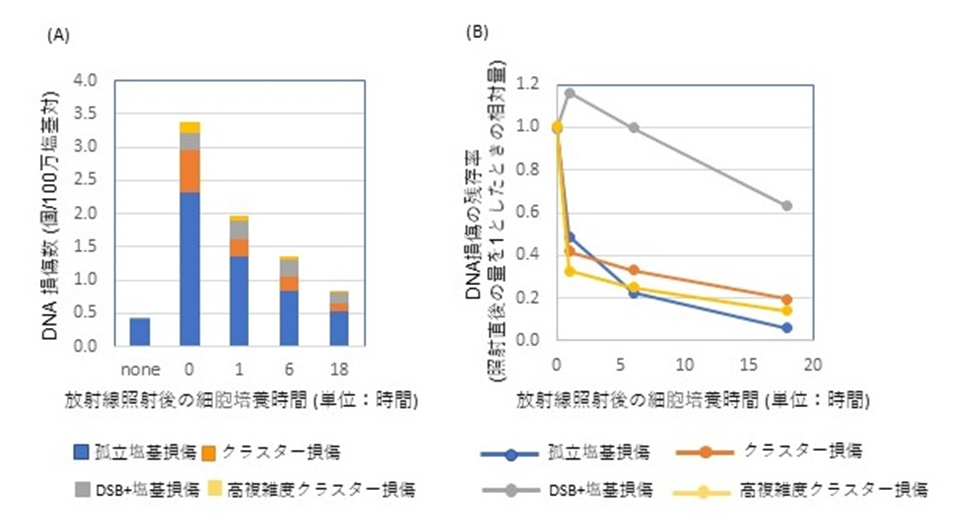
図3.鉄イオン線照射した細胞に生じたさまざまなDNA損傷の修復性(A:各種DNA損傷量の修復時間依存性、B:照射直後の各DNA損傷量を1としたときの相対量変化)
今後の展開
細胞内のDNA損傷を一つひとつ調べることを可能とした本成果により、DNA損傷の特徴に応じてその修復速度を調べることができるようになりました。
炭素線などの重粒子線を用いた放射線治療は、従来のエックス線、ガンマ線と比べて治療効果が高いと言われています。これは、重粒子線が細胞に照射されることによって「クラスター損傷」が引き起こされることが理由とされていましたが、その具体的な知見はこれまで得られていませんでした。今回の成果により、一般に修復がされにくい損傷の形態が明らかになったこと、また様々な条件下において重粒子線によって引き起こされる損傷とその修復されにくさを解析する手段が得られたことから、今後、治療効果をより一層向上させることに貢献できると期待されます。
他にも、今回開発した技術をさらに発展させることにより、発がんメカニズムの解明、老化を引き起こす原因の解明が可能になると考えられます。また、DNA修復に関するこれまでの分子生物学研究により、修復経路は一つではなく複数存在することがわかっています。今後は、DNA損傷の特徴に合わせて、それぞれどういった修復メカニズムが働きやすい、あるいは働きにくいのかを明らかにしていきたいと考えています。
論文掲載情報
Formation of clustered DNA damage in vivo upon irradiation with ionizing radiation: Visualization and analysis with atomic force microscopy
Toshiaki Nakano, Ken Akamatsu, Masataka Tsuda, Ayane Tujimoto, Ryoichi Hirayama, Takeshi Hiromoto, Taro Tamada, Hiroshi Ide, and Naoya Shikazono
Proceedings of the National Academy of Sciences of the United States of America
用語解説
1)原子間力顕微鏡(AFM)
観察対象とする試料の表面を一定の間隔を維持して探針でなぞることで、観察したい試料を構成している原子と、探針の先の原子との間にはたらく力を検出し、それを画像化するための装置
2)塩基(対)
ヌクレオチドと呼ばれるリン酸、糖、塩基からなる高分子化合物が鎖状に連なったもの(ポリヌクレオチド鎖)を核酸と呼び、RNAとDNAが存在する。DNAを構成する塩基には、アデニン(A)、チミン(T)、グアニン(G)、シトシン(C)の4種類あり、この4つの「文字」の並び(塩基配列)が遺伝情報を表している。塩基損傷とは、通常のDNA塩基の一部がさまざまな要因で壊れて化学構造が変化することを言い、DNA複製や転写の妨げとなるばかりか、突然変異やがん化の原因となる。
3)ナノメートル、マイクロメートル
1ナノメートルは10億分の1メートル。1マイクロメートルは100万分の1メートル。1ナノメートルは1マイクロメートルの1000分の1の長さ。
4)クラスター損傷:
DNAの狭い領域に損傷が多数生じたタイプのDNA損傷
5)ヒトリンパ芽球(TK6)細胞
ヒトのリンパ球細胞であるが、まだ成熟していな段階の幼若な細胞をいう。遺伝子突然変異試験に用いる細胞株としてよく使われている。
6)DNAグリコシラーゼ(塩基除去修復酵素)
DNAを構成する糖(デオキシリボース)と塩基をつないでいる化学結合(N-グリコシド結合)を加水分解する酵素の総称であり、DNA修復過程において傷害のある塩基をDNAから取り除く。塩基が除去されてむき出しになった糖の部分を脱塩基部位(APサイト)という。
7)ARP
Aldehyde Reactive Probe(アルデヒド反応性プローブ)。APサイトのアルデヒド基に対して特異的に結合する部位と、アビジンとの親和性が極めて高いビオチン(ビタミンB7)部位から構成される。
8)制限酵素
DNAの特定の塩基配列を認識して、その部分を切断する酵素。例えば、本研究で用いたRsaIという制限酵素は、DNA上のGTAC(グアニン、チミン、アデニン、シトシン)配列のところのみを認識し、その場所のTとAの間を切断する機能を持っている。
9)アビジン
卵白に含まれるタンパク質の一種。ビオチンと極めて高い親和性をもち、ほぼ不可逆的に結合する。ビオチンとアビジンが互いに特異的に強く結合することは、今回の成果のように目的とする分子のみを狙った回収や可視化などに利用される。
10)鉄イオン線とエックス線
両者とも放射線の一種。鉄イオン線は、鉄のイオンを高速に加速したもの(ビーム)であり、放射線のうち「重粒子線」に分類される。物質にあたると、その速度が速いほど奥まで入り込む。一方、エックス線は可視光と同じく電磁波であり、レントゲン写真撮影や手荷物検査で使われることでおなじみ。両放射線によるDNA損傷のできかたの違いを端的にいうと、前者では大砲で打ち抜いたような大きな傷が一気に生じるのに対し、後者では小さな切り傷が散発的にできていくようなイメージになる。
参考文献
1.A simplified cluster analysis of electron track structure for estimating complex DNA damage yields, Yusuke Matsuya, Toshiaki Nakano, Takeshi Kai, Naoya Shikazono, Ken Akamatsu, Yuji Yoshii, Tatsuhiko Sato, International Journal of Molecular Science, 21(5), 1701, 2020
2.Direct observation of damage clustering in irradiated DNA with atomic force microscopy, Xu Xu, Toshiaki Nakano, Masataka Tsuda,Ryota Kanamoto, Ryoichi Hirayama,Akiko Uzawa,Hiroshi Ide, Nucleic Acids Research, 48(3), e18, 2019