2022-04-06 オークリッジ国立研究所(ORNL)
・この解決策は、1970年代に初めて開発された遺伝子研究ツールであるプロトプラスト融合によってもたらされた。この技術を使ってバチルス菌を交配し、遺伝的に異なるバチルス菌を大量に作り出すことができました。
・プロトプラスト融合は、細胞の外層を剥ぎ取って化学的に融合させ、親の染色体間の組み換えを可能にする古典的な遺伝子工学の手法である。研究者たちは何十年にもわたり、最初は日常的な遺伝子操作の手法として、次に微生物の抗生物質生産などの形質工学のツールとして、この技術を利用してきたが、その根底にある染色体再配列はあまり理解されていなかった。
・Nucleic Acids Research誌によると、ゲノム上で様々な長さの組換えを発見したとのことである。「DNAの小さな断片と大きな断片の両方が交換され、その過程はほとんどランダムであるように見えました。これらはまさに、遺伝子マッピングを可能にするために欲しかった特性です。・チームは、それ以来、合計4回の組み換えを行い、約500の完全に配列決定され、高度にシャッフルされた子孫を生み出しています。ORNLのDan Jacobsonが率いる計算システム生物学チームは、機械学習の手法を導入して、細菌のパネル全体の表現型とマッピングを高速化し、目的の身体的形質とその遺伝的基盤の間の関連性を特定しています。
・その結果、新しい微生物形質マッピング機能が生まれ、例えば、クリーンなバイオ燃料の生産に必要な植物バイオマスの分解に優れた微生物の設計を早めることができるかもしれません。
<関連情報>
- https://www.ornl.gov/news/retro-technique-advances-modern-bacterial-engineering-bioenergy
- https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkac025/6513571
Bacillus属のプロトプラスト融合は、頻繁に、偏りなく、ゲノム全体の相同組換えを起こす Protoplast fusion in Bacillus species produces frequent, unbiased, genome-wide homologous recombination
Delyana P Vasileva, Jared C Streich, Leah H Burdick, Dawn M Klingeman, Hari B Chhetri, Christa M Brelsford, J Christopher Ellis, Dan M Close, Daniel A Jacobson, Joshua K Michener
Nucleic Acids Research Published:21 January 2022
https://doi.org/10.1093/nar/gkac025

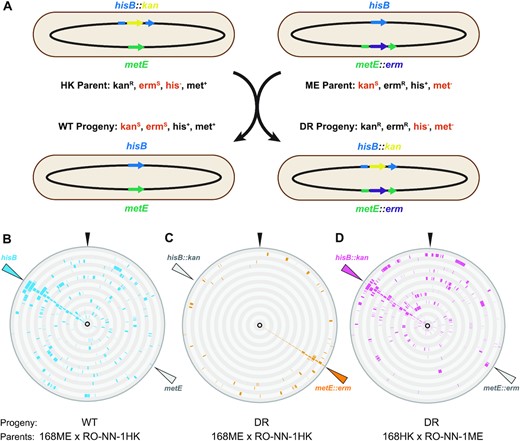
Analysis of genome shuffling in B. subtilis. (A) Replacing amino acid biosynthesis genes with antibiotic resistance markers allows flexible identification of recombinant progeny following genome shuffling. Crossing mutants of 168 and RO-NN-1 yielded prototrophic (B) and double-resistant (C, D) progeny. Each concentric circle represents a different resequenced individual from this cross. The colored bars indicate sequences recombined from strain 168, with the remaining genomic sequence coming from RO-NN-1. Orange, blue, pink and gray arrows indicate locations of selection markers. Black arrows indicate the origin of replication. WT: wild type; DR: double resistant.
Abstract
In eukaryotes, fine-scale maps of meiotic recombination events have greatly advanced our understanding of the factors that affect genomic variation patterns and evolution of traits. However, in bacteria that lack natural systems for sexual reproduction, unbiased characterization of recombination landscapes has remained challenging due to variable rates of genetic exchange and influence of natural selection. Here, to overcome these limitations and to gain a genome-wide view on recombination, we crossed Bacillus strains with different genetic distances using protoplast fusion. The offspring displayed complex inheritance patterns with one of the parents consistently contributing the major part of the chromosome backbone and multiple unselected fragments originating from the second parent. Our results demonstrate that this bias was in part due to the action of restriction–modification systems, whereas genome features like GC content and local nucleotide identity did not affect distribution of recombination events around the chromosome. Furthermore, we found that recombination occurred uniformly across the genome without concentration into hotspots. Notably, our results show that species-level genetic distance did not affect genome-wide recombination. This study provides a new insight into the dynamics of recombination in bacteria and a platform for studying recombination patterns in diverse bacterial species.