人間の脳からヒントを得た人工ニューラルネットワークが、人間の目よりも早く、遺伝子配列の微小な変異を発見するよう訓練された。 Inspired by the human brain, an artificial neural network was trained to spot tiny mutations in genetic sequences better and faster than human eyes
2023-01-02 カリフォルニア大学サンディエゴ校(UCSD)
現在のDNA変異ソフトウェア検出器は、ヒトゲノムの30億の塩基をスキャンする一方で、正常なDNA配列の中に潜むモザイク変異を見分けるにはあまり適していない。多くの場合、医療遺伝学者はDNA配列を目で見て確認し、モザイク状の突然変異を特定・確認しなければならないが、これは間違いの可能性をはらんだ時間のかかる作業である。
カリフォルニア大学サンディエゴ校医学部とRady Children’s Institute for Genomic Medicineの研究者らは、『Nature Biotechnology』誌の2023年1月2日号に、「ディープラーニング」を用いて、コンピュータにモザイク変異の見つけ方を教える方法について述べている。
カリフォルニア大学サンディエゴ校医学部のレディ神経科学教授で、レディ子供ゲノム医学研究所の神経科学研究ディレクターである研究主任のジョセフ・グリーソン医学博士は、「未解決疾患の一例として、焦点てんかんがあります」と述べています。「てんかんは人口の4%が罹患しており、てんかん発作の約4分の1は一般的な薬物療法が効きません。これらの患者さんでは、発作を止めるために、脳の短絡した焦点部分の外科的切除が必要になることがよくあります。 このような患者さんの中には、脳内のモザイク状の変異がてんかんの焦点の原因となることがあります。
私たちはこれまで、原因を特定できないてんかん患者さんを数多く診てきましたが、『DeepMosaic』という手法をゲノムデータに適用したところ、変異が明らかになりました。これにより、ある種のてんかんにおけるDNA配列決定の感度が向上し、脳疾患の新たな治療法を指し示す発見がありました。”
グリーソン氏は、モザイク状の突然変異を正確に検出することは、多くの疾患の治療法開発に向けた医学研究の第一歩だと語っています。
Gleesonの研究室の博士研究員である共同筆頭著者であり共同責任著者であるXiaoxu Yang博士は、DeepMosaicは、「最終的に、これまで出会ったことのないデータから変異体を検出する能力に満足するまで、ゲノム全体のほぼ20万個のシミュレーションと生体変異で訓練されました」と述べています。
コンピュータを訓練するために、著者達は、信頼できるモザイク変異の例と、多くの正常なDNA配列を与えて、コンピュータにその違いを見分けるように教え込みました。より複雑なデータセットで訓練と再訓練を繰り返し、12種類のモデルから選択することで、最終的にコンピューターは、人間の目や以前の方法よりもはるかに優れたモザイク変異を識別できるようになりました。また、DeepMosaicは、これまで見たことのないいくつかの独立した大規模なシーケンスデータセットでテストされ、先行するアプローチを凌駕した。
DeepMosaicは、科学者が自由に利用できるようになっています。それは、単一のコンピュータープログラムではなく、むしろ、他の研究者が、同様の画像ベースの設定を使用して、より的を射た変異の検出を達成するために、独自のニューラルネットワークを訓練することができるオープンソースのプラットフォームであると、研究者は述べています。
<関連情報>
- https://today.ucsd.edu/story/new-computer-program-learns-to-identify-mosaic-mutations-that-cause-disease
- https://www.nature.com/articles/s41587-022-01559-w
DeepMosaicによる制御不能なモザイク一塩基バリアント検出 Control-independent mosaic single nucleotide variant detection with DeepMosaic
Xiaoxu Yang,Xin Xu,Martin W. Breuss,Danny Antaki,Laurel L. Ball,Changuk Chung,Jiawei Shen,Chen Li,Renee D. George,Yifan Wang,Taejeong Bae,Yuhe Cheng,Alexej Abyzov,Liping Wei,Ludmil B. Alexandrov,Jonathan L. Sebat,NIMH Brain Somatic Mosaicism Network & Joseph G. Gleeson
Nature Biotechnology Published:02 January 2023
DOI:https://doi.org/10.1038/s41587-022-01559-w
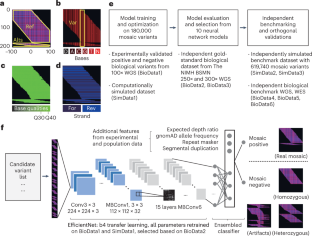
Abstract
Mosaic variants (MVs) reflect mutagenic processes during embryonic development and environmental exposure, accumulate with aging and underlie diseases such as cancer and autism. The detection of noncancer MVs has been computationally challenging due to the sparse representation of nonclonally expanded MVs. Here we present DeepMosaic, combining an image-based visualization module for single nucleotide MVs and a convolutional neural network-based classification module for control-independent MV detection. DeepMosaic was trained on 180,000 simulated or experimentally assessed MVs, and was benchmarked on 619,740 simulated MVs and 530 independent biologically tested MVs from 16 genomes and 181 exomes. DeepMosaic achieved higher accuracy compared with existing methods on biological data, with a sensitivity of 0.78, specificity of 0.83 and positive predictive value of 0.96 on noncancer whole-genome sequencing data, as well as doubling the validation rate over previous best-practice methods on noncancer whole-exome sequencing data (0.43 versus 0.18). DeepMosaic represents an accurate MV classifier for noncancer samples that can be implemented as an alternative or complement to existing methods.

