2024-04-15 理化学研究所,東京工業大学
理化学研究所(理研)生命機能科学研究センター 先端NMR開発・応用研究チームの大山 貴子 研究員、石井 佳誉 チームリーダー(東京工業大学 生命理工学院 生命理工学系 教授)らの研究チームは、SARS-CoV-2のゲノム転写開始に関与する3′非翻訳領域[1]で実験的には存在が確認されていなかったユニークなRNA構造[2]が形成可能であることを示しました。
本研究成果は、ヒトやその他の動物のコロナウイルス感染症の予防薬や、感染後に症状を軽いものにとどめる薬の開発に貢献すると期待できます。
SARS-CoV-2やMERS[3]を含むベーターコロナウイルス[3]のRNAゲノムには、タンパク質の遺伝情報を持たない非翻訳領域と呼ばれる部位が存在しています。この部位は、特定のRNA構造を形成して、RNAポリメラーゼ[4]などのタンパク質との結合の制御に関与していることが知られています。コロナウイルスの3′側の非翻訳領域は二種類の構造を形成し、構造がスイッチすることでRNAの複製が開始すると考えられている領域があります。
研究チームは今回、二種類のRNA構造のうち、実験的に存在が確認されていなかったシュードノット構造[5]といわれる特徴的な構造が形成可能であることを確認し、高磁場NMR[6]を用いて原子レベルでその2次構造を同定しました。
本研究は、科学雑誌『JACS au』オンライン版(3月20日付)に掲載されました。
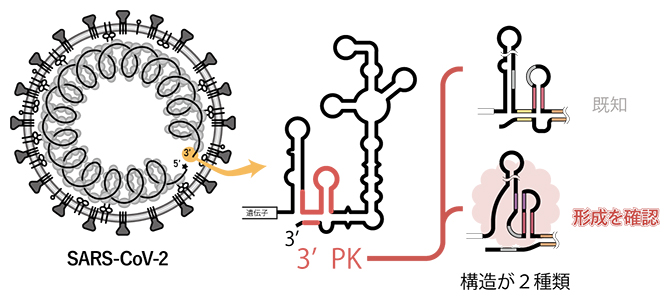
SARS-CoV-2ウイルスRNAの転写開始モデルの模式図
背景
COVID-19の原因ウイルスであるSARS-CoV-2は、1本鎖RNAのゲノムを持っています。このRNAゲノムの両端には、タンパク質の情報を持たない非翻訳領域と呼ばれる配列が存在します。非翻訳領域はRNAウイルスのゲノムだけでなく、一般的な生物のmRNAにも見られます。非翻訳領域はタンパク質の情報を持ちませんが、RNA構造の形成に必要な配列を含んでおり、転写、翻訳、エピジェネティック制御[7]などの生体中で起こるさまざまな反応に関与していることが近年明らかになりつつあります。
SARS-CoV-2を含むベータコロナウイルス属の3′側の非翻訳領域には、ウイルスゲノムの転写開始に必須である3′PKと呼ばれる領域があります。この領域は、極めて遺伝的変異が少なく、この領域のRNA構造が失われるとウイルスが複製不能になります。3′PKは特定の2種類のRNA構造を形成することで、RNA依存RNAポリメラーゼ(RdRp)[4]のゲノムRNAへの結合を制御していると考えられています。具体的には、このRNA領域はステムループ構造[8]をとっており、①そこにRdRpの補助因子(コファクターと呼ばれるタンパク質複合体)がまず結合してRNA転写の準備をします(図1-①)。そうすると、②RNAがシュードノット構造にスイッチし(図1-②)、③そこにRdRpの本体が結合する(図1-③)ことでRdRpが完成するという仮説が提案されています。
これまでの研究で、①のステムループ構造は形成可能であることが示されています。しかし、RNA複製の鍵になる②のシュードノット構造は、過去の研究から形成が予測されていましたが、実験的にその存在が確認されていませんでした。シュードノット構造はRdRpの構成タンパク質の一部と結合した時に形成されると考えられている構造です。言い換えると、通常はステムループ構造がシュードノット構造よりも安定で、RNA複製の準備ができた特別な条件でのみシュードノット構造がステムループ構造よりも安定化すると考えられます。研究チームは、高磁場NMRを用いて3′PKのRNA構造形成を詳しく解析しました。
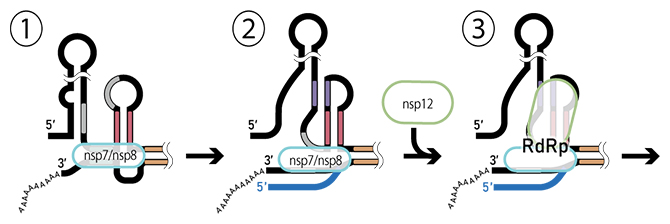
図1 ベータコロナウイルスRNAの転写開始モデルの模式図
ベータコロナウイルス属のゲノム複製(RNAの転写)の模式図。ウイルスゲノムの転写開始に必須である3′側非翻訳領域の3′PK領域の2次構造を図示し、色分けした部分は塩基対を形成する配列の位置を示す。nsp7/nsp8はRNA依存RNAポリメラーゼ(RdRp)のコファクター。nsp12はRdRpの本体。①~③の転写開始の仕組みについては本文を参照。
研究手法と成果
コロナウイルスのゲノムRNAは、そのままでは大きすぎてNMRでの構造解析ができません。そこで研究チームは、部分的に切り出したゲノムRNA配列の中からシュードノット構造を形成できる領域を注意深く探し、溶液も細胞中の条件に似せることで、シュードノット構造を安定な形で観測することを試みました。まず、SARS-CoV-2の3′PK領域と同じ配列を持つRNAを合成し、この合成RNAを細胞中と似た溶液条件下に置くことでシュードノット構造が形成されるかを高磁場NMRを用いて確かめました。NMRでは、RNAが塩基対を形成しているかどうかを10~14ppm[9]の領域に観測されるNMRシグナルの化学シフト[6]から判別することが可能で、これによりRNAの構造が判定できます。3′PK領域の全体を含むPKP4と名付けたRNAと、PKP4の一部が欠けているSLP4と名付けたRNAの2次構造を調べました。その結果、PKP4と名付けたRNAはシュードノット構造を、SLP4と名付けたRNAはステムループ構造を形成していることが分かりました。SLP4が形成するステムループ構造は他の研究グループが過去に報告したSARS-CoV-2の同じ領域の2次構造とよく似ていましたが、3′PK領域でのシュードノット構造の形成は本研究が初めての実験的な報告例となります。
次にシュードノット構造をとるPKP4が、RdRpのコファクターと結合するかを調べ、今まで提唱されていたウイルスゲノムの転写開始の仮説と同じ反応が起こり得るかを確かめました。もし形成が確認されたシュードノット構造のRNAがRdRpと結合すれば、RNAだけあるいはタンパク質だけの時とは異なるNMRシグナルが検出されます。実際に測定すると、シュードノット構造のRNA(PKP4)はRdRpのコファクターと結合することが分かりました(図2左)。同様の実験をステムループ構造RNA(SLP4)でも行ったところ、ステムループ構造もRdRpのコファクターと結合することが分かりました(図2右)。さらにRdRpのコファクターはステムループ構造かシュードノット構造かで異なる構造をしている可能性が判明しました。
以上の結果から、コロナウイルスのゲノムRNAのRdRpが完成するという図1に示したモデルの①と②の反応が起こり得ると実験的に初めて示されました。
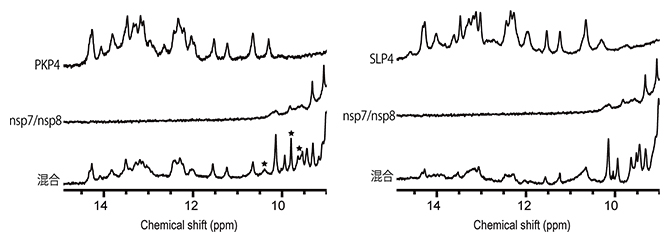
図2 SARS-CoV-2の3′PK領域の部分構造とRdRpコファクターの複合体形成
SARS-CoV-2の3′PK領域とRdRpのコファクターが結合すると、それぞれの構造に変化が起こる。この構造変化により、NMRスペクトルでシグナルが元の位置とは異なる位置に観測される。
左)上から、シュードノット構造をとるPKP4、コファクター(nsp7/nsp8)、およびPKP4とコファクター混合溶液のNMRスペクトル。★は、PKP4とコファクターの混合溶液でのみ観察されたコファクターのスペクトルのピークを示す。
右)上から、ステムループ構造をとるSLP4、コファクター(nsp7/nsp8)、SLP4とコファクターの混合溶液のNMRスペクトル。PKP4とSLP4では混合溶液のスペクトルにおけるコファクターのシグナルが異なったことから、コファクターの立体構造は、結合相手のRNAがシュードノット構造かステムループ構造かで異なることが示唆された。
今後の期待
今回、SAS-CoV-2ゲノムRNAの3′側の非翻訳領域(3′PK領域)に形成が確認された二つのRNA構造は、RdRpのコファクターとの結合に関与することが示唆されました。従ってこの結合を阻害すれば、ウイルスゲノムの転写が開始できなくなり、ウイルス増殖を抑制することができると考えられます。この結合阻害は、今までの抗コロナウイルス薬のメカニズムとは異なった新しい抗コロナウイルス薬の創薬に応用できる可能性があります。また3′PK領域の配列はSAS-CoV-2ウイルス間でかなり変異が少ないものの、まれな変異が起こった場合でもRNA構造は保持したままであることが分かっており、このような変異株に対する創薬デザインにも役立つと考えられます。
本研究ではSAS-CoV-2を用いてRNAのシュードノット構造の形成を確認しましたが、同様のシュードノット構造はベータコロナウイルス属で共通に形成可能であることがRNA配列の比較から推測されます。このシュードノット構造は、COVID-19だけではなく、今後登場が予想されるコロナウイルスが原因の重篤な疾病や、ウシ、ブタ、ネコなどの動物に致死的なコロナウイルスが原因の疾病にも幅広く効果を示す「汎コロナウイルス薬剤」の開発につながる新たな創薬ターゲットとして期待できます。
補足説明
1.非翻訳領域
DNAから転写されたmRNAなど、タンパク質の翻訳に使われるRNAのうちタンパク質に翻訳されない領域。5′側と3′側の両方に存在し、それぞれ5′UTR、3′UTRと呼ぶ。
2.RNA構造
RNAはDNAと同様に塩基対によって構造が形成される。塩基対によって組まれた平面に書き下したRNA構造を2次構造、実際に塩基対で形成された立体的なRNA構造を3次構造と呼ぶ。
3.MERS、ベータコロナウイルス
MERS(Middle East Respiratory Syndrome)は、2012年9月以降に中東地域で広く発生した重症呼吸器感染症。MERS-CoVウイルスを病原体とする。コロナウイルスはα、β、γ、δのグループに分類され、MERS-CoVやSARS-CoV2はベータコロナウイルスに含まれる。
4.RNAポリメラーゼ、RNA依存RNAポリメラーゼ(RdRp)
RNAポリメラーゼはRNAを合成する酵素。一般的な生物はDNAを鋳型にRNAを合成するが、RNA依存RNAポリメラーゼはRNAを鋳型としてRNAを合成する酵素。RNAウイルスなどのごく一部のウイルスにのみ存在する特殊な酵素で、ヒトには存在していない。
5.シュードノット構造
RNAが形成する特殊な構造の一つ。下記[8]で示すステムループと呼ばれる構造のループ部分にもう一つ別のステム構造が入れ子になった構造で、RNAの機能に重要であることが知られている。結び目ではないが結び目のように見えることからシュード(疑似)ノット(結び目)構造と名付けられた。
6.高磁場NMR、化学シフト
強い磁場中に置かれた原子核に電磁波を照射すると、核スピンの共鳴により、原子核の性質や周囲の環境に応じた周波数(共鳴周波数)の電磁波の吸収や放出が起こるが、NMRは、その電磁波をNMR信号として捉えることで、物質の分子構造の解析や物性の解析を行う手法。共鳴周波数は同じ核種であっても周辺の化学環境によって異なるため化学シフトと呼ばれ、ppmの単位で表される。NMRでは磁場が高いほど分解能が高くなり、本研究では最高で900MHz(21.1テスラ)の高磁場NMR装置を用いた。NMRはNuclear Magnetic Resonanceの略。
7.エピジェネティック制御
DNAの塩基配列に依存しない遺伝子発現の制御機構。
8.ステムループ構造
RNAが形成する普遍的な構造。1本のひも状のRNAの両端に塩基対が数個以上連続して形成されると、塩基対の部位をステムと呼び、ステムの間をつなぐ部分をループと呼ぶ。さらに両方合わせてステムループ構造と呼ぶ。
9.ppm
英語で百万分の1を意味する「parts per million」の頭文字をとって作られた単位。
研究チーム
理化学研究所 生命機能科学研究センター
先端NMR開発・応用研究チーム
チームリーダー 石井 佳誉(イシイ・ヨシタカ)
(東京工業大学 生命理工学院 生命理工学系 教授)
研究員 大山 貴子(オオヤマ・タカコ)
転写制御構造生物学研究チーム
チームリーダー 関根 俊一(セキネ・シュンイチ)
研究員 大澤 拓生(オオサワ・タクオ)
研究支援
本研究は、理化学研究所運営費交付金(生命機能科学研究、戦略的研究展開事業)で実施し、科学技術振興機構(JST)未来社会創造事業大規模プロジェクト型「エネルギー損失の革新的な低減化につながる高温超電導線材接合技術」研究領域の研究課題「高温超電導線材接合技術の超高磁場NMRと鉄道き電線への社会実装(研究代表者:小野通隆、JPMJMI17A2)」による助成を受けて行われました。
原論文情報
Takako Ohyama, Takuo Osawa, Shun-ichi Sekine, Yoshitaka Ishii, “NMR Studies of Genomic RNA in 3′ Untranslated Region Unveil Pseudoknot Structure that Initiates Viral RNA Replication in SARS-CoV-2”, JACS au, 10.1021/jacsau.3c00641
発表者
理化学研究所
生命機能科学研究センター 先端NMR開発・応用研究チーム
チームリーダー 石井 佳誉(イシイ・ヨシタカ)
(東京工業大学 生命理工学院 生命理工学系 教授)
研究員 大山 貴子(オオヤマ・タカコ)
報道担当
理化学研究所 広報室 報道担当
東京工業大学 総務部 広報課