2024-05-21 理化学研究所,九州大学
理化学研究所(理研)開拓研究本部 岩崎RNAシステム生化学研究室の岩崎 信太郎 主任研究員、七野 悠一 研究員、生命機能科学研究センター 翻訳構造解析研究チームの伊藤 拓宏 チームリーダー、柏木 一宏 研究員、九州大学 大学院医学研究院 薬理学分野の久場 敬司 教授、山口 智和 助教らの国際共同研究グループは、長年翻訳[1]を促進すると考えられてきた翻訳開始因子[1]eIF4A1が、栄養飢餓時に翻訳に対して抑制的に働くという想定と逆の役割があることを発見しました。
本研究成果は、翻訳の制御機構に対する根本的な理解と、これらの機構が関与するがんなどの疾患の治療法の開発につながるものと期待されます。
メッセンジャーRNA(mRNA)[2]上の遺伝情報を読み取ってタンパク質を合成する翻訳過程で、その頻度を決定する翻訳開始は、多数の翻訳開始因子によって厳密に制御されており、特に重要です。翻訳開始因子の中で最も豊富に存在するのがeIF4Aです。eIF4Aは翻訳を阻害するmRNA上の二次構造[3]を解消して翻訳を促進するために必要と考えられていました。一方、二次構造の解消以外の機能も示唆されていますが、その機能の全貌は不明でした。
国際共同研究グループは、elF4Aの一つであるeIF4A1がRNA結合タンパク質LARP1を介してTOP mRNA[4]に強く結合することを発見しました。LARP1は栄養飢餓時にTOP mRNAの翻訳を強く抑制するために必要な因子ですが、eIF4A1はLARP1とTOP mRNAの結合を強化することで、TOP mRNAに対する翻訳抑制を強めていることが分かりました。
本研究は、科学雑誌『Nature Structural & Molecular Biology』オンライン版(5月21日付:日本時間5月21日)に掲載されました。
背景
mRNA上の遺伝情報からタンパク質を合成する翻訳過程のうち、リボソーム[5]がmRNAに誘導されて翻訳開始点を探索する過程を翻訳開始と呼びます。この過程は翻訳をいつ、どの程度行うかを決定しており、翻訳開始因子と呼ばれる多数の因子によって厳密に制御されています。翻訳開始因子の中でも最も豊富に存在する因子がeIF4Aです。eIF4Aは40年以上前に発見され、翻訳開始を阻害するmRNA上の二次構造を解消し、翻訳を促進すると考えられてきました。しかし、eIF4Aには二次構造の解消以外の機能があることが近年報告され注1)、多面的な機能を持つ因子である可能性が示唆されてきました。
eIF4Aはがんや感染症などの疾患と深く関係しており、eIF4Aを標的とする薬剤注2)も多数開発されています。しかし、その機能には不明な点が多く残されています。そのため、eIF4Aの機能を正確に理解することは疾患治療の観点からも重要な課題です。
注1)Sen, N. D., Zhou, F., Ingolia, N. T. & Hinnebusch, A. G. Genome-wide analysis of translational efficiency reveals distinct but overlapping functions of yeast DEAD-box RNA helicases Ded1 and eIF4A. Genome Res. 25, 1196-1205 (2015).
注2)Zotatifin(eFT226)など。
研究手法と成果
まず、国際共同研究グループはRNA pulldown-Seq法[6]と呼ばれる手法を使って、eIF4Aと結合するRNAを次世代シーケンサー[7]により網羅的に解析しました。ヒト培養細胞HEK293は配列のよく似た2種類のeIF4A(eIF4A1とeIF4A2)を持っていますが、そのうち量の多いeIF4A1に着目して実験を行いました。その結果、eIF4A1はTOP mRNAと特に強く結合することが分かりました(図1A)。
次に、eIF4A1がTOP mRNAと強く結合する機構を探りました。eIF4A1はどのような配列のRNAにも結合するため、TOP mRNAとの結合には何らかの選択的なRNA結合タンパク質が介在していると考えられました。そこで、eIF4A1に結合するタンパク質を質量分析[8]で網羅的に解析したところ、LARP1というRNA結合タンパク質を同定しました(図1B)。LARP1はmRNAのキャップ構造[9]とTOP配列[4]に選択的に結合することが報告されていました。そこで、LARP1の量を低下させたところ、eIF4A1とTOP mRNAの結合が弱まりました(図1C)。よって、eIF4A1はLARP1を介してTOP mRNAに結合していることが分かりました。
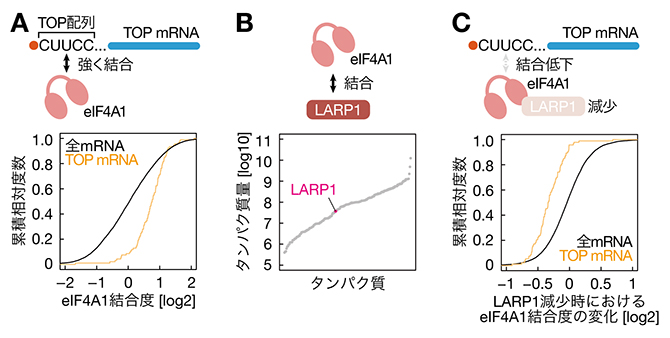
図1 LARP1を介したeIF4A1とTOP mRNAの結合
(A)eIF4A1のTOP mRNAとの強い結合。RNA pulldown-Seq法により、eIF4A1と結合するRNAを網羅的に解析した。TOP mRNAは全mRNAに比べeIF4A1と強く結合する傾向にあった。
(B)eIF4A1とLARP1の結合。質量分析によりeIF4A1と結合するタンパク質を網羅的に同定し、その中にLARP1を発見した。
(C)LARP1を減少させた際のeIF4A1とTOP mRNAの結合変化。LARP1に対しsiRNA(small interfering RNA:RNA干渉をする低分子2本鎖RNA)を用いてその量を減少させ、RNA pulldown-Seq法によりeIF4A1と結合するRNAを網羅的に解析した。TOP mRNAはLARP1減少に応じてeIF4A1との結合度が顕著に低下した。
翻訳はエネルギーを大量に消費するため、細胞は生育環境における栄養の量に応じて翻訳量を厳密に調節しています。TOP mRNAは翻訳制御に重要なタンパク質をコードしており、その翻訳は栄養、特にアミノ酸の飢餓時にLARP1によって急速に抑制されます。アミノ酸が豊富な場合は栄養源認識に重要な因子であるmTOR[10]がLARP1を不活性化するためTOP mRNAの翻訳は効率的に行われていますが、アミノ酸飢餓時にはmTORが不活性化するため、LARP1が活性化され、LARP1とTOP mRNAとが強固に結合して翻訳を抑制します(図2A)。このようにLARP1を介した翻訳抑制機構は細胞がアミノ酸飢餓に対応する上で重要です。しかし、LARP1が単独でTOP mRNAを翻訳抑制しているのかは不明でした。
そこで、翻訳抑制機構におけるeIF4A1の影響を調べるため、アミノ酸飢餓状態においてmTORの阻害剤をeIF4A1欠損細胞に与え、リボソームプロファイリング法[11]を用いて細胞内の翻訳状況を網羅的に解析しました。その結果、eIF4A1欠損細胞ではmTORの阻害時のTOP mRNAの翻訳抑制が弱くなることが分かりました(図2B)。
次に、精製タンパク質を用いてTOP mRNAとLARP1との結合を測定し、翻訳抑制に必要な結合強度を調べました。その結果、eIF4A1がLARP1とTOP mRNAとの結合を強めていることが判明しました(図2C)。
長期間mTORを阻害してTOP mRNAの翻訳を抑制し続けると、細胞の増殖に必要な翻訳が行えなくなり生存率が低下していきます。しかし、eIF4A1欠損細胞では、mTORを阻害してもあまり生存率が低下しませんでした(図2D)。また、マウスのeIF4A1欠損細胞でも同様の結果が見られ、種を超えて保存された機構であることも分かりました。

図2 TOP mRNA翻訳抑制機構におけるeIF4A1の影響
(A)通常時ではmTORがLARP1を不活性するため、TOP mRNAの翻訳は効率的に行われている。アミノ酸飢餓時ではmTORが不活性化するため、LARP1が活性化してTOP mRNAと結合し翻訳を抑制する。
(B)eIF4A1欠損細胞におけるTOP mRNAの翻訳抑制。細胞にmTOR阻害剤を与え、リボソームプロファイリング法によりTOP mRNAの翻訳変化を網羅的に解析した。eIF4A1の欠損によりTOP mRNAの翻訳抑制は減弱した。
(C)TOP配列とLARP1の試験管内結合実験。eIF4A1とLARP1の精製タンパク質と放射線標識したTOP配列の合成RNAを混合し、紫外線により強固に結合させた後、電気泳動によりLARP1とeIF4A1を分離させた。タンパク質と結合したTOP配列の量は対応するバンド強度に反映される。eIF4A1を加えると添加量依存的にバンド強度が強くなり、eIF4A1がLARP1とTOP配列の結合を強化していることが分かった。
(D)mTOR阻害剤を加えた際の生存率。野生型とeIF4A1欠損細胞に対してmTOR阻害剤を添加して生存率を測定した。eIF4A1を欠損するとmTOR阻害剤に対して耐性を持つようになった。
(C)(D)にあるμMはμmol/L。
最後に、eIF4A1とよく似た配列を持つ因子であるeIF4A2の影響も調べました。eIF4A2はTOP mRNAとあまり強く結合せず、LARP1との結合もeIF4A1と比べて弱いことが分かりました。また、eIF4A2欠損細胞ではTOP mRNAの翻訳抑制が弱まる効果が見られず、eIF4A2はLARP1によるTOP mRNAの翻訳抑制機構には必要ないことが分かりました。
以上から、eIF4A1はmTOR阻害時においてLARP1の持つ翻訳抑制能力を高めている、つまり翻訳に対して抑制的に働くという、これまでの想定とは逆の機能を持つことが明らかになりました(図3)。
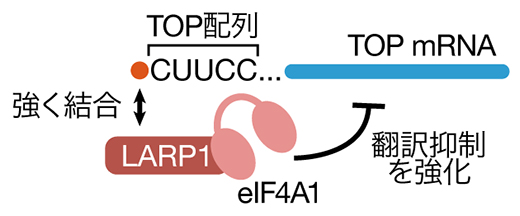
図3 eIF4A1による翻訳抑制の増強
アミノ酸飢餓によってmTORが阻害された際、LARP1が活性化されTOP mRNAの翻訳が抑制される。eIF4A1はLARP1と結合し、その後TOP mRNAとの結合および翻訳抑制能を強化する。
今後の期待
本研究は翻訳に対して促進的に働くと考えられてきたeIF4A1が、LARP1という結合因子を介して抑制的に働くという新しい機能を明らかにしました。岩崎主任研究員らの研究グループは、eIF4A1がロカグレートという薬剤と結合した際に翻訳を抑制することを発見していましたが注3)、今回の結果は薬剤処理のない生理的な条件でも翻訳阻害効果を示すという初めての発見となります。
mTORはがん細胞の増殖にも必要なためその阻害剤は抗がん剤として期待されていますが、一部のがん細胞は耐性を持つため治療における障害となっています。今回得られたeIF4A1を欠損するとmTOR阻害剤に対して耐性を示すという結果は、がん細胞のmTOR阻害剤耐性を予測するための指標となる可能性があり、mTOR阻害剤を用いたより良いがん治療へつながることが期待されます。
注3)2018年12月28日プレスリリース「植物由来抗がん剤の仕組み」
補足説明
1.翻訳、翻訳開始因子
翻訳とは、mRNAへコピーされた塩基配列をアミノ酸配列へ変換して、リボソームでアミノ酸を順番につなげてタンパク質を合成すること。翻訳開始因子とは、細胞内でタンパク質の合成(翻訳)を行うリボソームが、合成を開始する際に協調的に働くタンパク質群。真核生物の翻訳開始因子は真核生物型開始因子(eIF:eukaryotic Initiation Factor)と呼ばれる。
2.メッセンジャーRNA(mRNA)
タンパク質のアミノ酸の並び方の情報(コドン)を持つRNAのこと。リボソームによってそのコドンが読み取られ、タンパク質が合成される。
3.二次構造
1本鎖RNA内の塩基が結合することにより部分的に形成された2本鎖構造。翻訳開始においては障害となるため、塩基間の結合を解消する必要がある。RNAの塩基配列によって結合の強弱が異なるため、二次構造が翻訳開始を阻害する強さもRNAごとに異なる。
4.TOP mRNA、TOP配列
TOP配列とは、末端のキャップ構造の直後にシトシン(C:Cytosine)塩基とウラシル(U:Uracil)塩基が連続するTandem Oligo Pyrimidine(TOP)と呼ばれる配列のこと。TOP mRNAとは、TOP配列を持つmRNAのこと。
5.リボソーム
リボソームRNA(rRNA)とリボソームタンパク質から構成される超巨大複合体。リボソームはメッセンジャーRNA(mRNA)にコードされているコドンを読み取り、タンパク質を合成する。
6.RNA pulldown-Seq法
細胞の破砕液から目的タンパク質をプルダウンし、結合しているRNA配列を次世代シーケンサーにより同定する解析法。
7.次世代シーケンサー
DNAの塩基配列を決定するための装置。複数のDNA断片の塩基配列を同時並行で、高速・高精度に決定できる。
8.質量分析
物質を原子・分子レベルの微細なイオンにし、その質量数と数を測定することで物質の同定や定量を行う。
9.キャップ構造
真核生物のmRNAの5’末端に特徴的な構造。核酸の重合は通常5’→3’で結合が進むが、mRNAの5’末端は修飾反応によりグアノシンが5′-5’結合となっている。mRNAの5’末端の保護や、翻訳開始因子とmRNAの結合に関わっている。
10.mTOR
細胞外から入力されるさまざまなシグナルに応じて、細胞増殖やオートファジーなどを制御しているセリン/スレオニンキナーゼ。mTORはmammalian/mechanistic Target Of Rapamycinの略。
11.リボソームプロファイリング法
組織や細胞から翻訳装置であるリボソームを抽出し、リボソームと結合しているRNA配列を同定することで、どの遺伝子がどの程度の効率で翻訳されているかを知る解析法。リボソームは大きな複合体であるため、一定のmRNA領域を覆うように結合する。これらのリボソームとmRNAの複合体をRNA分解酵素で処理すると、リボソームが保護するmRNA断片だけが分解されずに回収される。
国際共同研究グループ
理化学研究所
開拓研究本部 岩崎RNAシステム生化学研究室
主任研究員 岩崎 信太郎(イワサキ・シンタロウ)
研究員 七野 悠一(シチノ・ユウイチ)
テクニカルスタッフⅠ 水戸 麻理(ミト・マリ)
生命機能科学研究センター 翻訳構造解析研究チーム
チームリーダー 伊藤 拓宏(イトウ・タクヒロ)
研究員 柏木 一宏(カシワギ・カズヒロ)
テクニカルスタッフⅠ 髙橋 真梨(タカハシ・マリ)
九州大学大学院 医学研究院 基礎医学部門 生体情報科学講座
教授 久場 敬司(クバ・ケイジ)
助教 山口 智和(ヤマグチ・トモカズ)
カリフォルニア大学バークレー校(米国)Department of Molecular and Cell Biology
助教授 ニコラス・インゴリア(Nicholas T. Ingolia)
研究支援
本研究は、理化学研究所運営費交付金組織横断連携プロジェクト「ライフサイエンスの横断的取組による超高齢社会課題解決への貢献(研究分担者:岩崎信太郎、伊藤拓宏)」、RIKEN Pioneering Projects「Biology of Intracellular Environments(研究分担者:岩崎信太郎、伊藤拓宏)」、Dynamic Structural Biology Project(伊藤拓宏)、奨励課題(七野悠一)、基礎科学特別研究員制度(七野悠一)で実施し、日本学術振興会(JSPS)科学研究費助成事業学術変革領域研究(A)「APEX-Ribo-Seq:近傍標識による非典型局所翻訳の網羅解析(研究代表者:七野悠一)」、同学術変革領域研究(B)「新規Disome-Seq法:パラメトリックなリボソーム渋滞の網羅的探索(研究代表者:岩崎信太郎)」、同若手研究(A)「翻訳開始因子パラログによる選択的翻訳の網羅的解析(研究代表者:岩崎信太郎)」、同基盤研究(B)「複合体間の動的な相互作用による翻訳制御の構造基盤(研究代表者:伊藤拓宏)」「循環代謝病におけるRNA時空間制御機構の解明(研究代表者:久場敬司)」、同挑戦的研究(萌芽)「癌の転移・再発における細胞外Galectin-Xを介した免疫抑制機構の解明(研究代表者:久場敬司)」、同若手研究「翻訳開始因子eIF4A1とグルタミン代謝による協調的な発現制御機構(研究代表者:七野悠一)」「ストレス環境における翻訳開始因子eIF2Bの活性阻害機構の解明(研究代表者:柏木一宏)」による助成、および理化学研究所脳神経科学研究センター生体物質分析支援ユニットの支援を受けて行われました。
原論文情報
Yuichi Shichino, Tomokazu Yamaguchi, Kazuhiro Kashiwagi, Mari Mito, Mari Takahashi, Takuhiro Ito, Nicholas T. Ingolia, Keiji Kuba, and Shintaro Iwasaki, “eIF4A1 enhances LARP1-mediated translational repression during mTORC1 inhibition”, Nature Structural & Molecular Biology, 10.1038/s41594-024-01321-7
発表者
理化学研究所
開拓研究本部 岩崎RNAシステム生化学研究室
主任研究員 岩崎 信太郎(イワサキ・シンタロウ)
研究員 七野 悠一(シチノ・ユウイチ)
生命機能科学研究センター 翻訳構造解析研究チーム
チームリーダー 伊藤 拓宏(イトウ・タクヒロ)
研究員 柏木 一宏(カシワギ・カズヒロ)
九州大学 大学院医学研究院 薬理学分野
教授 久場 敬司(クバ・ケイジ)
助教 山口 智和(ヤマグチ・トモカズ)
報道担当
理化学研究所 広報室 報道担当
九州大学 広報課