2018/11/09 ライフサイエンス統合データベースセンター(DBCLS)
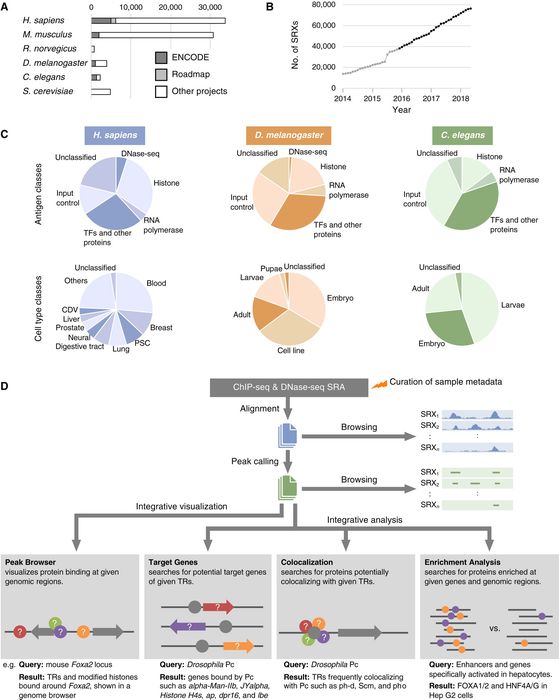
ChIP-Atlas https://chip-atlas.org/ は、大規模シーケンスデータのための公共データベースである Sequence Read Archive から ChIP-Seq, DNase-Seq のデータを取得し、共通の解析パイプラインで再解析したデータを集積したデータベースです。集積した再解析データを用いて、利用者はゲノムブラウザでのデータ可視化、転写因子が制御する遺伝子、共局在する転写因子の解析や、ユーザーのデータを利用したエンリッチメント解析を行うことができます。今回、ChIP-Atlas を用いた解析によって、組織特異的に発現する遺伝子を制御する転写因子の発見に繋がることを論文で示しました。ChIP-Atlas のデータは継続的に更新されており、今後さらに様々な研究で有用なデータリソース・解析アプリケーションとして活用されることが期待されます。
Shinya Oki, Tazro Ohta, Go Shioi, Hideki Hatanaka, Osamu Ogasawara, Yoshihiro Okuda, Hideya Kawaji, Ryo Nakaki, Jun Sese, and Chikara Meno. ChIP-Atlas: a data-mining suite powered by full integration of public ChIP-seq data. EMBO Reports https://doi.org/10.15252/embr.201846255
Abstract
We have fully integrated public chromatin chromatin immunoprecipitation sequencing (ChIP-seq) and DNase-seq data (n > 70,000) derived from six representative model organisms (human, mouse, rat, fruit fly, nematode, and budding yeast), and have devised a data-mining platform—designated ChIP-Atlas (http://chip-atlas.org). ChIP-Atlas is able to show alignment and peak-call results for all public ChIP-seq and DNase-seq data archived in the NCBI Sequence Read Archive (SRA), which encompasses data derived from GEO, ArrayExpress, DDBJ, ENCODE, Roadmap Epigenomics, and the scientific literature. All peak-call data are integrated to visualize multiple histone modifications and binding sites of transcriptional regulators (TRs) at given genomic loci. The integrated data can be further analyzed to show TR–gene and TR–TR interactions, as well as to examine enrichment of protein binding for given multiple genomic coordinates or gene names. ChIP-Atlas is superior to other platforms in terms of data number and functionality for data mining across thousands of ChIP-seq experiments, and it provides insight into gene regulatory networks and epigenetic mechanisms.