新しいバイオ素材合成などの応用研究の加速に向けて
2018-01-22 理化学研究所
要旨
理化学研究所(理研)環境資源科学研究センター合成ゲノミクス研究グループの蒔田由布子研究員、松井南グループディレクターらの国際共同研究チーム※は、パラゴムノキ[1]の研究基盤となる遺伝子・転写関連データベースを構築・公開しました。
天然ゴムは、車や航空機のタイヤ、医療用装置の部品などに使われ、私たちの日常生活においても重要な天然資源です。パラゴムノキはプランテーションで大規模に栽培されていますが、食料やオイルパームとの耕地の競合や、森林系生態保護の観点から、耕地を増やさず、天然ゴムの原料であるラテックスを増産することが望まれています。そのため、増産に向けたバイオテクノロジーによる研究が進められていますが、研究はまだ始まったばかりで、基礎情報が不足しているのが現状です。
国際共同研究チームは、研究推進に向けた研究基盤データベースを構築するため、2016年にパラゴムノキのドラフトゲノム配列を決定し注1)、その後、天然ゴム高生産株・病害抵抗株の遺伝子発現解析を行いました注2)。今回、これらのゲノムや遺伝子発現情報、世界の関連研究成果をまとめ、パラゴムノキの遺伝子・転写関連データベースを構築しました。これにより、研究者がこれまで膨大な時間と労力をかけて集めていた情報に簡単にアクセスできます。また、遺伝子情報を活用することで、ラテックス合成メカニズムの解明や、遺伝子改変による新しいバイオ素材合成などの応用研究が加速されると考えられます。
パラゴムノキの研究は中国をはじめ、世界的な競争が激化しつつある分野です。研究基盤データベースを公開することで、応用研究への貢献につながると期待できます。
本研究成果は、米国の科学雑誌『BMC Genomics』に掲載されるのに先立ち、オンライン版(1月19日付け)に掲載されました。
注1)2016年6月24日プレスリリース「天然ゴムのドラフトゲノムを解読」
注2)2017年2月2日プレスリリース「天然ゴム高生産株・病害抵抗株の遺伝子発現解析」
※国際共同研究チーム
理化学研究所 環境資源科学研究センター 合成ゲノミクス研究グループ
グループディレクター 松井 南 (まつい みなみ)
研究員 蒔田 由布子 (まきた ゆうこ)
テクニカルスタッフI 川島 美香 (かわしま みか)
マレーシア科学大学 生物学部
教授 アフメド・ソフィマン・オスマン(Ahmad Sofiman Othman)
博士 ニョクシン・ラウ(Nyok Sean Lau)
背景
天然ゴムは、車や航空機のタイヤ、医療用装置の部品など、私たちの日常で広く使われています。合成ゴムに比べて、耐久性が高く、発熱性が低いなどの利点があり、耐久性が求められるゴム製品において欠かせない材料です。また新興国の産業発展に伴い、その需要も年々高まっています。一方、パラゴムノキは、食料やオイルパームとの競合や、森林生態系の保護のため、耕地面積の拡大による増産は望めないという問題があります。そのため単位面積あたりのラテックス(天然ゴムの原料、樹皮を傷つけると分泌する乳白色の液体)の生産性を高めたパラゴムノキ優良株の作出が求められています。
ラテックスの合成経路は、中間産物であるイソプレン[2]の合成過程までは明らかになっていますが、イソプレンが高分子化され、ラテックス(主成分はポリイソプレン[2])が合成されるパラゴムノキ特有の経路においては、その全容は明らかにされていません。いくつかの重要な遺伝子(SRPP、REF、CPT、CPTLなど)は知られていますが、これらに加え、他にも必要な因子があると考えられています。機能が未知な遺伝子を探索するため、既存の情報を整理したうえでデータベース化し、活用していく必要があります。
そのようなデータベースはこれまで、パラゴムノキには存在していませんでした。そこで国際共同研究チームは、パラゴムノキの研究基盤データベースの構築・公開を試みました。
研究手法と成果
国際共同研究チームが構築したパラゴムノキのデータベースは、遺伝子やその機能予想といったゲノム配列に基づく情報と、それらの遺伝子が、いつ、どこで、どのように働くかを調べた転写関連情報から構成されています。
全ての配列データはゲノムブラウザ上から閲覧できます。図1に示すように、ゲノム配列、遺伝子予測、完全長cDNA[3]をはじめ、天然ゴム高生産株・病害抵抗株の遺伝子発現解析の結果や、CAGE法[4]による転写開始点などの転写関連データもサンプルごとに閲覧できるようになっています。
また、パラゴムノキはモデル生物ではなく、機能アノテーション[5]も少ないことから、検索機能を充実させています。通常のキーワード検索に加え、例えば、葉に比べラテックスで10倍以上の発現量を示す遺伝子を検索するなど、ユーザーは自分で発現量を計算することなく検索結果の遺伝子にアクセスできます。また、パラゴムノキにおいて重要なラテックス合成経路の図を用意することで、既にゲノム上で見つかった各遺伝子へ進むこともできます(図2)。
今後の期待
近年、中国を含む東南アジア諸国ではパラゴムノキの研究が活発に取り組まれています。パラゴムノキの遺伝子やその機能、発現の様子などを確認することができるデータベースは、生産性向上や、耐病性獲得などのため、応用研究に向けた活用が期待できます。また、ラテックスの生合成経路が明らかになれば、新しい素材合成のための遺伝子改変を考慮する際に活用されると期待できます。
原論文情報
- Yuko Makita, Mika Kawashima, Nyok Sean Lau, Ahmad Sofiman Othman, and Minami Matsui, “Construction of Pará rubber tree genome and multi-transcriptome database accelerates rubber researches”, BMC Genomicss, doi: 10.1186/s12864-017-4333-y
発表者
理化学研究所
環境資源科学研究センター バイオマス工学研究部門 合成ゲノミクス研究グループ
研究員 蒔田 由布子 (まきた ゆうこ)
グループディレクター 松井 南 (まつい みなみ)
報道担当
理化学研究所 広報室 報道担当
産業利用に関するお問い合わせ
理化学研究所 産業連携本部 連携推進部
補足説明
-
- パラゴムノキ
- トウダイグサ科パラゴムノキ属の常緑高木。幹を傷つけて得られる乳液(ラテックス)は天然ゴムの原料となる。「パラ」は原産地であるブラジルの北部にあるパラ州に由来する。学名はHevea brasiliensis。
-
- イソプレン、ポリイソプレン
- イソプレンは、構造式がCH2=C(CH3)CH=CH2の炭化水素。イソプレンが重合してできた高分子化合物をポリイソプレンと呼ぶ。さまざまな植物が生合成するポリイソプレンは重合の度合いが異なり、いろいろな長さのものがある。特にゴムノキが生合成するイソプレンは重合度が高く高分子化している。
-
- 完全長cDNA
- cDNAは、mRNAを鋳型にして作られたDNAのこと。mRNAはゲノムDNAの中から不要な配列を除き、タンパク質をコードする配列のみに整理された遺伝情報物質である。完全長cDNAは、断片cDNAと異なり、タンパク質を合成するための設計情報を全て持っているため、タンパク質を合成することができる。cDNAはcomplementary DNAの略。
-
- CAGE法
- すべての遺伝子の発現量と転写開始部位を正確に調べる技術。理研が開発した。mRNAの最初のCAPと呼ばれる構造を利用する。組織や生育時期などで転写の開始部位が変化するスイッチングなどの貴重な情報が得られる。CAGEは、Cap Analysis of Gene Expressionの略。
-
- アノテーション
- 見いだされた遺伝子配列やアミノ酸配列について、既知の遺伝子、タンパク質などとの比較から機能を予測し、注釈として記述すること。
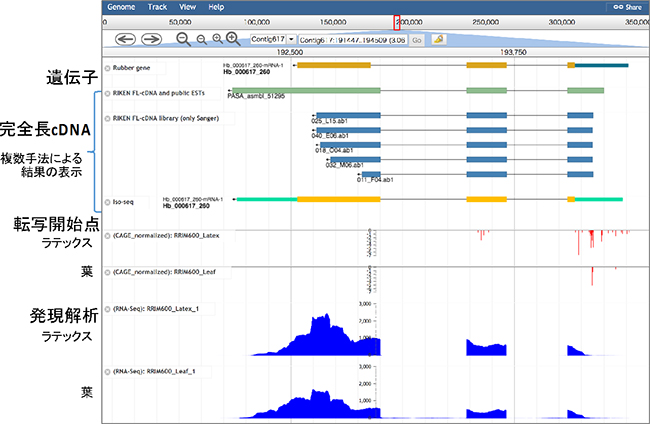
図1 本データベースで公開するデータの一例
ゲノムブラウザ上で、ゲノム配列、遺伝子予測、完全長cDNA、天然ゴム高生産株・病害抵抗株の遺伝子発現解析の結果、転写開始点などデータがサンプルごとに閲覧できる。横軸は遺伝子のゲノム中での位置を示し、縦軸方向に多様なトランスクリプトームデータを選択して表示する。
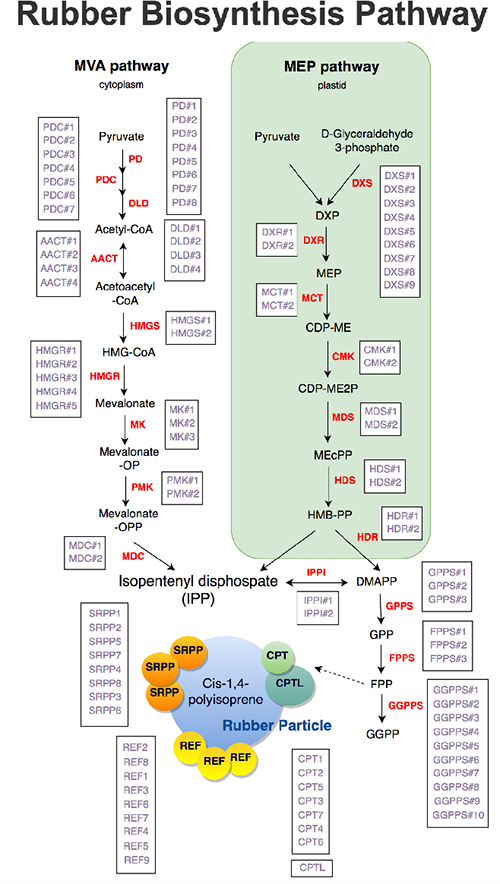
図2 ラテックス合成経路と遺伝子リスト
ラテックスの主成分であるポリイソプレンの合成経路に、ゲノム解読で見つかった遺伝子リストを配置することで、パスウェイ中の酵素を合成すると考えられる遺伝子群の詳細情報へアクセスできる。