2019-11-08 大阪大学,日本医療研究開発機構
研究成果のポイント
- 微生物叢の全ゲノム情報(メタゲノム※1)に対するショットガンシークエンス※2により、網羅的な解析を行うメタゲノムワイド関連解析を実施
- 日本人集団の関節リウマチ※3患者の腸内微生物叢※4に特異的な菌種や遺伝子量の変動を明らかに
- 関節リウマチの大規模疾患ゲノム解析の結果とメタゲノム解析の結果を比較し、メタゲノムと宿主のヒトゲノムの間にアジア人集団特有の関連があることを同定
概要
大阪大学 大学院医学系研究科 大学院生の岸川敏博さん(博士課程)、岡田随象 教授(遺伝統計学)らの研究グループは、日本人集団の関節リウマチ患者と健常者の腸内微生物叢が持つゲノム情報(メタゲノム)から、菌種や遺伝子、パスウェイ※5などの情報を網羅的に解析するメタゲノムワイド関連解析を実施しました(図)。その結果、関節リウマチ患者由来のメタゲノムにはPrevotella(プレボテラ)属に属する複数の種の増加や酸化還元反応に関連する遺伝子の減少など、特異的な変動が生じていることを明らかにしました。また、関節リウマチの大規模疾患ゲノム解析(ゲノムワイド関連解析※6)より得たパスウェイ解析結果をメタゲノムによるパスウェイ解析結果と比較したところ、メタゲノムとヒトゲノムで関節リウマチに関与する多くのパスウェイがアジア人特異的に共有されていることを同定しました。
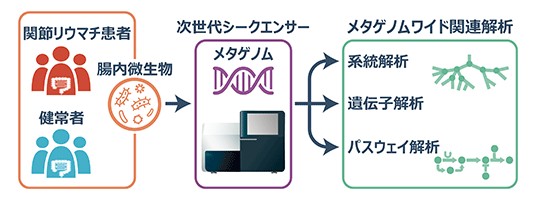
腸内微生物叢の全ゲノム情報を用いた網羅的な解析
これまで、関節リウマチを含む多くの疾患で微生物叢と病原性との関連が指摘されてきましたが、微生物叢のゲノム全体(メタゲノム)の解析は、膨大なゲノムデータ量と複雑な情報解析行程のために、普及は進んでいません。
今回、岡田教授らの研究グループは、菌種組成を算出する系統解析、遺伝子解析、パスウェイ解析の3つを軸に網羅的な解析を行うメタゲノムワイド関連解析のパイプラインを構築しました。この手法を日本人の関節リウマチ患者と健常者の腸内微生物叢に対して施行し、疾患の発症に関与している可能性のある菌種や遺伝子量の変動を明らかにしました。本研究成果により、腸内微生物叢が関節リウマチの発症に関与する機序の解明に貢献することが期待されます。
本研究成果は、英国科学誌「Annals of the Rheumatic Diseases」に11月8日(金)午前1時に公開されます。
研究の背景
腸内や皮膚など、私たちの体に生息する微生物は、免疫反応や代謝応答に大きな影響を及ぼしており、様々な微生物叢の個人差が、2型糖尿病や心血管障害、炎症性腸疾患、がんなど多くの疾患の病因に関与しています。免疫異常により全身の関節が破壊される関節リウマチにおいても、Prevotella copri(プレボテラ・コプリ)によるTh17細胞を介した免疫応答の活性化など、免疫異常につながる微生物叢の働きが報告されています(Maeda Y et al. Arthritis Rheumatol 2016)。また、関節リウマチは、ヒトゲノムの個人差を対象としたゲノムワイド関連解析により多くの感受性ゲノム領域が発見された代表的な疾患の1つですが(Okada Y et al.Nature 2014)、宿主のヒトゲノムと微生物叢のメタゲノムの間の相互作用など、微生物叢がもたらす関節リウマチの病因の全容は、未だ解明されていません。
これまで、微生物叢の研究は細菌がもつ一部の遺伝子(16SリボソームRNA)のみに着目した解析手法が中心でしたが、今日では、微生物叢のゲノム全体(メタゲノム)を対象に次世代シークエンサーを用いるショットガンシークエンスという手法が推奨され、微生物叢の病原性の機序の一端が解明されつつあります。メタゲノム解析では、菌種の組成だけでなく、メタゲノムが含む遺伝子やパスウェイなど、生物学的機能特性の詳細な分析を行うことができ、薬物療法や糞便移植療法、プロバイオティクスなどの新しい指標を提示できる可能性を持っています。一方で、メタゲノム解析は、膨大なゲノムデータ量と複雑な情報解析行程のために、普及は進んでいません。さらに、人種集団性および食習慣は微生物叢の組成に影響を与えることが知られていますが、非欧米人集団の微生物叢研究は数少ないのが現状です。
本研究の成果
岡田教授らは、日本人集団(関節リウマチ患者82名、健常者42名)の腸内微生物叢に対して、ショットガンシークエンスによる包括的なメタゲノムワイド関連解析を、独自に構築した情報解析パイプラインを用いて実施しました。患者と健常者間の菌種組成の比較において、関節リウマチ由来のメタゲノムにはPrevotella(プレボテラ)属に属する複数の種(P. denticola(デンティコーラ), P. marshii(マルシイ), P. disiens(ディシエンス), P. corporis(コーポリス), P. amnii(アムニー))が健常群より有意に増加していることを発見しました。これらの菌種は関節リウマチの病原性に重要な役割を担う可能性が考えられます。また、非線形モデルによる機械学習手法を用いて、患者と健常者間で相違のあったこれらの菌種を効率的に判別できることを明らかにしました。
患者と健常者間のメタゲノムに含まれる遺伝子量の比較においては、関節リウマチ由来のメタゲノムは、酸化還元反応に関連する遺伝子の一つ(R6FCZ7)が健常群と比較して有意に減少していることを同定しました。腸内微生物叢の酸化ストレスに対する脆弱性が関節リウマチの病因に関与している可能性が考えられます。
パスウェイ解析においては、脂肪酸生合成やグリコサミノグリカン分解など代謝応答領域のものを中心に様々なパスウェイが、患者と健常者間の比較で有意差を示しました。多くのパスウェイは関節リウマチの病原性との関連が示唆されているものであり、これらの関連に腸内微生物叢が影響している可能性が考えられます。また、この関節リウマチのメタゲノムによるパスウェイ結果に関して、関節リウマチのゲノムワイド関連解析より得たパスウェイ解析結果と比較したところ、メタゲノムとヒトゲノムの間で、アジア人特異的に疾患に関与するパスウェイが共有されていることを明らかにしました。一方で、これまで多くの疾患で報告されてきた微生物叢における多様性の低下についても詳細な検討を行った結果、本研究では関節リウマチと健常群の間に多様性の有意な相違は認められませんでした。
本研究成果が社会に与える影響(本研究成果の意義)
今回、ショットガンシークエンスを用いたメタゲノムワイド関連解析によって、腸内微生物叢とホストゲノム、関節リウマチの3者間における関連を見いだしました。これらの知見は、関節リウマチの病因における微生物叢の役割に関して、より深い理解をもたらすことが期待され、更なる疾患病態解明につながることが期待されます。
また、関節リウマチ患者に特徴的な腸内微生物叢由来のメタゲノム情報をバイオマーカーとして用いることで、関節リウマチの診断に貢献することが期待されます。
用語説明
- ※1 メタゲノム
- 環境中に含まれる全微生物のゲノム情報の集合。各微生物の単離・培養を行わず、集団から直接収集・抽出される。
- ※2 ショットガンシークエンス
- 微生物の全ゲノムDNA を物理的に切断することで作成した短い DNA 鎖に対して、次世代シーケンサーによって配列を決定し、コンピュータの解析プログラムを用いて元の遺伝子配列を再構築する手法。
- ※3 関節リウマチ
- 自己免疫疾患(病原体を体内から排除するために機能している免疫システムが、自分の体を攻撃することで発症する病気)の一つで、関節の炎症と破壊を生じる疾患。国内に約70~80万人の患者がいると推定されている。
- ※4 腸内微生物叢そう
- 宿主であるヒトや動物と共生関係にある多種多様な腸内微生物の集まり。代謝物の産生や病原体の排除、免疫の賦活化など、様々な宿主との相互作用を持ち、その乱れ(ディスバイオーシス)は代謝や免疫、心血管疾患の原因とされる。
- ※5 パスウェイ
- 遺伝子やタンパク質の相互作用、細胞内の連鎖的な化学反応など、生体内における様々なネットワークを示す経路。
- ※6 ゲノムワイド関連解析
- ヒトゲノム配列上に存在する数百万カ所の遺伝子変異とヒト疾患との発症の関係を網羅的に検討する、遺伝統計解析手法。数千人~百万人を対象に大規模に実施されることで、これまで1000を超えるヒト疾患に対する遺伝子変異が数多く同定されている。
特記事項
本研究成果は、英国科学誌「Annals of the Rheumatic Diseases」(オンライン)に11月8日(金)午前1時に掲載されます。
- 【タイトル】
- “ A metagenome-wide association study of gut microbiome revealed novel etiology of rheumatoid arthritis in the Japanese population.”
- 【著者名】
- Toshihiro Kishikawa1,2, Yuichi Maeda3,4, Takuro Nii3,4, Daisuke Motooka5, Yuki Matsumoto5, Masato Matsushita6,7, Hidetoshi Matsuoka7, Maiko Yoshimura7, Shoji Kawada8, Satoru Teshigawara7, Eri Oguro3,7, Yasutaka Okita7, Keisuke Kawamoto8, Shinji Higa8, Toru Hirano3, Masashi Narazaki3, Atsushi Ogata8, Yukihiko Saeki7,9, Shota Nakamura5, Hidenori Inohara2, Atsushi Kumanogoh3,10, Kiyoshi Takeda4,11, and Yukinori Okada*1,12,13(* 責任著者)
- 【所属】
-
- 大阪大学大学院 医学系研究科 遺伝統計学
- 大阪大学大学院 医学系研究科 耳鼻咽喉科・頭頸部外科学
- 大阪大学大学院 医学系研究科 呼吸器・免疫内科学
- 大阪大学大学院 医学系研究科 免疫制御学
- 大阪大学 微生物病研究所 感染症メタゲノム研究分野
- 大阪府済生会千里病院 リウマチ・アレルギー科
- 大阪南医療センター リウマチ・膠原病・ アレルギー科
- 第二大阪警察病院 膠原病・リウマチ科
- 大阪南医療センター 臨床研究部
- 大阪大学 免疫学フロンティア研究センター 感染病態分野
- 大阪大学 免疫学フロンティア研究センター 粘膜免疫学
- 大阪大学 免疫学フロンティア研究センター 免疫統計学
- 大阪大学 先導的学際研究機構 生命医科学融合フロンティア研究部門
本研究は、日本医療研究開発機構(AMED)革新的先端研究開発支援事業PRIME「微生物叢と宿主の相互作用・共生の理解と、それに基づく疾患発症のメカニズム解明」(研究開発総括:笹川千尋)の採択課題「遺伝統計学が紐解く微生物叢・宿主・疾患・創薬のクロストーク」(研究開発代表者:岡田随象)の一環として行われ、大阪大学大学院医学系研究科 熊ノ郷淳教授・前田悠一助教・新居卓朗大学院生(呼吸器・免疫内科学)、大阪大学大学院医学系研究科 竹田潔教授(免疫制御学)、大阪大学免疫学フロンティア研究センター 次世代主任研究者支援プログラム大阪大学先導的学際研究機構、大阪大学大学院医学系研究科 バイオインフォマティクスイニシアティブ、の協力を得て行われました。
本件に関するお問い合わせ先
研究に関すること
岡田 随象(おかだ ゆきのり)
大阪大学 大学院医学系研究科 遺伝統計学 教授
報道に関すること
大阪大学大学院医学系研究科
広報室
AMED事業に関すること
国立研究開発法人日本医療研究開発機構
基盤研究事業部 研究企画課