植物ウイルスの安定なキャプシド構造が支える遺伝子放出機構
2020-09-04 理化学研究所,岩手大学
理化学研究所(理研)放射光科学研究センター利用技術開拓研究部門生体機構研究グループの内藤久志先任研究員、浜口祐研究員、米倉功治グループディレクター、XFEL研究開発部門ビームライン研究開発グループイメージング開発チームの眞木さおり研究員、岩手大学農学部植物生命科学科植物病理学分野植物病理学研究室の吉川信幸教授、磯貝雅道准教授の共同研究グループは、クライオ電子顕微鏡[1]を用いて、リンゴに潜在感染[2]するリンゴ小型球形潜在ウイルス(ALSV)[3]の構造と、宿主の植物細胞内でALSVからウイルス遺伝子[4]が放出される様子を捉えることに成功しました。
本研究により、ALSVを含む属[5]のウイルスとして初めてその原子構造が明らかになったことで、植物ウイルスが宿主細胞に感染する際に起こるウイルス遺伝子の放出機構のより詳しい理解や、植物ウイルスの抗ウイルス薬開発、植物細胞内に外来遺伝子を送り込む方法への応用につながると期待できます。
植物ウイルスは多くの場合、宿主となる植物細胞の細胞壁表面の傷などを通り宿主細胞内に侵入し、自身のタンパク質合成のために速やかにウイルス遺伝子を放出します。しかし、ウイルスが宿主細胞内でどのようにウイルス遺伝子を放出するのか、詳しい仕組みは明らかではありませんでした。
今回、共同研究グループは、冷陰極電界放出型の電子銃を備えた新型の国産クライオ電子顕微鏡を用いて、ALSVの原子構造を2.87オングストローム(Å、1Åは100億分の1メートル)分解能で決定し、同時に自身の遺伝子を放出途中のウイルスのクライオ電子顕微鏡像の取得に成功しました。
本研究は、オンライン科学雑誌『Communications Biology』(9月4日付:日本時間9月4日)に掲載されます。
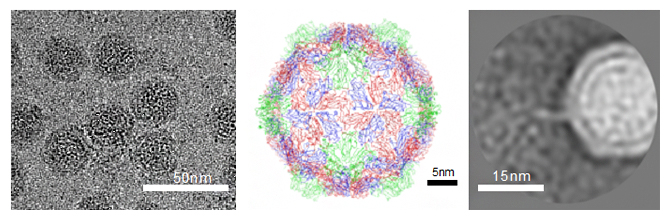
左からALSVの氷包埋像、ウイルスの全体構造、遺伝子放出途中のウイルスの二次元平均像
背景
植物ウイルスは多くの場合、宿主となる植物細胞の細胞壁表面の傷などから宿主細胞内に侵入すると、自身のタンパク質合成のために速やかにウイルス遺伝子を放出します。そのため、植物ウイルスは、ウイルス遺伝子をキャプシド[6]と呼ばれる構造の内部に保持し、宿主細胞内に侵入してからウイルス遺伝子を放出した後、その遺伝子を元に新たに合成されたキャプシドタンパク質[7]で複製されたウイルス遺伝子をカプセル化すると考えられてきました。
リンゴに潜在感染するリンゴ小型球形潜在ウイルス(ALSV)が所属する植物ウイルスグループでは、これまでキャプシドに関する広範な研究が行われてきましたが、その構造は不明でした。また、植物ウイルス遺伝子放出機構についてはほとんど分かっていませんでした。
研究手法と成果
理研が所有するクライオ電子顕微鏡装置(日本電子社CRYO ARM 300)は、干渉性の高い電子ビームを発生する冷陰極電界放出型の電子銃を備え、従来型の装置に比較して、少ない数の像から精度良く高い分解能の構造を取得できます注1)。
共同研究グループは、この新型クライオ電子顕微鏡を用いた単粒子解析法[8]により、ALSVの原子構造を2.87オングストローム(Å、1Åは100億分の1メートル)分解能で決定しました(図1)。この原子構造はCheravirus属に所属するウイルスとして初めての構造決定であり、植物ピコルナウイルスの空白域を埋める成果となります。その結果、ALSVの3種類のキャプシドタンパク質(Vp20、Vp24、Vp25)が独特の構造を形成することで、キャプシド構造が安定化されていることが明らかになりました。このユニークな構造は、Vp25の長いN末端[9]延長部分が、キャプシド内表面を経て隣接するVp20や別のVp25、ウイルス遺伝子と相互作用することで、一つの骨格構造を形作っていることが特徴です(図2)。このような構造の類似の報告は、これまでにありませんでした。

図1 リンゴ小型球形潜在ウイルス(ALSV)の氷包埋像(左)と原子構造(右)
左:ALSVの氷包埋像。スケールバーのnmは10億分の1メートル。
右:単粒子解析法により得られた三次元構造に基づき、ウイルスキャプシド(直径約30nm)の主鎖構造だけを描いた。キャプシドを構成する3種類のタンパク質分子はそれぞれ色分けされている(Vp25: 赤、Vp20: 青、Vp24: 緑)。
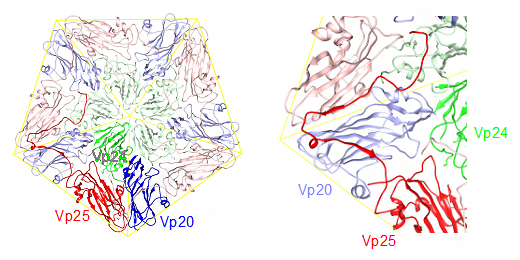
図2 ALSVのリボン図(左)とその一部の拡大図(右)
ウイルス内側から見た図。Vp20は青、Vp24は緑、Vp25は赤で表示しているが、1組のプロトマー(ウイルスのキャプシドを構成する最小ユニット)は濃色表示、残りの4組のプロトマーは淡色表示としている。左は、5回転軸周りの五つのプロトマーだけを表示している。右の拡大図では、濃い赤で表示されたVp25のN末端延長部分が、隣接するVp20(淡青)およびその先にある別のVp25(淡赤)と、濃赤の矢印で示すような水素結合を伴うβシートを作る一方、Vp24(淡緑)とはβシートを作らない。
さらに、この構造フレーム内部にあるウイルス遺伝子が、Vp24の5量体の蓋の中心の穴から漏れ出る様子をクライオ電子顕微鏡で捉えることに成功しました(図3)。
ウイルスのキャプシドは、相反する機能を持つことが必要です。一つは遺伝子を安定して保持することで、もう一つは宿主の細胞内で速やかに自己の遺伝子を放出することです。ALSVのキャプシドを構成するVp20、Vp24、Vp25のうち、Vp20とVp25が密に結合することで、安定なキャプシドのカプセル構造に作り上げます。そして、このカプセル構造の開口部をVp24の5量体が蓋をするように配置しており、この蓋がALSVの突起部分となります。一部のクライオ電子顕微鏡像の画像処理の結果、この突起部分からウイルス遺伝子が流出しているのが分かりました。この部分は揺らぎが大きいことが構造解析の結果から明らかになり、ある一定の割合で常にウイルス遺伝子の放出が起こっていると考えられます。

図3 ALSVの静電ポテンシャルマップ断面図(左)と再構成像(右)
左は、ウイルスキャプシド内部にある遺伝子部分を黄色で示す。右は、一部の像を使って得られた二次元平均像で、ウイルス表面から紐状の遺伝子が放出されているのが分かる。
注1)2019年5月21日プレスリリース「タンパク質やその複合体の高分解能・高精度解析に成功」
今後の期待
今回、ALSVが含まれるCheravirus属のウイルスとして、初めてその原子構造が明らかになりました。本研究結果は、他の植物ウイルスにおいてウイルス遺伝子を放出させない薬剤(抗ウイルス薬)や、逆にALSVが潜在感染する特徴を生かして植物細胞内へ効率良く外来遺伝子を送り込むことで、形質転換や有用な物質の合成を促すウイルスベクター[10]等への応用開発が期待できます。
補足説明
1.クライオ電子顕微鏡
タンパク質などの生体分子を、水溶液中の生理的な環境に近い状態で、電子顕微鏡で観察するために開発された手法。まず、試料を含む溶液を液体エタン(約-170℃)中に落下させ急速凍結し、アモルファス(非晶質、ガラス状)な薄い氷に包埋する。これを液体窒素(-196℃)冷却下で、電子顕微鏡観察する。電子顕微鏡内の真空中で試料は氷中に保持でき、また、冷却することで電子線の照射による損傷を減らせる。Jacques Dubochetは氷包埋の手順を作り、2017年のノーベル化学賞の受賞者の一人に選ばれた。
2.潜在感染
宿主にウイルス感染が成立しても、目立った病徴や症状を示さない感染状態。ALSVの場合、潜在感染の状態で新しいウイルスが作られる。
3.リンゴ小型球形潜在ウイルス(ALSV)
ピコルナウイルス目セコウイルス科Cheravirus属に所属する直径約300オングストローム(Å、1Åは100億分の1メートル)の1本鎖RNAウイルス。宿主範囲が広く、リンゴ(自然感染宿主)、モモ、ナシ、ウリ科、マメ科、ナス科植物に潜在感染する。
4.ウイルス遺伝子
ウイルスが宿主細胞内で自己複製するのに必要なタンパク質分子の遺伝情報がコードされたもの。
5.属
生物分類学上のリンネ式階級分類における基本的階級の一つ。属は科の下、種の上に位置する。ウイルス分類の場合、ウイルスの特性を決定する重要な性質(宿主範囲、毒性の違いなど)に一部違いはあっても、共通する性質を持つウイルスグループを表す。
6.キャプシド
ウイルス遺伝子を包み込むウイルスのタンパク質構造。最小構成ユニットであるプロトマーが多数集合して構築される。ALSVの場合は60個のプロトマーが集合して一つのALSVが構築される。
7.キャプシドタンパク質
キャプシドを構成するタンパク質のこと。キャプシドを構成するタンパク質の分子数はウイルスによって異なる。
8.単粒子解析法
電子顕微鏡で撮影した多数の生体分子の像から、その立体構造を決定する構造解析手法。結晶を作製しなくても分子の構造が得られる。技術革新により、理想的な試料ではX線結晶構造解析に勝る空間分解能で構造が決定できるようになった。2017年のノーベル化学賞受賞者の一人、Joachim Frankらにより単粒子解析法の基礎が作られた。
9.N末端
タンパク質はアミノ酸がつながったポリマーであり、隣接するアミノ酸は、それぞれのアミノ基とカルボキシル基がペプチド結合をしている。このポリマーの末端のフリーのアミノ基側をN末端、カルボキシル基側をC末端と呼ぶ。
10.ウイルスベクター
ウイルスの感染、増殖能力を利用して外来遺伝子を生体内で簡便に発現できるツール。遺伝子の機能解析等の基礎研究から医薬品生産等の応用分野まで活用範囲が広い。
共同研究グループ
理化学研究所 放射光科学研究センター
利用技術開拓研究部門 生体機構研究グループ
グループディレクター 米倉 功治(よねくら こうじ)
(科技ハブ産連本部 バトンゾーン研究推進プログラム 理研-JEOL連携センター 次世代電子顕微鏡開発連携ユニット ユニットリーダー)
先任研究員 内藤 久志(ないとう ひさし)
研究員 浜口 祐(はまぐち たすく)
XFEL研究開発部門 ビームライン研究開発グループ イメージング開発チーム
研究員 眞木 さおり(まき さおり)
岩手大学 農学部 植物生命科学科 植物病理学分野 植物病理学研究室
教授 吉川 信幸(よしかわ のぶゆき)
准教授 磯貝 雅道(いそがい まさみち)
研究支援
本研究は、日本学術振興会(JSPS)科学研究費補助金基盤研究(B)「イオン駆動力供給体の電子線とX線による作動機構の解明 (研究代表者:米倉功治)」、同挑戦的萌芽研究「電子線結晶構造解析によるクーロンポテンシャルの可視化 (研究代表者:米倉功治)」、日本医療研究開発機構(AMED)医療研究開発革新基盤創成事業(CiCLE)(研究代表者:米倉功治)などによる支援を受けて行われました。
原論文情報
Hisashi Naitow, Tasuku Hamaguchi, Saori Maki-Yonekura, Masamichi Isogai, Nobuyuki Yoshikawa and Koji Yonekura, “Apple latent spherical virus structure with stable capsid frame supports quasi-stable protrusions expediting genome release”, Communications Biology, 10.1038/s42003-020-01217-4
発表者
理化学研究所
放射光科学研究センター 利用技術開拓研究部門 生体機構研究グループ
グループディレクター 米倉 功治(よねくら こうじ)
(科技ハブ産連本部 バトンゾーン研究推進プログラム 理研-JEOL連携センター 次世代電子顕微鏡開発連携 ユニット ユニットリーダー)
先任研究員 内藤 久志(ないとう ひさし)
研究員 浜口 祐(はまぐち たすく)
XFEL研究開発部門 ビームライン研究開発グループ イメージング開発チーム
研究員 眞木 さおり(まき さおり)
岩手大学 農学部 植物生命科学科 植物病理学分野 植物病理学研究室
教授 吉川 信幸(よしかわ のぶゆき)
准教授 磯貝 雅道(いそがい まさみち)
報道担当
理化学研究所 広報室 報道担当
岩手大学 総務広報課