2018-06-13 理化学研究所
理化学研究所(理研)開拓研究本部眞貝細胞記憶研究室の眞貝洋一主任研究員、福田渓基礎科学特別研究員らの国際共同研究チーム※は、ウイルスに由来するDNA配列であるレトロエレメント[1]の抑制に関わる新しい遺伝子を発見しました。
本研究成果は、遺伝子やウイルスを抑制する機構の解明に貢献すると期待できます。
哺乳動物のゲノムの約半分は、レトロエレメントというウイルスに由来するDNA配列で占められています。レトロエレメントの異常な発現は、近傍遺伝子の発現への影響や、転移によるゲノムの損傷を引き起こすため、その発現はエピジェネティック修飾[2]により抑制されています。しかし、その抑制機構の詳細は明らかではありません。
今回、国際共同研究チームは、CRISPR-Cas9システム[3]を用いた遺伝子ノックアウトスクリーニングにより、50以上もの新しいレトロエレメント抑制因子を同定しました。そして、それらのうちMORC2AとRESF1の分子機能を解析したところ、MORC2AとRESF1はレトロエレメント抑制機構の異なる段階で働くことが分かり、レトロエレメントの抑制が確立する機構の一端が明らかになりました。
本研究は、国際科学雑誌『Genome Research』オンライン版(5月4日付け)に掲載されました。
※国際共同研究チーム
理化学研究所 開拓研究本部 眞貝細胞記憶研究室
主任研究員 眞貝 洋一(しんかい よういち)
基礎科学特別研究員 福田 渓(ふくだ けい)
英国 ウェルカムサンガー研究所
グループリーダー 遊佐 宏介(ゆさ こうすけ)
埼玉医科大学ゲノム医学研究センター
教授 奥田 晶彦 (おくだ まさひこ)
※研究支援
本研究は、日本学術振興会(JSPS)科学研究費補助金基盤研究A「H3K9メチル化による転写抑制の実体の解明(研究代表者:眞貝洋一)」による支援を受けて行われました。
背景
哺乳動物のゲノムの約半分は、レトロウイルス[1]に由来する配列(レトロエレメント)で占められています。レトロエレメントには、LINE(長鎖散在反復配列)[4]やERV(内在性レトロウイルス)[5]などさまざまな種類があります。そして、レトロエレメントが異常に発現すると、近傍遺伝子の発現異常やゲノムの不安定性など宿主に有害な作用を引き起こすため、DNAやヒストンの化学修飾によって、その発現が抑えられています。
リジンメチル化酵素SETDB1によって媒介されるヒストンH3の9番目のリジンのトリメチル化(H3K9me3)は、レトロエレメントやウイルスの抑制に中心的な役割を果たすエピゲノム[2]であることが知られています。しかし、SETDB1が多種多様なレトロエレメントを抑制する詳しい機構は明らかになっていません。
研究手法と成果
CRISPR-Cas9システムは効率的に標的DNAに変異を導入することができるため、遺伝子ノックアウトスクリーニングに適しています。また、マウスES細胞[6]では、ウイルス由来DNAがSETDB1により効率的に抑制されることが知られています。そこで、国際共同研究グループは、SETDB1による転写抑制に関わる未知の遺伝子を探索するため、CRISPR-Cas9システムとマウスES細胞を用いた遺伝子ノックアウトスクリーニング系を開発しました(図1)。そしてスクリーニングの結果、50以上の新しい抑制遺伝子の同定に成功しました。
次に、レトロエレメント抑制機構の詳細を明らかにするため、マウスの細胞を用いて、機能が不明な新しい抑制遺伝子MORC2AとRESF1の機能解析を行いました。 まず、抑制因子MORC2AにはATPaseドメインとCWドメインがあり、CWドメインの機能は不明でした。ATPaseドメインあるいはCWドメインを欠損させた変異細胞の解析から、これらのドメインがMORC2Aによる転写抑制に必須であり、CWドメインを介したヒストンH3への結合により、MORC2Aは標的DNAに結合することが示されました。また、網羅的発現解析とクロマチン免疫沈降法[7]により、MORC2A欠損細胞ではレトロエレメントのH3K9me3に著しい減少が見られなかったことから、MORC2AはH3K9me3と共局在することで転写抑制機能を示すことが明らかになりました。このことは、MORC2AはH3K9me3依存的に転写抑制を行うことを示しています。 一方、抑制因子RESF1はSETDB1と結合しており、RESF1欠損細胞ではSETDB1のレトロエレメントへの結合およびH3K9me3が減少しました。したがって、RESF1にはSETDB1によるレトロエレメントへのH3K9me3の蓄積を助ける働きがあることが分かりました(図2)。
さらに、本研究で同定された種々のレトロエレメント抑制因子の欠損細胞における網羅的発現解析により、MORC2AはHUSH複合体[8]と協調してLINEを抑制すること、一方、RESF1はATRX-DAXX複合体[9]と協調してERVを抑制することが示されました(図3)。 以上のことから、MORC2AとRESF1は異なる機構によりレトロエレメントを抑制すること、そして、SETDB1はレトロエレメントのタイプによって異なる遺伝子と協調してその発現を抑制することが分かりました。
今後の期待
本研究のスクリーニングにより、数多くの新しいレトロエレメント抑制遺伝子を同定することができました。今後、新しい抑制遺伝子の機能解析により、ウイルスや遺伝子の発現抑制機構が明らかになり、抑制機構の破綻により生じるさまざまな病気の発症機序の解明につながると期待できます。
原論文情報
Kei Fukuda, Akihiko Okuda, Kosuke Yusa, Yoichi Shinkai, “A CRISPR knockout screen identifies SETDB1-target retroelement silencing factors in embryonic stem cells”, Genome Research, 10.1101/gr.227280.117.
発表者
理化学研究所
主任研究員研究室 眞貝細胞記憶研究室
主任研究員 眞貝 洋一(しんかい よういち)
基礎科学特別研究員 福田 渓(ふくだ けい)
報道担当
理化学研究所 広報室 報道担当
産業利用に関するお問い合わせ
理化学研究所 産業連携本部 連携推進部
補足説明
-
- レトロエレメント、レトロウイルス
- レトロウイルスは、DNA→RNAへの転写と、RNA→DNAへの逆転写によって増殖する性質を持つRNAウイルス。レトロエレメントは、逆転写反応によりレトロウイルスが染色体に取り込まれたDNA配列のこと。
-
- エピジェネティック修飾、エピゲノム
- エピジェネティック修飾は、DNAやヒストンタンパク質の化学修飾のこと。転写制御やDNA修復、染色体組換えなど、さまざまな生命現象に関わる。エピジェネティック修飾によって制御されるメチル化、アセチル化などの情報をエピゲノムという。
-
- CRISPR-Cas9システム
- 標的DNAを切断し、変異を導入するシステムで、標的DNAに相補的な配列を持つガイドRNA(gRNA)とDNA切断酵素のCas9で構成される。Cas9はgRNAと複合体を形成し、gRNAに相補的なゲノム領域に誘導され、その領域を切断する。
-
- LINE(長鎖散在反復配列)
- レトロエレメントの一種で、ヒトとマウスのゲノムの20%以上を占める。逆転写酵素とエンドヌクレアーゼを保持しており、自身のRNAをDNAに逆転写し、染色体に取り込ませることで、自らのコピーを増幅させる。LINEは、Long Interspersed Nuclear Elementの略。
-
- ERV(内在性レトロウイルス)
- レトロエレメントの一種で、ヒトゲノム中の約8%、マウスゲノムの約10%を占めると考えられる。Long Terminal Repeat (LTR)、Group-specific antigen (Gag)、Polymerase、Envelopeなどを持ち、レトロウイルスと似た構造をしている。外来性レトロウイルスが生物に感染し、生殖細胞ゲノムに取り込まれ、次世代に受け継がれて生物ゲノム中に定着したもの。ERVは、Endogenous Retrovirusesの略。
-
- ES細胞
- ES細胞は、哺乳類の着床前胚(胚盤胞)に存在する内部細胞隗から作成された、さまざまな種類の体細胞に分化する能力を持つ細胞。
-
- クロマチン免疫沈降法
- 生体内におけるタンパク質とゲノムDNAの結合部位を検出するための実験手法。ホルムアルデヒドによってDNAとDNAに結合しているタンパク質を架橋したのちに、DNAの断片化を行い、タンパク質の抗体を用いてタンパク質-DNA複合体を回収する。さらにDNAとタンパク質を脱架橋し、DNAのみを回収し配列を調べることでタンパク質がゲノムのどこに結合していたか分かる。
-
- HUSH複合体
- TASOR、MPP8、PPHLN1からなる複合体。H3K9me3領域を広げる働きがあり、ヒトにおいて、LINEの抑制に関わる。
-
- ATRX-DAXX複合体
- クロマチンリモデリング因子のATRXとヒストンシャペロンのDAXXからなる複合体。ヒストンバリアントのH3.3を染色体に入れ、レトロエレメントを抑制する。
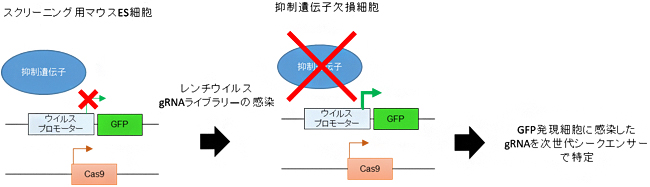
図1 レトロエレメント抑制遺伝子のノックアウトスクリーニング
レトロエレメントに由来するプロモーター配列の下流に緑色蛍光遺伝子(GFP)をつなげたDNAを、Cas9を発現するマウスES細胞に組み込む。通常は抑制遺伝子により、GFP遺伝子の発現は抑制されている。この細胞にレンチウイルスベクターを使って、ほぼ全ての遺伝子を網羅するガイドRNA(gRNA)ライブラリーを感染させると、抑制遺伝子に対応するgRNAが感染した細胞では、抑制遺伝子が欠損しGFPが発現する。GFP発現細胞に感染したgRNAを次世代シークエンサーで特定することで、抑制遺伝子を網羅的に特定することができる。
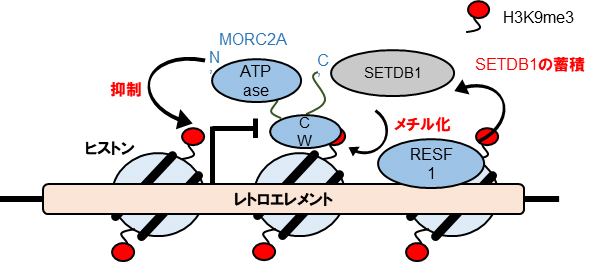
図2 レトロエレメント抑制モデル
新しい抑制因子のRESF1はリジンメチル化酵素SETDB1をレトロエレメントに蓄積させ、効率的にH3K9me3を行う。もう一つの抑制因子MORC2AはCWドメインを介してレトロエレメントに結合し、H3K9me3があると転写抑制能を示す。
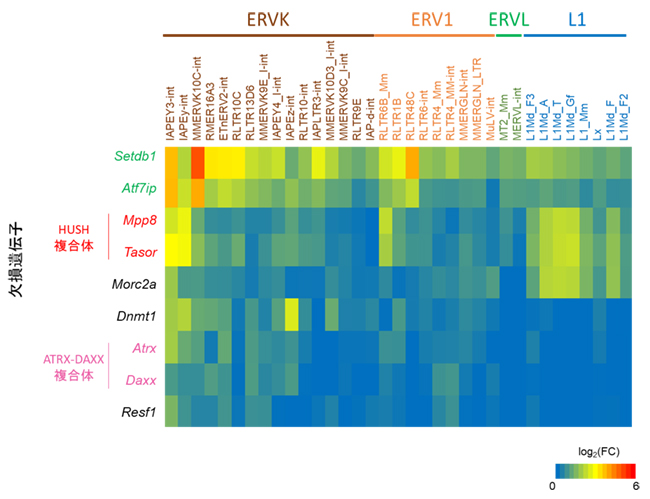
図3 レトロエレメント抑制遺伝子の欠損細胞におけるレトロエレメント発現解析
レトロエレメント抑制遺伝子欠損細胞におけるレトロエレメントの発現量をRNA-seqで解析した結果、MORC2AやHUSH複合体はLINEの一種であるL1を抑制する一方、RESF1やATRX-DAXX複合体はERVの一種のERVKを主に抑制することが明らかとなった。右下は発現変動の度合いを示す(赤くなるほどレトロエレメントの発現が大きく上昇したことを意味する)。