2021-08-19 鳥取大学,摂南大学,理化学研究所,日本医療研究開発機構
概要
鳥取大学大学院工学研究科の永野真吾教授ら、摂南大学農学部の加藤直樹准教授、国立開発法人理化学研究所(以下、理化学研究所)環境資源科学研究センター天然物生合成研究ユニットの高橋俊二ユニットリーダーら、同計算科学研究センター粒子系生物物理研究チームの杉田有治チームリーダーら、および東京大学大学院薬学系研究科の滝田良准教授らの共同研究グループは、ディールス・アルダー反応※1を触媒する2つの酵素が、天然物※2を合成する過程で鏡像異性体※3を作り分ける仕組みを解明しました。実験と理論計算を効果的に組み合わせたことで、ディールス・アルダー反応を触媒する酵素の形とその働きの関係性の理解を飛躍的に深めた本成果は、天然物誘導体の創出に向けた酵素の合理的デザインに指針を与えることが期待できます。
研究背景
ディールス・アルダー反応(Diels-Alder反応、[4+2]環化付加反応)は、その有用性から有機合成化学の分野で最も重要な反応の1つとして知られています。近年、天然物の生合成経路において、この反応を触媒する酵素が次々と発見されており、それらが自然界で複雑多様な環構造を作り出す上で重要な役割を担っていることがわかってきました。2015年に理化学研究所の加藤直樹研究員(研究当時、現摂南大学農学部准教授)が発見した「Fsa2」もその1つであり、ディールス・アルダー反応を触媒することでHIV-Iインテグラーゼ阻害活性を持つエキセチン※4のデカリン骨格※5を作ります。また、2018年に同研究員が発見したFsa2酵素群※6に属する「Phm7」は、エキセチンと鏡像関係にある類縁化合物フォマセチン※4のデカリン骨格を作ります。Fsa2とPhm7がどのように鏡像異性体の関係にあるデカリン骨格を作り分けているのか、その仕組みを明らかにすることが出来れば、立体配置の異なるデカリン骨格を持つ天然物誘導体を自在に創出できると期待されるため、本研究に着手しました。
研究内容・成果
今回、研究グループは鏡像異性体の関係にあるデカリン骨格を作るFsa2とPhm7(図1)に注目し、実験と理論計算の組み合わせによって、これら酵素が立体選択的なディールス・アルダー反応を触媒するメカニズムの解明に取り組みました。はじめに、研究グループはX線結晶構造解析※7によってPhm7とFsa2の立体構造を決定し、両酵素の中心に基質が結合するくぼみ(基質結合ポケット)が存在することを明らかにしました(図2)。Fsa2およびPhm7両酵素の基質結合ポケットは非常に大きく、直鎖状の長い基質を収容できることがわかりました。しかしながら、「鎖」のように長くて柔軟な構造の基質が結合ポケットにどのように収まるか(基質結合様式)が分からず、酵素がデカリン骨格の形を作り分ける仕組みについて十分な議論ができませんでした。
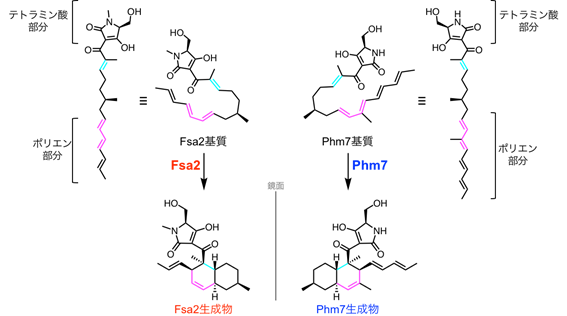
図1 Fsa2とPhm7による立体選択的なディールス・アルダー反応Fsa2、およびPhm7の各基質はテトラミン酸部分とポリエン部分、およびそれらを連結する中間部分からなる直鎖状の構造をしている。これらが折りたたまれた後にFsa2とPhm7によって立体選択的なディールス・アルダー反応が進行し、それぞれFsa2生成物、Phm7生成物へと変換される。構造式中のシアン、マゼンタ部分はそれぞれ基質では反応に関わるアルケンとジエンを、生成物ではアルケンとジエンに由来する部分を示している。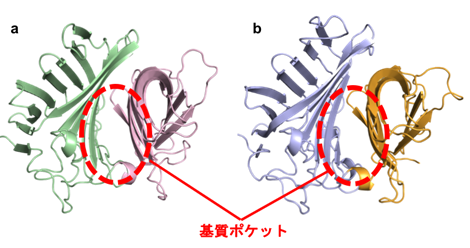
図2 Fsa2(a)とPhm7(b)の結晶構造Fsa2とPhm7は2つのドメイン(タンパク質を構成する複数のアミノ酸によって構築された1つのまとまり)で構成されている。緑とピンクはFsa2の各ドメインを、青とオレンジはPhm7の各ドメインを示している。Fsa2とPhm7の基質ポケットは2つのドメインの間に存在する。
そのため、研究グループは分子動力学(MD)シミュレーション※8により、基質の結合様式を推定しました。通常のMDシミュレーションとは異なり、幅広い構造空間を効率よく探索するレプリカ交換MD法の1つgREST法※8を用いることで、鎖のように折れ曲がることができる基質分子の多様な結合様式を網羅的に調べました。その結果、Fsa2およびPhm7に取り込まれた基質は、どちらもテトラミン酸部分とポリエン部分がそれぞれポケットの手前と奥側に配置された結合様式であると推定されました(図3)。また、2つの酵素では基質の結合様式に明らかな違いが見られ、それぞれがエキセチン、もしくはフォマセチンのデカリン骨格の構造を擬似的に再現しており、ディールス・アルダー反応で鏡像異性体の関係にあるデカリン骨格を作り出せる状態にありました。つまり、基質の結合様式がこれら酵素の触媒するディールス・アルダー反応の立体選択性を決定していると推定されました。
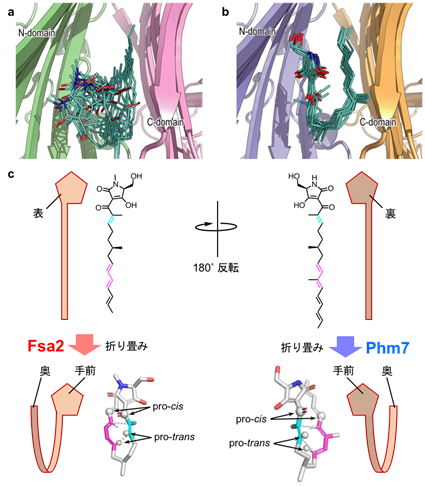
図3 gREST法により推定したPhm7とFsa2の基質の結合モデルMDシミュレーションでは、生体分子の動きを原子レベルで調べることができ、酵素に取り込まれた基質の多様な結合様式を推定することができる。aはFsa2、bはPhm7に取り込まれた基質の推定結合様式を示している。中央の棒状のモデルがそれぞれの基質を示している。Fsa2では基質結合ポケット内で基質が多様な基質結合様式をとることが推定されたが、これに対し、Phm7では基質結合様式が収束していることが分かった。cはgREST法の結果により推定されたFsa2とPhm7のポケット内での基質の折りたたみモデルを示している。五員環のテトラミン酸部分を基準にすると、Fsa2とPhm7の両者では、結合した基質のポリエン部分が奥側に折りたたまれる。この時、Phm7では裏返された基質が折りたたまれることで、鏡像異性体が作り分けられることが説明できる。
次に、gREST法によるシミュレーションで得られた基質結合様式が妥当であることを実験で確認するために、部位特異的変異導入により作製した変異酵素を用いた解析※9を行いました。基質との相互作用への関与が予想されたアミノ酸残基に変異を導入した変異酵素では、酵素活性が低下しており、シミュレーションで得られた基質結合モデルが妥当であることが示されました。
さらに、研究グループは密度汎関数理論(DFT)計算※10を用いて、Phm7が触媒するディールス・アルダー反応の立体選択性を詳細に調べました。その結果、反応の立体選択性をPhm7が制御していることが確認出来ました。また、図3で示したgREST法で得られているPhm7の基質結合モデルでは、反応に関与する基質のC1およびC2’のカルボニル基(図4a)がPhm7の356番目のリジン残基(K356)や82番目のグルタミン酸残基(E82)と水素結合ネットワークを形成していました。これらの相互作用を考慮したDFT計算から、フォマセチン型のデカリン骨格を作るディールス・アルダー反応が加速されていることが見積もられました(図4b)。この結果を受け、E82やK356についても同様に部位特異的変異を導入した変異酵素を作製し評価したところ、それぞれの変異酵素では酵素活性が大幅に低下していることが確認されました。このことから、Phm7はディールス・アルダー反応を加速させる触媒残基を有していることが明らかとなりました。
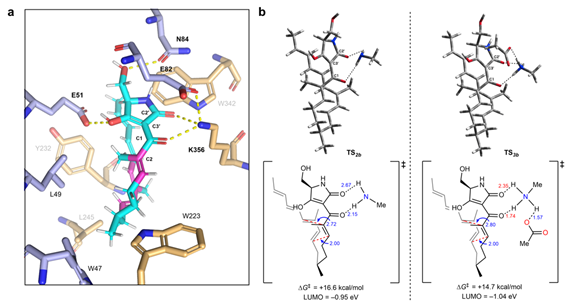
図4 gREST法によるPhm7の基質の推定結合様式とDFT計算で求めた遷移状態構造aはMDシミュレーションで推定されたPhm7に取り込まれた基質の結合様式を示している。基質はK356と直接相互作用し、K356を介してE82とも相互作用していた。bはK356、およびE82とK356の両アミノ酸残基の影響を考慮したDFT計算により求めた遷移状態構造を示している。活性化エネルギー(∆G‡)は、アミノ酸残基の影響を考慮しない場合(+16.5 kcal/mol)と、K356との水素結合を考慮した場合(∆G‡は+16.6 kcal/mol)とではほとんど変化がなかった。一方、K356に加えE82も含めた水素結合ネットワークが関与すると∆G‡は+14.7 kcal/molまで減少しており、Phm7のこれらのアミノ酸残基によって反応が加速されることが示された。
以上の結果から、本研究では実験と理論計算の効果的な組み合わせにより、Fsa2とPhm7による立体選択的なディールス・アルダー反応のメカニズムや、Phm7の触媒残基の存在を明らかにしました。
本研究成果はドイツ化学会誌『Angewandte Chemie International Edition』への掲載に先立ち、2021年6月13日にオンラインで公開されました。また、近年急速に発展し高い関心が持たれている分野の中でも重要な論文として位置づけられる『Hot Paper』として選出され、カバーイラスト(インサイドカバー)にも採用されました(図5)。

図5 Inside Coverの図
今後の展望
近年、ディールス・アルダー反応により天然物の環状骨格を作る酵素が次々と見つかっており、それら酵素の分子メカニズムは注目を集めています。本研究による基本的かつ重要な発見は、天然物の生合成に関わるディールス・アルダー反応を触媒する酵素に共通する機能の全容解明に向けた研究を促進するだけでなく、変異酵素のデザインやそれらの産業応用など、研究の幅広い展開が期待されます。
共同研究グループ
- 鳥取大学大学院工学研究科
- 教授 永野 真吾
准教授 日野 智也
大学院生 藤山 敬介(現日本学術振興会特別研究員PD、理化学研究所 環境資源科学研究センター 適応制御研究ユニット所属) - 摂南大学農学部
- 准教授 加藤 直樹
- 国立研究開発法人医薬基盤・健康・栄養研究所AI健康・医薬研究センター
- プロジェクト研究員 李 秀栄
- 理化学研究所
- 環境資源科学研究センター 天然物生合成研究ユニット
ユニットリーダー 高橋 俊二
大学院生リサーチ・アソシエイト 衣笠 清美 - 計算科学研究センター 粒子系生物物理研究チーム
チームリーダー 杉田 有治 - 環境資源科学研究センター ケミカルバイオロジー研究グループ
グループディレクター 長田 裕之
研究員 野川 俊彦 - 東京大学大学院薬学系研究科
- 准教授 滝田 良
特任助教 渡邉 康平
原論文情報
- 著者
- Keisuke Fujiyama, Naoki Kato, Suyong Re, Kiyomi Kinugasa, Kohei Watanabe, Ryo Takita, Toshihiko Nogawa, Tomoya Hino, Hiroyuki Osada, Yuji Sugita, Shunji Takahashi, and Shingo Nagano
- タイトル
- “Molecular Basis for Two Stereoselective Diels-Alderases that Produce Decalin Skeletons”.
- 掲載誌
- Angewandte Chemie International Edition, 2021,
- DOI
- 10.1002/anie.202106186
研究支援
本研究は、日本学術振興会科学研究費補助事業の各課題番号「19H04665および20K05872、研究代表者:加藤直樹」、「19K12229、研究代表者:李秀栄」、「19K06992、研究代表者:滝田良」、「20K15273、研究代表者:渡邉康平」、「19H05645、研究代表者:杉田有治」、「20H00416、研究代表者:高橋俊二」、「19H04658および19H05780、研究代表者:永野真吾」の支援を受けて行われました。また、X線結晶構造解析は、日本医療研究開発機構(AMED)創薬等ライフサイエンス研究支援基盤事業(BINDS)の課題番号JP21am0101070による支援を受け、高輝度放射光施設「SPring-8」のビームラインBL32XU及びBL41XUで実施されました。分子動力学シミュレーションは、理研HOKUSAI GreatWave/BigWaterfall、HPCIシステム利用研究課題(課題番号:hp190181)を通じてスーパーコンピュータ「京」の計算資源の提供を受けて実施されました。DFT計算は自然科学研究機構 計算科学研究センターの計算資源の提供を受けて実施されました。
用語解説
- ※1 ディールス・アルダー反応
- ジエンとアルケン(この場合は親ジエン体と呼ばれる)の間で電子が移動することで起こる反応。同時に2箇所の炭素-炭素結合を生み出すことから、有機合成の中でも非常に強力な反応として利用されている。1950年のノーベル化学賞の受賞理由となった有機化学における代表的な反応としても知られている。
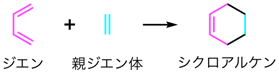
補足図1 典型的なディールス・アルダー反応の模式図 - ※2 天然物
- 天然物は主に植物や微生物によって産生される化合物を指す。天然物は香料や医薬品、およびそれらの原料などに利用されることが多く、それらのほとんどは微生物による発酵技術や有機合成によって大量生産される。一部の天然物については、構造が複雑で合成等が困難なため、それらは現代でも生物から直接抽出されたものが供給されている。
- ※3 鏡像異性、鏡像異性体
- 化学的な組成(分子式)は同一だが、立体的な構造が異なる化合物の関係を立体異性体という。鏡像異性体は立体異性体の1つであり、文字通り、互いの構造を鏡に映したような関係を指す。鏡像異性体はよく「右手」と「左手」の関係に例えられる。このように、対象を回転させた場合でも完全に重ね合わせができない「鏡像」の関係を指す。
乳酸には1つの鏡像異性体が存在する。鏡像異性体は規則に基づいてS体とR体に分類される。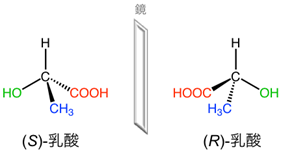
補足図2 乳酸の鏡像異性体と構造 - ※4 エキセチン、フォマセチン
- エキセチンはフザリウム属糸状菌から単離された化合物で、HIV-Iインテグラーゼ阻害活性を示すことが知られている。フォマセチンはエキセチンと鏡像異性の関係にあるデカリン骨格を有しており、エキセチンと同様にHIV-Iインテグラーゼ阻害活性を持つ。
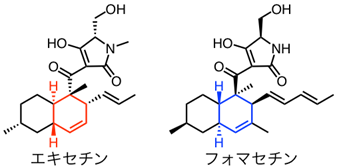
補足図3 エキセチンとフォマセチンの化学構造 - ※5 デカリン骨格
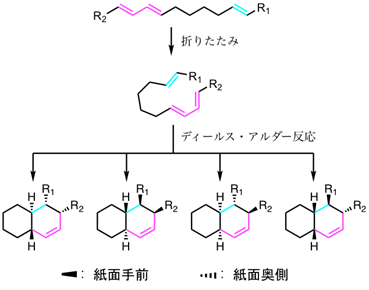
- デカリン骨格は10個の炭素原子から構成され、2つの環構造を有している。デカリン骨格は立体的な構造が異なる異性体(立体異性体)がいくつか存在するが、共通する直鎖状ポリエン基質からディールス・アルダー反応を介して構築されるデカリン骨格は4種類存在する。
補足図4 ディールス・アルダー反応によって生産されるデカリン骨格の構造 - ※6 Fsa2酵素群
- 多くのアミノ酸から構成されるタンパク質には、機能や構造が類似したタンパク質が複数存在している。Fsa2酵素群はFsa2と類似した構造と機能を持つタンパク質群を指しており、現在では、様々な糸状菌からFsa2酵素群に分類されると考えられる遺伝子が見つかっている。
- ※7 X線結晶構造解析
- 結晶は分子が3次元的に規則正しく並んだ状態であり、結晶に対してX線を照射することで回折と呼ばれる現象が起こる。回折によって得られる斑点状の回折の強度には、結晶を構成する分子の構造に由来する情報が反映されているため、回折像を解析することで、結晶を構成する分子の立体構造を求めることができる。X線結晶構造解析ではタンパク質の結晶を作製する困難を伴うが、立体構造を決定する上で有効な手段の一つとしてよく用いられている。
一般的に、酵素反応は「鍵」と「鍵穴」の関係で例えられる。「鍵」である基質が「鍵穴」である酵素(の基質結合ポケット)に対してどのように結合するかを調べることは、酵素反応のメカニズムを明らかにする第一歩である。多くの場合、「鍵穴」の構造だけでは、基質がどのように結合しているか分からないため、基質や生成物、あるいはその類似物質が結合した酵素の結晶構造解析が行われる。 - ※8 MDシミュレーション、gREST
- 分子動力学(MD)シミュレーションは、原子間に働く力を計算し、運動方程式を繰り返し解くことで、分子の動きを追跡する方法。MDはMolecular Dynamicsの略。タンパク質のように複雑な分子のMDシミュレーションは、しばしば局所的な安定状態に捕らわれてしまい、可能な立体構造を十分に調べることができない。レプリカ交換MD法は、構造探索効率を上げるための一つの方法で、系の複数コピー(レプリカ)に対して温度の異なるMD計算を実施して、レプリカ間で温度を適宜交換し構造探索の効率を上げる。少ないレプリカ数で構造探索の効率を上げるために考案されたREST法(Replica-Exchange with Solute Temperingの略)では、系の一部(溶質分子)の温度のみ異なるレプリカを用いることで必要となるレプリカ数を抑える。本研究で用いたgREST法(generalized RESTの略)は、さらに「溶質分子」の定義を拡張した方法で、タンパク質―基質の結合予測などに有効である。
- ※9 部位特異的変異導入
- 部位特異的変異導入(site-directed mutagenesis)は、人為的に変異導入したタンパク質と、変異のない本来のタンパク質(野生型)の機能を比較し、その変異したアミノ酸残基の機能や役割などを調べる手法。酵素の研究では野生型の酵素とその変異酵素の活性を比較することで、基質の結合や酵素反応の触媒に関わるアミノ酸残基など、機能的に重要なアミノ酸残基であることを確認することができる。多くの場合、基質と酵素を混ぜて酵素反応を起こし、得られた反応物の量を分析・比較することで評価される。Fsa2やPhm7の基質は化学合成による調製が困難であったため、研究グループはフォマセチン生産糸状菌に由来するphm7遺伝子欠失株を作製し、菌体内にPhm7の基質を蓄積させ、その細胞抽出液を基質溶液として用いることで、試験管内での酵素活性測定を可能にした。
- ※10 密度汎関数理論(DFT)計算
- 「密度汎関数理論計算」は、原子や分子などの物性を明らかにする量子化学計算手法の一つ。例えば、今回の研究では分子の構造変化のエネルギーを明らかにし、反応経路の検証に用いられている。また、反応が進行する際に最もエネルギーが高い状態を「遷移状態」と呼び、基底状態とのエネルギー差が「活性化エネルギー」である(図4)。遷移状態は、エネルギーが高く不安定なため実験的には観測は困難であるが、量子化学計算によりその構造を見ることができる。密度汎関数法は、正確なエネルギーが電子密度の汎関数として一意的に定められているというHohenberg-Kohn定理に基づいている。計算コストに対して、比較的良い計算精度が得られることから、近年化学分野で広く活用されている。
お問い合わせ先
研究内容に関する問い合わせ
鳥取大学大学院工学研究科
教授 永野 真吾
摂南大学農学部
准教授 加藤 直樹
理化学研究所
環境資源科学研究センター 天然物生合成研究ユニット
ユニットリーダー 高橋 俊二
計算科学研究センター 粒子系生物物理研究チーム
チームリーダー 杉田 有治
東京大学大学院薬学研究科
准教授 滝田 良
各機関への問い合わせ
鳥取大学総務企画部総務企画課広報企画室
常翔学園 広報室(摂南大学担当)
理化学研究所 広報室 報道担当
AMED事業の問い合わせ
日本医療研究開発機構(AMED)創薬事業部 医薬品研究開発課
創薬等ライフサイエンス研究支援基盤事業(BINDS)