心筋症の臨床的意義不明変異のメカニズム解明に貢献
2021-09-03 理化学研究所
理化学研究所(理研)生命医科学研究センター循環器ゲノミクス・インフォマティクス研究チームの伊藤薫チームリーダーらが参加する国際共同研究グループは、難病である特発性拡張型心筋症(DCM)[1]におけるタイチン遺伝子(TTN遺伝子)[2]の「臨床的意義不明変異(VUS)[3]」を解析し、DCM患者では「異常RNAスプライシング[4]」の変異が重要であることを明らかにしました。
本研究成果は、臨床遺伝子診断で問題となっているVUSの臨床的意義を解明するのに役立つものと期待できます。
今回、国際共同研究グループは、DCM患者203人と対照者3,329人のTTN遺伝子エクソンの遺伝子高発現部位に存在するVUSを同定しました。これらのVUSがRNAスプライシングに及ぼす影響をin silico[5]スプライス予測ツールで優先順位を付け、in vitroアッセイ[6]で評価したところ、異常RNAスプライシングVUSは対照群よりもDCM患者群に多く見られました。さらに、この方法を臨床遺伝子変異データベースに適用した結果、TTN遺伝子のスプライス部位[7]に存在する類似VUSのうち約20%がRNAスプライシングに影響を与えることが分かりました。また、SpliceAI[8]は、異常RNAスプライシングVUSの検出において高い陽性的中率を示したものの、感度は低いことが分かりました。さらに、TTN遺伝子の異常RNAスプライシングVUSはDCM症例の1~2%に存在し、DCM症例のTTN遺伝子スプライス部位VUSの診断力を10~20%向上させることを示しました。
本研究成果は、科学雑誌『Circulation: Genomic and Precision Medicine』オンライン版(8月31日付)に掲載されました。
背景
拡張型心筋症(DCM)は、収縮機能障害、心室肥大、心不全を特徴とする進行性の心筋疾患で、30~50%の患者に疾患の原因となる遺伝子変異があると考えられています。近年、次世代シークエンサー[9]を用いた臨床研究により、ヒト最大のタンパク質であるタイチン(TTN)をコードするTTN遺伝子の変異が、DCMの主要な遺伝的原因であることが明らかになりました。この変異の中で、「TTN切断変異[10](TTNtv)」は家族性発症の25~30%を占めています。しかし、実際の遺伝子検査では、TTNtv以外のまれな同義変異[11]、ミスセンス変異[12]またはタンパク質をコードしないイントロンの遺伝子変異が検出されることが多く、その病原性は不明です。
国際共同研究グループはこれまでに、「異常RNAスプライシング」を阻害する遺伝子変異を同定する方法を開発しました。遺伝子変異のうち、臨床的に病気を引き起こすものか、それとも良性のものか不明な変異を「臨床的意義不明変異(VUS)」と呼びます。今回、TTN遺伝子のスプライス部位近くのVUSを解析することで、疾患を引き起こす遺伝子変異の範囲を拡大し、VUSを定義できるのではないかという仮説を立てました。さらに、TTN遺伝子のタンパク質をコードするエクソン数は多く、通常利用されるスプライス部位は数百にも及ぶことから、研究データに存在するパターンがRNAスプライシングの生物学に関する洞察を与え、それにより他の疾患遺伝子の解釈に役立つのではないかと考えました。
研究手法と成果
国際共同研究グループはまず、DCM患者群と対照群においてTTN異常RNAスプライシングを引き起こすVUSの割合を比較するために、次世代シークエンサーを用いて、DCM患者203人と対照者3,329人からTTN遺伝子変異を検出しました。その結果、DCM患者群の59.6%、対照群の39.2%に、希少なTTN遺伝子変異が同定されました。そして予想通り、DCM患者群では、エクソンの遺伝子高発現部位に存在するTTNtvが有意に濃縮されていました(DCM患者群:対照群=23.1%:0.5%、p=3.7×10-47、フィッシャーの正確検定[13])。また、これらのコホート(集団)におけるその他の希少遺伝子の変異のうち、DCM患者群の3.4%と対照群の1.1%が既知のスプライス部位の近くに希少なVUSを持っていました。
これらを全てのVUSを、in silicoスプライス予測ツールを用いて優先順位を付け、in vitroアッセイでRNAスプライシングに対する影響に評価しました。その結果、DCM患者群では2.0%、対照群では0.2%の割合で、異常RNAスプライシングを引き起こすVUS(異常RNAスプライシングVUS)が存在することが分かりました(p=0.002、フィッシャーの正確検定)(下図)。
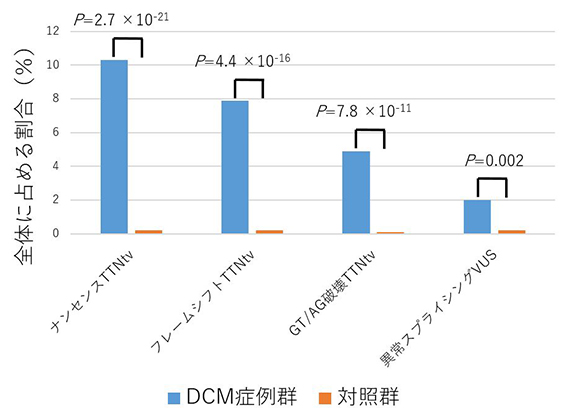
DCM群と対照群の臨床的意義不明変異(VUS)中の異常RNAスプライシング変異の割合
DCM患者群ではTTN切断変異(TTNtv)が多いことは知られていたが(左3本)、異常RNAスプライシングを引き起こすVUSもDCM患者群で有意に多いことが示された(右端)。
続いて、二つのヒト遺伝的変異大規模データベースにより、異常RNAスプライシングVUSを特定しました。一つは米国パートナーヘルスケアの分子医学研究室が提供するデータベース、もう一つは米国国立衛生研究所が提供するClinvarデータベースであり、それぞれ6,882個、3,070個のTTN遺伝子変異が報告されていました。それらの中でエクソンの遺伝子高発現部位のスプライス部位の近くに存在する201個のVUSを評価したところ、45個(22.4%)は異常RNAスプライシングを引き起こす可能性が高いと判断され、残りの156個(77.6%)は良性の可能性が高いと判断されました。これは、元々報告されていた異常RNAスプライシングVUSの総数(28個)を約60%も増加させる結果となりました。
さらに、TTN遺伝子に加えて心筋症で重要とされるMYBPC3遺伝子とLMNA遺伝子の三つについて、同様の手法で調べた異常RNAスプライシングVUSのデータを、現在の異常RNAスプライシング検出のベンチマークであるin silico予測ツールSpliceAIの結果注1)と比較しました。すると、SpliceAIスコア[14]の閾値を0.2以上にすると、陽性予測値(PPV)[15]が85.7%、陰性予測値(NPV)[16]が80.9%、感度[17]が49.2%となりました。一方、SpliceAIスコアの閾値を0.8以上に設定すると、PPVは94.7%に向上しましたが、NPVは72.3%、感度は14.8%に低下しました。この結果から、現在のin silico予測だけでは、PPVは高いが感度は低いということが判明しました。
以上のように本研究では、in silicoスプライス予測ツールによる優先順位付けとin vitroアッセイを用いて、TTN遺伝子のスプライス部位近くに存在する異常RNAスプライシングを引き起こす変異がTTNtv心筋症に寄与するかどうかを調べ、TTN遺伝子のスプライス部位のVUSの約20%が正常なmRNAのスプライシングを妨げていることを明らかにしました。加えて、これらの異常RNAスプライシングVUSがDCM患者群で有意に多く、DCM症例の1~2%に存在し、DCM症例のTTN遺伝子スプライス部位VUSの診断力を10~20%向上させることを示しました。
注1)Jaganathan K et al. Predicting Splicing from Primary Sequence with Deep Learning. Cell 176:535,2019
今後の期待
本研究で得られたデータは、一般に公開されているデータベースの遺伝子変異を再分類したもので、異常スプライシングを引き起こす遺伝子変異群を同定しました。臨床医は遺伝子変異の病原性を判断するために、これらの公的リソースに依存しているため、今回の結果を各データベースに含めることで、最終的にはこれらの発見を臨床に応用できるものと期待できます。
さらに、これらのデータは、機能テストが行われていない他のVUSの病原性/非病原性を判断しようとしている臨床検査機関に、in silico予測だけでなく実際の機能解析の必要性があることを示しました。
補足説明
1.特発性拡張型心筋症(DCM)
心臓は拡張と収縮を繰り返すことで、全身に血液を送り出すポンプとしての機能を果たしているが、特発性拡張型心筋症は心臓にダメージを与える疾患、例えば心臓の弁の異常や心筋梗塞などの病気がなくても心臓の筋肉の収縮力が低下し、心臓が拡張してしまう病気である。この病気は慢性進行性であり、最終的には心移植が必要となる難治性疾患である。DCMはdilated cardiomyopathyの略。
2.タイチン遺伝子(TTN遺伝子)
心臓の筋肉の収縮に必要なサルコメアという収縮構造を構成するタンパク質をコードする遺伝子の一つである。DCMの原因として最も多いことが知られているだけでなく、アルコール心筋症、産褥性心筋症など、心筋脆弱性を規定する重要な遺伝子として注目されている。
3.臨床的意義不明変異(VUS)
近年、次世代シークエンサーの登場により、何千万という遺伝子変異が一度に検出できるようになったが、それらが臨床的に病気を引き起こすものであるのか、それとも良性のものであるのか不明なものを指す。次世代シークエンサーの単価の低下とともに臨床研究に多く用いられるようになったため、VUSが増加してきており、近年の臨床遺伝診断で重要な問題となっている。VUS はVariant of Unknown Significanceの略。
4.RNAスプライシング
ヒトの遺伝情報は、DNAを鋳型にしてRNAに転写されタンパク質に翻訳されるという生命のセントラルドグマに従っている。この過程で、RNAはタンパク質をコードするエクソンとそうでないイントロン部分に分けられるが、タンパク質への翻訳の際にイントロン部分は除去され、それをRNAスプラシングと呼ぶ。
5.in silico
実際に実験を行うことなく、コンピューターを用いた計算で生物学的挙動を予測すること。SpliceAIはin silicoで異常RNAスプライシングの発生を予測している。
6.in vitroアッセイ
in silicoとは反対に、実際に細胞実験などにより生物学的挙動を確かめる手法。
7.スプライス部位
RNAスプラシングが起きる開始部位または終了部位の総称。具体的にはタンパク質をコードするDNA領域であるエクソンとタンパク質をコードしないDNA領域であるイントロンの境界部位にあたる。
8.SpliceAI
2019年に米国イルミナ社が中心となって発表した、異常RNAスプライシングを引き起こす遺伝子変異を予測する深層学習アルゴリズム。従来の予測アルゴリズムの性能を大きく上回り、現在の予測ツールのゴールデンスタンダードとなっている。
9.次世代シークエンサー
過去のある1点の塩基や数百塩基対を読む古典的なシークエンス法とは異なり、一度にヒトゲノム全体のDNA配列を読めるほどのアウトプットを誇るシークエンサー。次世代シークエンサーは、米国イルミナ社が出している一つのリード長が短い第2世代シークエンサーと、第2世代シークエンサーの弱点であるリード長の短さを克服した第3世代シークエンサーに分けられる。本研究では、第2世代シークエンサーを用いた。
10.切断変異
遺伝子変異はタンパク質に与える影響により分類される。切断変異は、タンパク質の翻訳が途中で止まってしまうストップゲイン変異、タンパク質の読み枠がずれてしまうフレームシフト変異、RNAスプライシングに異常を来す変異(イントロン開始2塩基のGTやイントロン終了2塩基のAGを置換する)などが該当する。
11.同義変異
DNA配列は変化するが、翻訳されるタンパク質のアミノ酸が変化しないもの。一般に良性の変異とされる。
12.ミスセンス変異
DNA配列の変化とともに、翻訳されるタンパク質のアミノ酸が置換されるもの。アミノ酸の置換の種類により生物学的影響の大きさはさまざまであるが、一般に切断変異よりは良性のことが多い。
13.フィッシャーの正確検定
二つの集団の2カテゴリーに分類されたデータを扱う場合に、「二つの集団における2カテゴリーの人数の割合は等しい」という帰無仮説を検定する。本研究では、DCM患者群と対照群で、異常RNAスプライシングを起こす遺伝子変異の割合が有意に異なるかを検定した。
14.SpliceAIスコア
SpliceAIアルゴリズムは、異常RNAスプライシングが起こる確率を0から1までのスコアで与える。0は可能性なし、1は最も可能性があることを意味しており、目的により閾値を設定して使う。原著論文では、スクリーニングには0.2を閾値とする、つまりスコアが0.2を超えたものを、異常RNAスプライシングを起こす可能性があると判断することを薦めている。
15.陽性予測値(PPV)
ある検査や予測において「陽性と判定された場合に、真の陽性である確率」として定義される値を指す。PPVはPositive Predictive Value の略。
16.陰性予測値(NPV)
陽性予測値とは反対に、ある検査や予測において「陰性と判定された場合に、真の陰性である確率」として定義される値を指す。NPVはNegative Predictive Valueの略。
17.感度
ある検査や予測において、「陽性と判定されるべきものを、正しく陽性と判定する確率」として定義される値を指す。
国際共同研究グループ
理化学研究所 生命医科学研究センター
循環器ゲノミクス・インフォマティクス研究チーム
チームリーダー 伊藤 薫(いとう かおる)
ハーバード大学 メディカル・スクール
研究員 パース・パテル(Parth Patel)
教授 ジョン・サイドマン(Jon Seidman)
教授 クリスティーン・サイドマン(Christine Seidman)
本研究は、日本、米国、英国、オーストラリア、シンガポールの研究機関に所属する17人の研究者が参加して行われました。
原論文情報
Parth N. Patel, Kaoru Ito, Jon A. L. Willcox, Alireza Haghighi, Min Young Jang, Joshua M. Gorham, Steven R. DePalma, Lien Lam, Barbara McDonough, Renee Johnson, Neal K. Lakdawala, Amy Roberts, Paul J.R. Barton, Stuart A. Cook, Diane Fatkin, Christine E. Seidman and J. G. Seidman., “Contribution of Noncanonical Splice Variants TTN Truncating Variants Cardiomyopathy”, Circulation: Genomic and Precision Medicine, 10.1161/CIRCGEN.121.003389
発表者
理化学研究所
生命医科学研究センター 循環器ゲノミクス・インフォマティクス研究チーム
チームリーダー 伊藤 薫(いとう かおる)
報道担当
理化学研究所 広報室 報道担当