2023-12-04 東京大学
発表のポイント
- アゴハゼの種内系統の1つが、絶滅した“ゴースト系統”との古代の交雑により誕生したことを示しました。
- 絶滅したゴースト系統由来のゲノムが受け継がれたゲノム領域や、逆にゴースト系統由来のゲノムが失われてしまったゲノム領域の特徴を初めて解明しました。
- 本研究の成果は、絶滅系統が現在の生物多様性に果たす役割や、生物の交雑ゲノム構成を決める普遍的なルールの解明に繋がると期待されます。

本解析の主役であるハゼ科魚類のアゴハゼ
このごくありふれた魚のゲノムに「幽霊」が潜んでいた
発表内容
東京大学大学院農学生命科学研究科附属水産実験所の加藤柊也大学院生、平瀬祥太朗助教、菊池潔教授らの研究グループは、アゴハゼというハゼ科魚類の種内系統の1つが絶滅した“ゴースト系統(注1)”との交雑により誕生した系統であることを示しました。さらに、この種内系統について詳細な集団ゲノミクス解析(注2)を行うことで、絶滅系統のゲノムが生き残ったゲノム領域や、逆に絶滅系統由来のゲノムが失われてしまったゲノム領域の特徴を解明しました。
研究の背景
生命の進化の歴史は絶え間のない試行錯誤の歴史であり、これまでに誕生したほとんどの生物系統は既に絶滅しています。こうした絶滅系統は既にこの世にはいないため、化石のような稀な記録がない限り、我々がその存在を知ることは困難です。しかし、近年では、現存系統の祖先と絶滅系統の間に生じた古代の交雑と遺伝子浸透(注3)により、現在の生物のゲノムに絶滅系統のゲノムの一部が受け継がれうることが明らかになってきました。例えば、我々ヒト自身のゲノムにも、絶滅した古代人類に由来するゲノムがごくわずかに生き残っています。こうした絶滅系統のゲノムがどの領域で生き残り、どの領域で失われたかという遺伝子浸透のゲノミックランドスケープ(注4;遺伝子浸透ランドスケープ)を調べることは、絶滅系統が現在の生物多様性に与える影響を理解するうえで重要です。しかし、古代人類のような稀なケースを除いて、我々は基本的に絶滅系統のDNAを入手することができません。このような現時点でサンプリングできない系統を“ゴースト系統”と呼びますが、手元に存在しないゴースト系統を研究することは難しく、ゴースト系統に由来するゲノム領域を特定するどころか、本当にゴースト系統との交雑があったかを確かめることさえ一苦労です。こうした困難ゆえに、絶滅系統由来のゲノムの分布についてはほとんど研究されてきませんでした。
本研究では、アゴハゼ(Chaenogobius annularis)という魚類に着目し、この困難な課題に挑みました。アゴハゼは日本列島周辺の岩礁性海岸に生息する小型のハゼ科魚類で、海水浴場の磯遊びでも簡単に観察できる普通種です。これまでの分子系統地理学(注5)的研究により、本種には過去の日本海隔離により誕生した日本海系統と太平洋系統、そしてこれら2系統の交雑で誕生した可能性がある東シナ海系統の3つの種内系統が発見されていました。特にこのうちの東シナ海系統は、日本海系統と太平洋系統の直接の交雑ではなく、太平洋系統に近縁なゴースト系統との交雑により誕生した可能性が疑われていました。そこで私たちは、この東シナ海系統のゴースト系統起源仮説を検証するとともに、ゴースト系統由来の遺伝子浸透ランドスケープを特徴づけることを目的として、集団ゲノミクス解析を行いました。
研究の内容
まず、日本全国のアゴハゼについてRADシーケンス(注6)に基づく集団構造解析を行い、アゴハゼの3つの種内系統と系統間の交雑集団の分布を正確に把握しました(図1)。
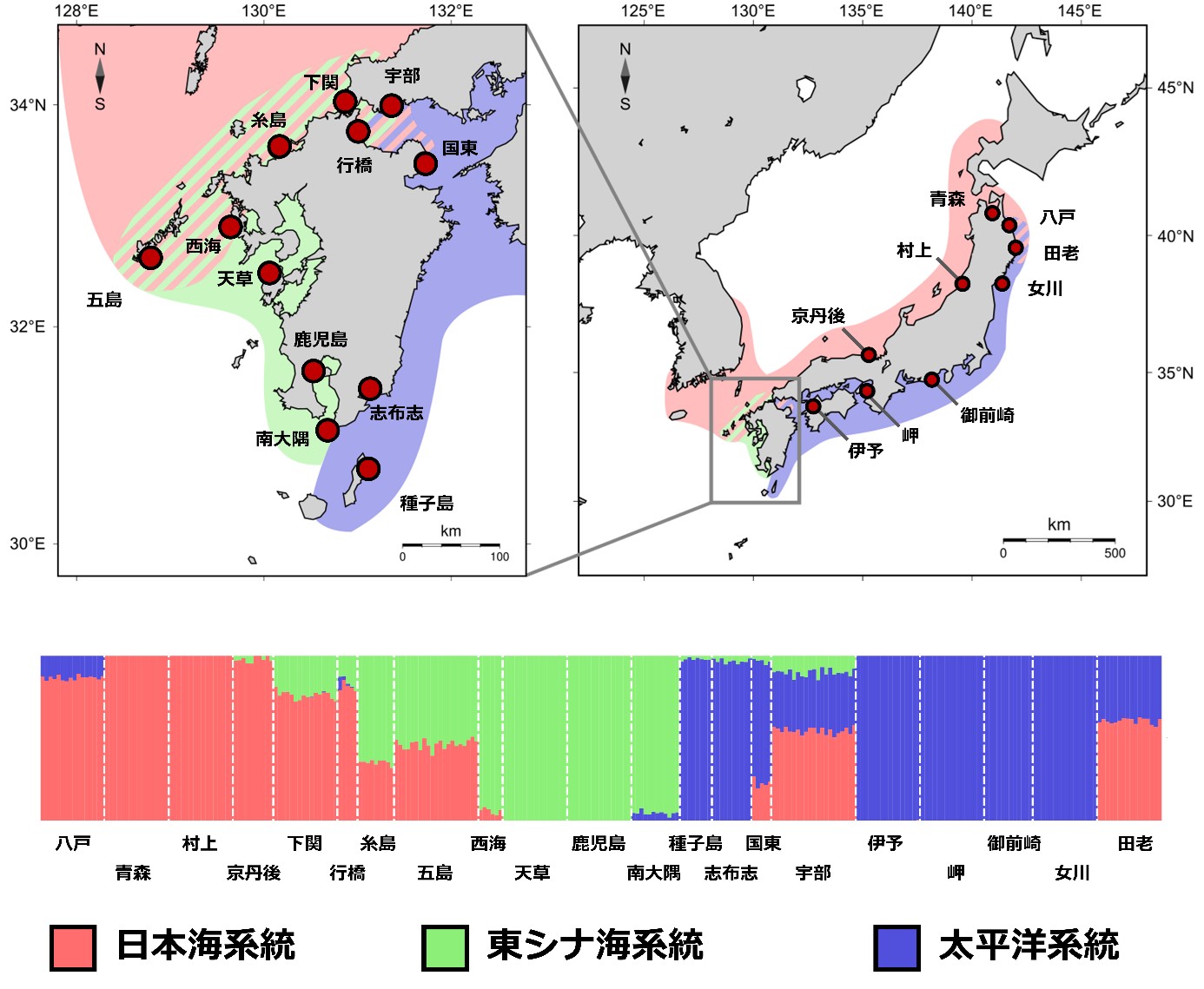
図1:本研究で推定されたアゴハゼの集団構造
上部の地図の沿岸域の色は、本研究で推定された各種内系統の分布域を示している。背景が縞々になっている地域は、系統間の交雑帯を示す。また、地図上の円は本研究で解析に用いた地域集団を示している。 下部の図はクラスタリング解析と呼ばれる集団構造解析の一種で、横に並ぶ一つ一つの縦の棒が各個体のゲノムの混ざり具合を示し、その色は各系統のゲノムを表している。本研究の解析で、アゴハゼの集団構造を高い精度で把握することができた。
次に、この結果を踏まえたうえで、デモグラフィックモデリング解析(注7)を行い、東シナ海系統の形成史を推測しました。この解析では、事前に定義した進化シナリオのモデルのうち、どれがもっともらしいかをシミュレーションにより調べることが出来ます。様々な進化シナリオを検証した結果、「太平洋系統から古くに分岐したゴースト系統が、日本海系統から分岐した系統にわずかに遺伝子浸透することで、東シナ海系統が誕生した」というシナリオが最も強く支持されました(図2)。
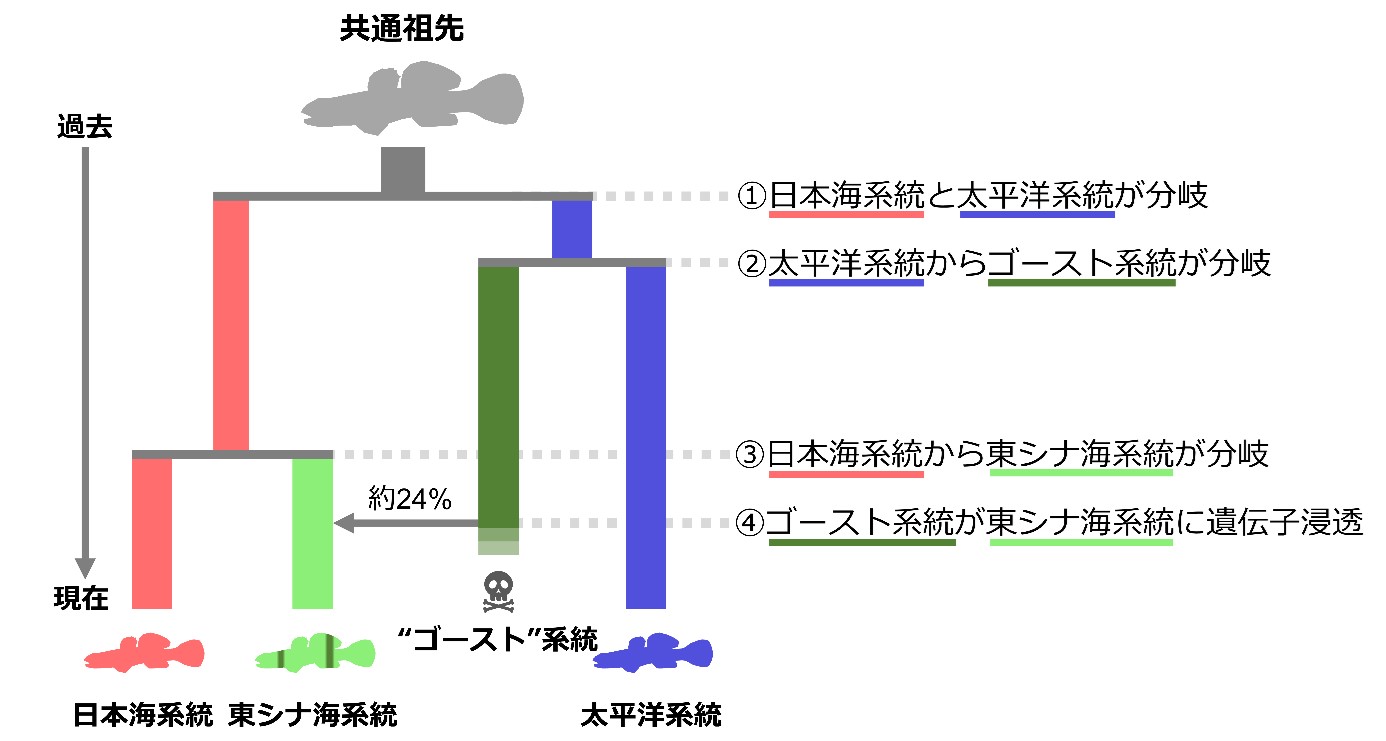
図2:デモグラフィックモデリング解析で推定されたアゴハゼ東シナ海系統の形成史
デモグラフィックモデリング解析で様々な進化シナリオをシミュレーションにより比較検証した結果、東シナ海系統は絶滅したゴースト系統との交雑に起因する系統であることが判明した。
東シナ海系統がゴースト系統との交雑に起源を持つことが示されたため、3系統の全ゲノムリシーケンス(注8)に基づくスライディングウィンドウ解析(注9)により、ゴースト系統由来の遺伝子浸透ランドスケープの特徴を調べました。ゴースト系統から遺伝子浸透した領域を調べることは容易ではありませんが、デモグラフィックモデリング解析の結果を参考にしたフォワードシミュレーション(注10)に基づく検証により、本研究ではこれが可能であることを示しました。遺伝子浸透ランドスケープの特徴を徹底的に調べた結果(図3)、ゴースト系統が遺伝子浸透できなかった領域は、遺伝子密度が高く、組み換え率が低いという特徴が判明しました。これは現存生物間の交雑ゲノムでも良く知られた特徴であり、機能的に重要な領域では絶滅系統のゲノムが排除されたことを示唆しています。一方で、ゴースト系統が遺伝子浸透できた領域には、リピート配列(注11)が多いことを発見しました。この特徴はこれまでの交雑ゲノム研究でもあまり知られていませんでしたが、先行研究のデータを再解析したところ、他の現存生物の交雑ゲノムでも同様の特徴が確認されました。これらの結果は、ゴースト系統のゲノム上における生存パターンは、現生生物の交雑ゲノムの特徴と良く類似していることを示しています。
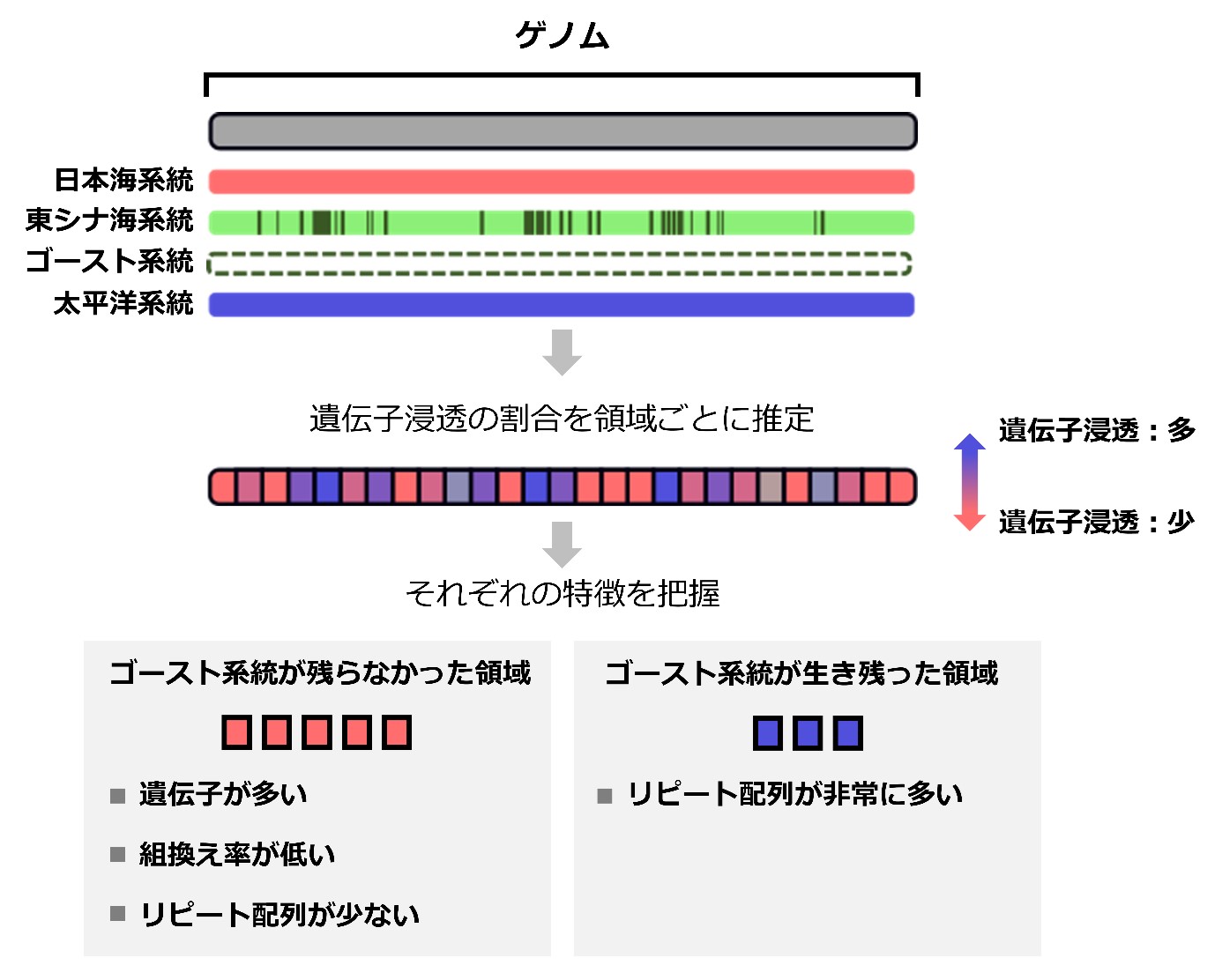
図3:本研究のスライディングウィンドウ解析の概要図
今後の展望
今回の研究では古代の交雑により誕生した東シナ海系統に着目しましたが、本種には最近の交雑により誕生した交雑帯も複数発見されています。こうした複数の交雑ゲノムの遺伝子浸透ランドスケープを同じ枠組みで比較することで、交雑ゲノムの形成過程における時空を超えた一般則を調べることができると考えています。
発表雑誌
- 雑誌
- Molecular Ecology
- 題名
- Genomic landscape of introgression from the ghost lineage in a gobiid fish uncovers the generality of forces shaping hybrid genomes
- 著者
- Shuya Kato*, Seiji Arakaki, Atsushi J. Nagano, Kiyoshi Kikuchi, Shotaro Hirase*
研究助成
本研究は、科研費「交雑起源の新規集団のゲノム進化に関する包括的研究(課題番号:18H02493)」、「急速な進化を逆利用して水産生物の性決定遺伝子をとらえる(課題番号:22H00377)」、「日本近海における異所的分化の遺伝的基盤:複数の交雑帯を基軸とした集団ゲノミクス(課題番号:22J12643)」の支援により実施されました。
発表者
東京大学 大学院農学生命科学研究科 附属水産実験所
加藤 柊也 博士課程(日本学術振興会特別研究員DC2)
平瀬 祥太朗 助教
菊池 潔 教授
九州大学 大学院理学研究院 理学部附属天草臨海実験所
新垣 誠司 准教授
龍谷大学 農学部 生命科学科
永野 惇 教授(兼:慶應義塾大学 先端生命科学研究所 特任教授)
用語解説
注1 ゴースト系統(ghost lineage)
現時点でサンプリングできておらず、DNAのような遺伝情報に基づく証拠が存在しない系統のこと。手でつかむことができない正体不明な存在であることを「幽霊」に例えた呼称である。絶滅系統のうちDNAが取得できるのはごく一部であるため、ほとんどの絶滅系統はゴースト系統であるといえる。
注2 集団ゲノミクス(population genomics)
生物の複数個体のゲノムを分析することで、集団の進化の歴史やメカニズムを解明しようとする学問のこと。
注3 遺伝子浸透(introgression)
交雑や戻し交雑の繰り返しによって、一方の系統のゲノムの一部や遺伝子がもう一方の系統の中に組み込まれる現象のこと。ゴースト系統からの遺伝子浸透のことを特に“ghost introgression”とも呼称する。
注4 ゲノミックランドスケープ(genomic landscape)
着目している配列や特徴についての、ゲノム上の配置や分布パターンの特性を表現する概念のこと。本研究では、ゲノム上のどの領域で遺伝子浸透が起きて、どの領域では遺伝子浸透が起きていないのかという「遺伝子浸透のゲノミックランドスケープ(genomic landscape of introgressionまたはintrogression landscape)」に着目している。
注5 分子系統地理学(Phylogeography)
近縁な種や集団の地理的な分布パターンや遺伝的な関係性を調べることで、その分布域形成の歴史を推定する学問のこと。
注6 RADシーケンス(Restriction Site Associated DNA sequencing)
ゲノム全体のうち、制限酵素認識サイト付近のゲノム領域のみを対象として、次世代シーケンサーにより配列を取得する手法のこと。ゲノムの様々な領域の多数の変異情報を比較的安価に取得することができるため、多数のサンプルを解析する集団構造解析などに適したシーケンス手法の一つである。
注7 デモグラフィックモデリング解析(demographic modeling analysis)
対象となる集団の遺伝的多様性のパターンに基づき、事前に定義した集団進化のモデルのうち、どれがもっともらしいかを合祖シミュレーションにより調べる解析のこと。集団の分岐年代や遺伝子浸透の強さなどのパラメータについて、実データの多様性を最もよく説明できる値も併せて推測することができる。
注8 全ゲノムリシーケンス(whole genome resequencing)
ゲノム全体を対象として、次世代シーケンサーにより配列を取得する手法のこと。既に基準となる全ゲノム参照配列が判明していることが前提となり、シーケンスのコストも高いが、ゲノム全体にわたる変異の情報を詳細に取得することができる。
注9 スライディングウィンドウ解析(sliding-window analysis)
ゲノム全体を“ウィンドウ”という小区分に区切り、ウィンドウごとに各種の特徴を観察する手法のこと。本研究ではゲノム全体を遺伝子浸透領域と非遺伝子浸透領域に二分することは困難であったが、ウィンドウを先に定義し、各ウィンドウにおける遺伝子浸透の強さを評価することで、遺伝子浸透ランドスケープを評価することができた。
注10 フォワードシミュレーション(forward simulation)
初期条件と進化のルールを定めたうえで、その後の進化をシミュレートする手法のこと。時間軸に沿ってシミュレート個体すべての進化を計算していくため計算コストが高いが、実データに依存しない遺伝的データをシミュレートすることができるため、解析手法の評価を行ううえで有用な手法である。なお、前述のデモグラフィックモデリング解析で用いられている合祖シミュレーションは、実データのサンプル間の共通祖先を推測するために時間軸を遡っていくシミュレーション手法であるため、フォワードシミュレーションとはアプローチや目的が大きく異なっている。
注11 リピート配列(repetitive elements)
生物のゲノム中に複数コピー存在する核酸配列のパターンや、その配列自体のこと。反復配列とも言う。同じ配列が繰り返し連続した「タンデムリピート」と、ゲノム中に散在する「散在反復配列」に大きく二分される。リピート配列は生物のゲノムのかなりの割合を占めているが、その機能は不明なものも多い。
問い合わせ先
<本研究に関するお問い合わせ>
東京大学大学院農学生命科学研究科
助教 平瀬 祥太朗(ひらせ しょうたろう)
<広報に関するお問い合わせ>
東京大学大学院農学生命科学研究科・農学部
事務部 総務課総務チーム 総務・広報情報担当(広報情報担当)