2024-12-16 理化学研究所,国立国際医療研究センター,国立がん研究センター,近畿大学
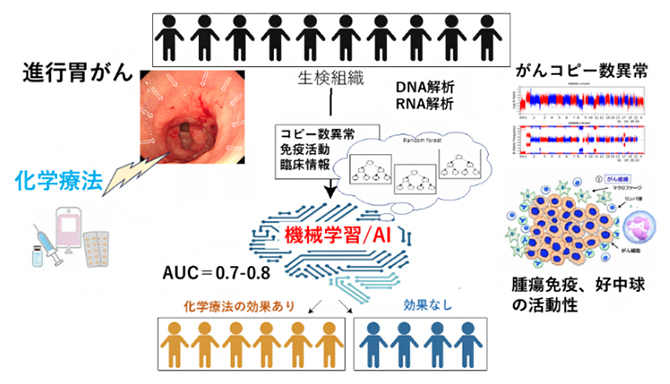
理化学研究所(理研)生命医科学研究センター がんゲノム研究チームの笹川 翔太 研究員、中川 英刀 チームリーダー、国立国際医療研究センターの山田 康秀 研究医療部長、国立がん研究センター中央病院 頭頸部・食道内科の本間 義崇 医長、近畿大学 医学部 免疫学教室の垣見 和宏 主任教授らの共同研究グループは、胃がん患者一人一人のゲノム変異およびRNA発現データから腫瘍内の免疫活動の特徴を解析し、人工知能(AI)[1]の一つの手法である機械学習[1]を用いて、それぞれの化学療法の効果を予測することに成功しました。
本研究成果は、事前にがん化学療法の効果を予測するがん精密医療および、新しいがん免疫療法の開発に貢献すると期待されます。
今回、共同研究グループは、化学療法の開始前に採取した65例の進行胃がん組織の全ゲノムシークエンス解析[2]およびRNAシークエンス解析[2]を行い、化学療法の効果との関連性を調べました。その結果、がん細胞のコピー数異常[3]や腫瘍内の好中球(TAN)[4]などの特徴が化学療法の効果と関連することが分かりました。また、TANについてシングル細胞RNA解析[5]も行い、腫瘍の成長や転移を抑制または促進する機能を持つ分画(構成成分)があることが分かりました。さらに、これら免疫ゲノム情報と臨床情報などの123項目の胃がんの特徴を用いて、機械学習によって化学療法の効果を予測するアルゴリズムを開発し、その高い予測精度を確認しました。
本研究は、科学雑誌『Gastric Cancer』オンライン版(12月2日付)に掲載されました。
免疫ゲノム情報とAIによる胃がん化学療法の効果予測
背景
日本およびアジアでは、胃がんは依然として重要な健康課題です。日本では胃がんの罹患(りかん)率は、医療技術の向上やヘリコバクター・ピロリ菌の除菌治療の普及によって減少しつつありますが、いまだに国内のがんの中で罹患率が高く、2020年では109,679例が胃がんと診断され、2022年の胃がんによる死亡者数は40,711人でした注1)。
胃がん治療には、病期や患者さんの体調に応じてさまざまな選択肢が用意されています。早期発見された場合、内視鏡による低侵襲治療が有効です。進行した胃がんに対しては、外科的切除が第一選択とされ、胃の一部または全摘出とリンパ節郭清(かくせい:切除)が行われます。また、手術後の再発予防や進行胃がんに対しては、薬物療法が治療の重要な柱となっています。
胃がんに対する薬物療法として、従来から使用されてきたプラチナ製剤(シスプラチンやオキサリプラチン)やフッ化ピリミジン系薬剤(S-1やカペシタビン)による化学療法に加えて、新たな分子標的薬や、免疫チェックポイント阻害剤(PD-1阻害薬)による免疫療法も導入されています。これにより、患者さんの特性に応じた個別化医療が進み、治療効果の向上が期待されています。特に進行胃がんの治療では、分子標的薬であるトラスツズマブが利用されており、トラスツズマブはがん細胞の増殖に関係するタンパク質HER2が陽性(HER2遺伝子が高発現またはコピー数増加)の胃がん患者に対して効果が期待できます。また、PD-1阻害薬であるニボルマブ(オプジーボ)やペムブロリズマブ(キイトルーダ)といった免疫チェックポイント阻害剤[6]も、化学療法後の治療としての適応が拡大され、胃がんの薬物療法の新たな選択肢として期待を持たれています。術前や術後の補助化学療法も進歩しており、術後の再発リスクを抑えるためにプラチナ製剤とフッ化ピリミジン系薬剤の併用療法が標準治療とされています。
そこで本研究では、化学療法を受けた進行胃がんを対象として、ゲノム変異情報や、遺伝子発現から推測した免疫細胞の働きから、化学療法の効果を予測するアルゴリズムの開発を試みました。同様の研究手法は、以前に食道がんについて理研で行っています注2)。
注1)国立がん研究センターがん情報サービス:がん種別統計情報[国立がん研究センター がん統計]
注2)2022年8月9日のプレスリリース「ゲノムとAIにより食道がんの術前化学療法の効果を予測」
研究手法と成果
共同研究グループは、国立がん研究センター中央病院において、プラチナ製剤とフッ化ピリミジン系薬剤の組み合わせを主とした化学療法の開始前に、内視鏡にて採取した65例の進行胃がん組織よりDNAとRNAを抽出しました。そして、網羅的な全ゲノムシークエンス解析およびRNAシークエンス解析を行い、がん細胞やがん組織の遺伝子変異や遺伝子発現に関する情報を取得し、これらのデータと実際の化学療法の効果との関連を調べました。
胃がんのdriver遺伝子変異(発がんに関わる遺伝子変異)、薬剤代謝酵素の多型、MSI状態[7]、EBVやピロリ菌感染[7]をDNA情報より同定しましたが、実際の化学療法の効果とは関連が認められませんでした。一方、全ゲノム解析の結果、がん細胞のコピー数異常における特定のパターンとの関連が認められました。
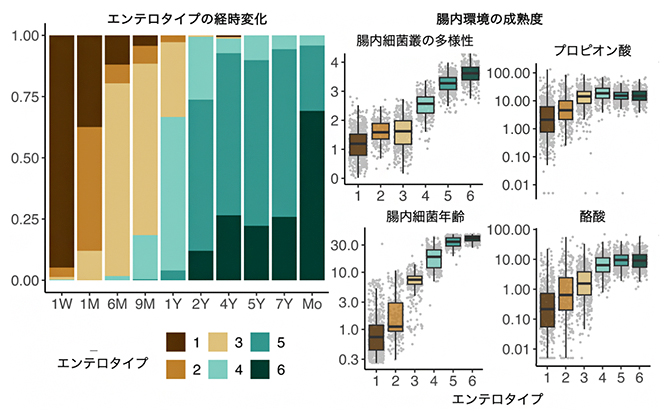
また、65例の胃がん組織のRNA発現解析の結果、492個の遺伝子発現が化学療法の効果と関連していることが分かりました。そこで、遺伝子発現の傾向を調べるためにGSEA[8]という解析を行ったところ、炎症関連のIFN-γやIL-6などのさまざまな遺伝子群の活性、つまりパスウェイ(遺伝子間の連続的な機能的連関)活性が、化学療法の効果が高い群で上昇していました。そして、がん細胞に対する免疫細胞の働きを遺伝子発現の程度から推測し、胃がん組織に浸潤している免疫細胞に着目することで、TAN、B細胞[9]、CD8+T細胞[9]、CD4+T細胞[9]がそれぞれ多く発現する四つの群に分類できました(図1上)。注目すべきことに、TANの多い群では他の群に比べて化学療法の効果が高く、TANの活動が化学療法の効果と機能的に関連していると考えられました(図1下右)。さらに興味深いことに、これは以前に報告した食道がんのTANの化学療法との相関とは逆の結果でした。
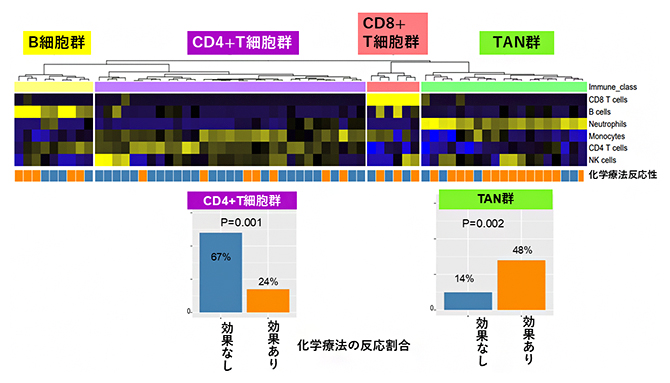
図1 胃がんの免疫分類
(上)胃がん組織のRNA発現データから免疫細胞の発現データを抽出して、四つの分類群(B細胞が多い群、CD4+T細胞が多い群、CD8+T細胞が多い群、TANが多い群)を作成した。各細胞の色は黄色いほど相対的高発現、青いほど相対的低発現であることを示す。
(下)TAN群は、化学療法の反応性が高く(オレンジ)、CD4+T細胞群では、化学反応の反応性が低い(青)傾向にあった。
次に、123項目のゲノム変異情報、コピー数異常、TANなどの腫瘍内の免疫情報、臨床情報を用いて、機械学習によって化学療法の効果を予測するアルゴリズムを開発しました。内視鏡で組織を採取した65例のうち、45例(70%)のデータを学習セットに用いたところ、化学療法の効果に最も寄与する因子は、TANの活動とがん細胞のコピー数パターンであることが分かりました。また、開発したアルゴリズムの予測精度を調べるために、残りの20例(30%)、および、中国にて行われた35例の臨床試験の公開データについても同様の解析を行いました。その結果、診断法の有用性を示すAUC[10]=0.7~0.8(1に近いほど精度が高い)の値が得られ、高い予測精度の再現性が確認できました(図2)。
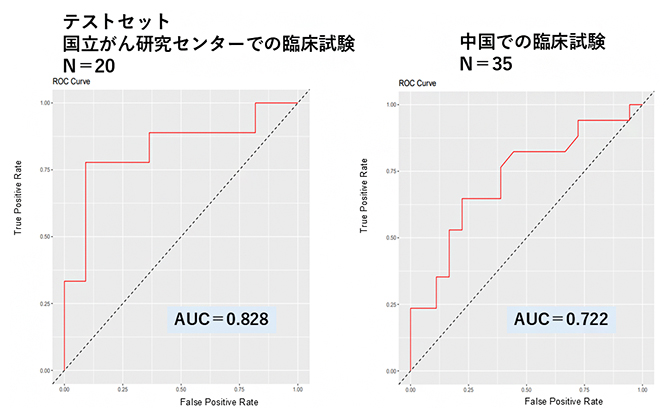
図2 機械学習による化学療法効果予測の予測精度
(左)本研究で用いた胃がん症例(20例)でのAUCカーブ。(右)中国で行われた臨床試験症例(35例)でのAUCカーブ。
最後に、化学療法の効果と最も高い関連性が見られた腫瘍内に浸潤するTANに着目し、まず、胃がんと食道がん組織に浸潤している免疫細胞を抽出しました。これらのシングル細胞解析を行い、59,584個の免疫細胞のRNA発現データから3,822個のTANのRNA発現データを抽出し、その特徴を調べました。これらTANを12個に分類することができ、そのうち、二つの分画は腫瘍の成長や転移を抑制して抗腫瘍的に働き、三つの分画は腫瘍の成長や転移を促進して親腫瘍的に働いていることが分かりました。また、これら抗腫瘍的または親腫瘍的に働いているTANは、化学療法の反応性と胃がん患者の予後と関連していることも証明しました(図3)。
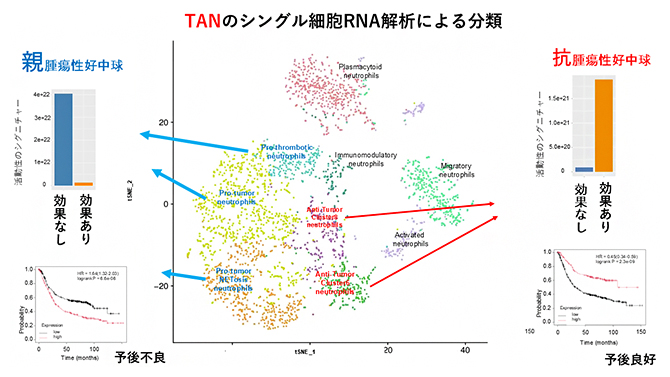
図3 TANのシングル細胞RNA解析
3,822個のTANのRNA発現プロファイルから、12個の分画が構成でき、三つの分画(青矢印)は親腫瘍的に機能しており、胃がんの化学療法の不応性(左上図の効果なし)と予後不良(左下図の予後曲線の赤線)と関連していた(左)。一方、二つのTANの分画(赤矢印)は、抗腫瘍的に働いており、胃がんの化学療法の良好な反応性(右上図の効果あり)と予後良好(右下図の予後曲線の赤線)と関連が認められた(右)。
今後の期待
本研究により、胃がん組織のゲノム情報、免疫情報などのさまざまな情報を組み合わせることで、胃がんの化学療法の効果予測が可能であることが証明されました。この研究手法は、胃がんのみならず他の腫瘍の化学療法の効果予測や精密医療にも応用できる可能性があります。
また、化学療法の効果に、腫瘍免疫、特にTANが密接に関連することが分かりました。胃がんに対する免疫細胞の活動についてさらに詳細な研究を進めることで、化学療法と免疫チェックポイント阻害剤の併用療法、または新しい複合免疫治療の開発が期待できます。
補足説明
1.人工知能(AI)、機械学習
人工知能とは、機械に人間と同様の知能を実現させようとする取り組みやその技術を指す。機械学習は人工知能に内包されるもので、機械(コンピュータ)が膨大なデータを学習し、データの背景にあるルールやパターンを発見する手法。今回は、ランダムフォレストというディシジョンツリー(決定木)に基づいたアルゴリズムを用いており、重複を許すランダムサンプリングによって多数のディシジョンツリーを作成し、各ツリーの予測結果の多数決を採ることで最終予測値を決定する。AIはArtificial Intelligenceの略。
2.全ゲノムシークエンス解析、RNAシークエンス解析
個人やがん細胞の全ゲノム情報を解読し、塩基配列の違いや変化を同定することを全ゲノムシークエンス解析という。がん細胞の場合は、がんのDNAと同一患者由来の正常DNAの全ゲノムシークエンス解析を行い、その差分を調べる。RNAも、cDNA(相補的DNA)に変換した後に、RNAのほぼ全長にわたってシークエンス解析ができ、このRNAシークエンス解析によって、遺伝子の発現の有無などを調べることができる。
3.コピー数異常
正常な体細胞には同じ染色体が二つ(2コピー)あるが、がん細胞では、染色体が部分的にまたは染色体そのものが欠失したり、2コピー以上に増幅したりする。これをコピー数異常と呼ぶ。欠失した領域にはがん抑制遺伝子が、増幅した領域にはがん遺伝子が存在すると考えられている。コピー数異常のある領域の大きさや、コピー数異常の構造の特徴などの情報を組み合わせ、それをパターン化したものをコピー数シグニチャーという。DNAへのダメージやその修復機構の有無、細胞周期異常と関連する10~20個のコピー数シグニチャーが報告されている。
4.腫瘍内の好中球(TAN)
通常の好中球は、血液中を流れており、強い貪食(どんしょく)能力を持ち、病原体を貪食する。さらには顆粒やサイトカインの放出によって感染部位へ他の免疫細胞を動員し、免疫応答を促進する。好中球のうち腫瘍組織内で活動するものはTANと呼ばれ、腫瘍の成長や転移を助けることがある親腫瘍的なものと、逆に抗腫瘍効果を示すものの2種類に分類されることがある。親腫瘍的なものは、腫瘍の成長や転移を助ける分子(例えばプロテアーゼや血管新生因子)を分泌し、腫瘍細胞が周囲の組織に侵入するのを助け、また、免疫抑制的な環境を作り出すことで、他の免疫細胞が腫瘍を攻撃するのを防ぐ。抗腫瘍効果とは、逆に、TANが腫瘍細胞の増殖を抑制するサイトカインを放出し、他の免疫細胞と協力して腫瘍細胞の破壊を促進するものと考えられている。TANはTumor-Associated Neutrophilの略。
5.シングル細胞RNA解析
単一の細胞をフローサイトメトリーやマイクロ流体デバイスを用いて単離し、個々の細胞のRNAを解析する技術。1細胞ごとに遺伝子発現を計測する方法であり、この技術は、細胞集団内での異なる遺伝子発現のプロファイルを解析することで、同じ組織内で異なる細胞の種類や状態を明らかにするために使われる。
6.免疫チェックポイント阻害剤
免疫のチェックポイントであるPD-1分子を抗体で抑制することで、腫瘍細胞への免疫抑制を解除し、免疫細胞が腫瘍細胞を排除できるように導く薬剤。
7.MSI状態、EBVやピロリ菌感染
胃がんは分子分類において、MSI(MicroSatellite Instability)、EBV(Epstein-Barr Virus)感染、CIN群(Chromosome Instability)およびCS群(Chromosome Stability)の群に分けられる。MSIは、DNA修復機構が破綻した腫瘍で、変異が10倍集積する。EBVは胃がんや咽頭がん組織で観察され、発がんと関連する。ピロリ菌感染は、胃潰瘍や胃炎を引き起こし萎縮性胃炎になることから、胃の発がんと強く関連している。
8.GSEA
GSEAは、2群間で遺伝子発現が異なる遺伝子が特定の遺伝子セット(IFN-γに関わる遺伝子セットなど)に偏っているかどうかを調べる解析法。GSEAはGene Set Enrichment Analysisの略。
9.B細胞、CD8+T細胞、CD4+T細胞
免疫に関わる白血球は、B細胞、T細胞、マクロファージ、好中球、好酸球、好塩基球などに分類される。B細胞は、異物に対する抗体を産生する。T細胞のうち、CD8+T細胞は腫瘍や感染細胞に直接的に作用する(細胞障害性T細胞)。CD4+T細胞はマクロファージなどから抗原提示を受けると増殖、活性化して、炎症を促すサイトカインを産生し、他の免疫細胞を活性化させる(ヘルパーT細胞)。
10.AUC
診断法がどのくらい有用なのかを解析する統計学的な手法であるROC曲線(Receiver Operatorating Characteristic curve)を作成したときに、グラフの曲線より下の部分の面積をAUCという。AUCは0から1までの値を取り、値が1に近いほど判別能が高いことを示す。AUCはArea Under the Curveの略。
研究支援
本研究は、理化学研究所交付金、国立国際医療研究センター(NCGM)国際医療研究開発費による助成を受けて行われました。
原論文情報
Shota Sasagawa, Yoshitaka Honma, Xinxin Peng, Kazuhiro Maejima, Koji Nagaoka, Yukari Kobayashi, Ayako Oosawa, Todd A. Johnson, Yuki Okawa, Han Liang, Kazuhiro Kakimi, Yasuhide Yamada, and Hidewaki Nakagawa, “Predicting Chemotherapy Responsiveness in Gastric Cancer through Machine Learning Analysis of Genome, Immune, and Neutrophil Signatures”, Gastric Cancer, 10.1007/s10120-024-01569-4
発表者
理化学研究所
生命医科学研究センター がんゲノム研究チーム
研究員 笹川 翔太(ササガワ・ショウタ)
チームリーダー 中川 英刀(ナカガワ・ヒデワキ)
国立国際医療研究センター
研究医療部長 山田 康秀(ヤマダ・ヤスヒデ)
国立がん研究センター中央病院
頭頸部・食道内科 医長 本間 義崇(ホンマ・ヨシタカ)
近畿大学 医学部 免疫学教室
主任教授 垣見 和宏(カキミ・カズヒロ)
報道担当
理化学研究所 広報室 報道担当
国立国際医療研究センター 企画戦略局 広報企画室
国立がん研究センター 企画戦略局 広報企画室
近畿大学医学部・病院運営本部 総務広報課