2025-02-05 国立遺伝学研究所
日本の春を彩るサクラの中で、ひときわ存在感を放つオオシマザクラ。その完全なゲノム配列を国立遺伝学研究所(遺伝研)と森林総合研究所(森林総研)を中心とする「サクラ100ゲノムコンソーシアム」が解読に成功しました。研究チームは、伊豆大島にある樹齢800年以上といわれる国の特別天然記念物「大島のサクラ株」をサンプルとして使用し、高度なゲノム解析技術によりオオシマザクラの全染色体構造を明らかにしました。
オオシマザクラは、日本のサクラ品種の多様化に重要な役割を果たしてきました。特に注目すべきは、日本中で親しまれている‘染井吉野’の親の一つであるという点です。1960年代に遺伝研の竹中要博士が、染井吉野がオオシマザクラとエドヒガンの雑種であることを実験データに基づき示しましたが、今回の研究はその知見をさらに深化させるものです。オオシマザクラのゲノム解析は、単にこの種の理解を深めるだけではなく、今後はオオシマザクラが関与したと考えられる多くのサクラ栽培品種の由来や花の色、開花時期、形態などに関わる遺伝子の解明に活用できると期待されます。また、この「大島のサクラ株」は接ぎ木により増殖して遺伝研構内に植樹を行いました。
本研究を遂行した「サクラ100ゲノムコンソーシアム」は、遺伝研の藤原一道特任研究員、豊田敦特任教授、川本祥子准教授、佐藤豊教授、小出剛准教授、及び、森林総研の勝木俊雄博士をはじめ、サクラゲノム研究を目的として遺伝研や森林総研などの研究者が集まり作られました。
本研究は、情報・システム研究機構戦略的研究プロジェクトにより支援されました。
本研究は、2025年2月4日に「Scientific Data」にオープンアクセスとしてオンライン出版されました。
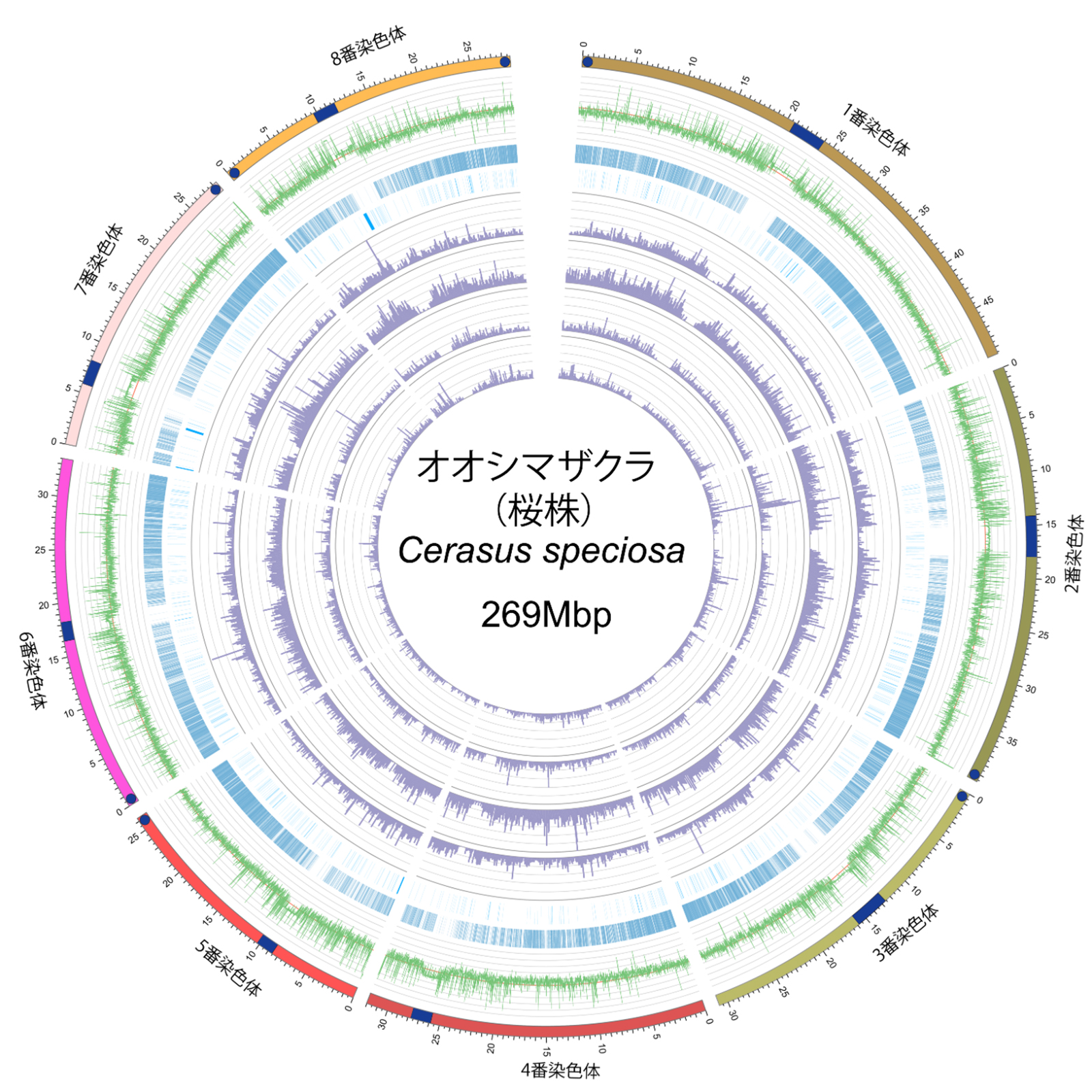 図: オオシマザクラ(桜株)の完全ゲノムの模式図。8本の染色体すべてについて、染色体の構造が明らかになった。外側のサークルは各染色体をあらわし、青いボックスはセントロメア、青い丸はテロメアを示している。多重の線は以下のゲノム配列を示している(外側から2番目:GC塩基の比率、3番目:遺伝子分布、4番目:ノンコーディングRNA遺伝子、5番目:LTR配列、6番目:TIR配列、7番目:LINEおよびSINE配列、8番目:Helitron配列。
図: オオシマザクラ(桜株)の完全ゲノムの模式図。8本の染色体すべてについて、染色体の構造が明らかになった。外側のサークルは各染色体をあらわし、青いボックスはセントロメア、青い丸はテロメアを示している。多重の線は以下のゲノム配列を示している(外側から2番目:GC塩基の比率、3番目:遺伝子分布、4番目:ノンコーディングRNA遺伝子、5番目:LTR配列、6番目:TIR配列、7番目:LINEおよびSINE配列、8番目:Helitron配列。
 写真提供:伊豆大島ジオパーク推進委員会
写真提供:伊豆大島ジオパーク推進委員会
写真: 「大島のサクラ株(さくらっかぶ)」 伊豆大島に自生するオオシマザクラの巨木。樹齢は800年以上と推定され、1952年に国の特別天然記念物に指定される。折れた主幹から大枝が横に張り出している。
A Near Complete Genome Assembly of the Oshima Cherry Cerasus speciosa
Kazumichi Fujiwara, Atsushi Toyoda, Bhim B. Biswa, Takushi Kishida, Momi Tsuruta, Yasukazu Nakamura, Noriko Kimura, Shoko Kawamoto, Yutaka Sato, Toshio Katsuki, Sakura 100 Genome Consortium, and Tsuyoshi Koide*
Sakura 100 Genome Consortium (full list): Kazumichi Fujiwara, Atsushi Toyoda, Bhim B. Biswa, Takushi Kishida, Momi Tsuruta, Yasukazu Nakamura, Noriko Kimura, Shoko Kawamoto, Yutaka Sato, Toshio Katsuki, Tsuyoshi Koide*, Akatsuki Kimura, Ken-Ichi Nonomura, Hironori Niki, Hiroyuki Yano, Kinji Umehara, Tazro Ohta, Chikahiko Suzuki.
*責任著者
Scientific Data volume 12, Article number: 162 (2025) DOI:10.1038/s41597-025-04388-z