2022-10-03 東京大学
発表概要
東京大学大学院新領域創成科学研究科の鎌谷洋一郎教授、小井土大助教、賀云雲大学院生、史明陽大学院生、東京大学医科学研究所バイオバンク・ジャパンの森崎隆幸客員教授、岩手医科大学の清水厚志教授、八谷剛史客員教授、大阪大学の岡田随象教授、難波真一大学院生、九州大学の二宮利治教授、秦淳准教授らの国際共同研究グループは、GIGASTROKEコンソーシアム(注1)の一員として、世界中から集めた250万人規模の遺伝情報を用いて史上最大規模の脳卒中のゲノムワイド関連解析(Genome-Wide Association Study:GWAS、注2)を行いました。5つの異なった祖先集団の20万人以上の脳卒中患者と200万人以上の対照者による大規模国際共同研究により、脳卒中と各病型に関連するシグナルを89の独立した遺伝的座位において発見し、そのうち61座位は新規に発見するものでした。
この研究は、脳卒中及び各病型の病態に関わる将来の生物学的研究に洞察を与えるものです。この研究により原因遺伝子が推定され、またF11、KLKB1、PROC、GP1BA、LAMC2とVCAM1を、脳卒中の予防と治療のための潜在的な創薬ターゲットとして同定しました。
さらに研究グループは、脳卒中の遺伝的予測ツールを開発しました。これは非ヨーロッパ系祖先集団での有効性が増強し妥当性を確認された最初のもので、精密な予防と精密な薬剤開発の進展が期待されます。
本研究成果は、2022年9月30日付で国際的な総合科学誌「Nature」に掲載されました。
この研究は、250万人以上の参加者のDNA検体を用いて行われ、そのうち20万人が脳卒中患者です。研究参加者はヨーロッパ、東アジア、アフリカ、南アジア、そしてラテンアメリカの各地域で収集されました(1/3の参加者は非ヨーロッパ系)。これらは多数の病院コホート、地域集団コホート、バイオバンク、そして5つの臨床研究からなります。
この研究は、ISGCやCHARGEなど複数の国際的コンソーシアムやネットワーク、そして20カ国以上の研究者からなるGIGASTROKEコンソーシアムのメンバーによって実施されました。総責任研究施設はボルドー大学(フランス)とルードヴィヒ・マクシミリアン大学(LMU)ミュンヘン(ドイツ)で、東京大学(日本)、タルトゥ大学(エストニア)、イバダン大学(ナイジェリア)、VAボストンヘルスケアシステムとハーバード医学校(USA)との共同で進められました。
研究内容
脳卒中は世界中の死因の第2位で、全死亡の約12%を占めており、早死損失年数や障害共存年数の主要な要因です。脳卒中の発生率と重症度は、全脳卒中死の70%を占める低・中所得国でとりわけ高いことが報告されています。したがって、脳卒中の予防と治療の改善を目的とした研究においては、グローバルな視点を取り入れることが極めて重要です。
今回、研究グループは、GIGASTROKEコンソーシアムの一員として、世界中から集めた250万人規模の遺伝情報を用いて史上最大規模の脳卒中のゲノムワイド関連解析(GWAS)を行いました。
第一段階の遺伝的研究は160万人のデータを用いて行われ、そのうち110,182人は脳卒中患者でした。脳卒中と関連する89領域(61は新規)を同定しました(図1)。これらのゲノム領域については引き続き多くはバイオバンクで収集された110万人で検討され、そのうち89,084人は脳卒中患者でしたが、第一段階の解析で検出されたうちのほとんどの関連について追加の証拠を得ました。
 図1. ゲノムワイド有意に関連する89座位のイデオグラム。円は集団横断的(CROSS-ANC)、ダイアモンドはヨーロッパ系集団(EUR)、三角形は東アジア集団(EAS)、四角はアフリカ系アメリカ人と南アジア人集団(AFR/SAS)でそれぞれ検出されたことを示す。緑色は全脳卒中(AS)、赤色は全脳梗塞(AIS)、薄い青はラクナ梗塞(SVS)、濃い青は心原性脳塞栓(CES)、紫色はアテローム血栓性脳梗塞(LAS)で検出されたことを示す。太字でアステリスクがついているのは高信頼度、太字でアステリスクがないものは中等度の信頼度、細字は低信頼度である(詳細は原論文を参照いただきたい)。イタリックは二次的解析であるMR-MEGAあるいはMTAG解析で検出された座位である。
図1. ゲノムワイド有意に関連する89座位のイデオグラム。円は集団横断的(CROSS-ANC)、ダイアモンドはヨーロッパ系集団(EUR)、三角形は東アジア集団(EAS)、四角はアフリカ系アメリカ人と南アジア人集団(AFR/SAS)でそれぞれ検出されたことを示す。緑色は全脳卒中(AS)、赤色は全脳梗塞(AIS)、薄い青はラクナ梗塞(SVS)、濃い青は心原性脳塞栓(CES)、紫色はアテローム血栓性脳梗塞(LAS)で検出されたことを示す。太字でアステリスクがついているのは高信頼度、太字でアステリスクがないものは中等度の信頼度、細字は低信頼度である(詳細は原論文を参照いただきたい)。イタリックは二次的解析であるMR-MEGAあるいはMTAG解析で検出された座位である。
共同研究グループは異なった祖先集団グループに渡って脳卒中に関わる多くの遺伝的感受性が共有されており、同定されたゲノム領域の効果は祖先集団の間で非常に似ていることを明らかにしました(図2)。
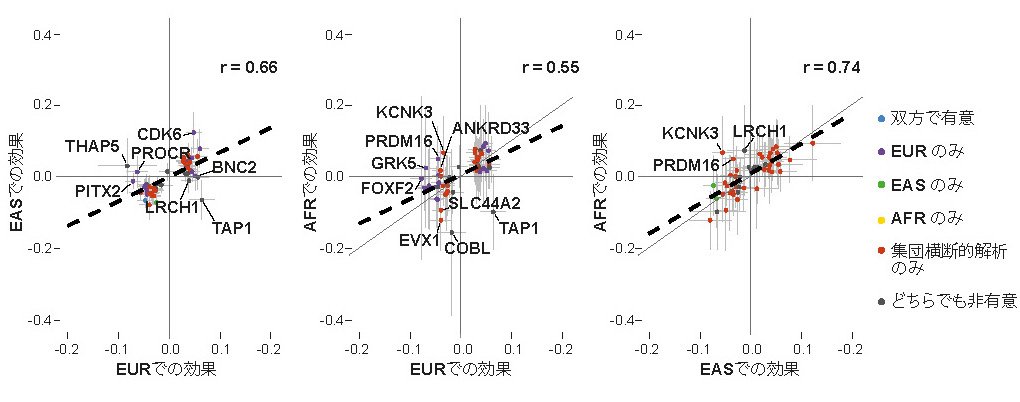
図2. 全脳卒中と関連する60の遺伝的アレルについて、祖先集団の間のピアソンの相関係数を求めた。集団間で効果量が異なる(0.05以上異なる)アレルの場合は、近傍の遺伝子名を表示した。点は効果量の大きさを示し、バーは95%信頼区間である。相関係数がゼロではないことを帰無仮説とした両側検定P値を示している。点の色については凡例を参照、また集団名の意味は図1の説明を参照いただきたい。
脳卒中は、突然発症する神経障害を特徴とし、その主な原因は脳梗塞(動脈に凝固が生じ、主な病型はアテローム、心疾患、小血管疾患と関連する)で、また頻度は低いものの脳の中で出血が起こる脳出血があります。本研究で観察された関連の多くは、脳卒中全体または脳梗塞全体との関連でしたが、一部は脳梗塞のどれかの病型に特異的でした。
共同研究グループは、この結果を、異なる組織や脳細胞の遺伝子発現、血液、脳脊髄液、脳内のタンパク質レベルに関する既存のデータと組み合わせることで、脳卒中発症に関与する特定の遺伝子やその生物学的メカニズムに関する予備的知見を得ることに成功しました。
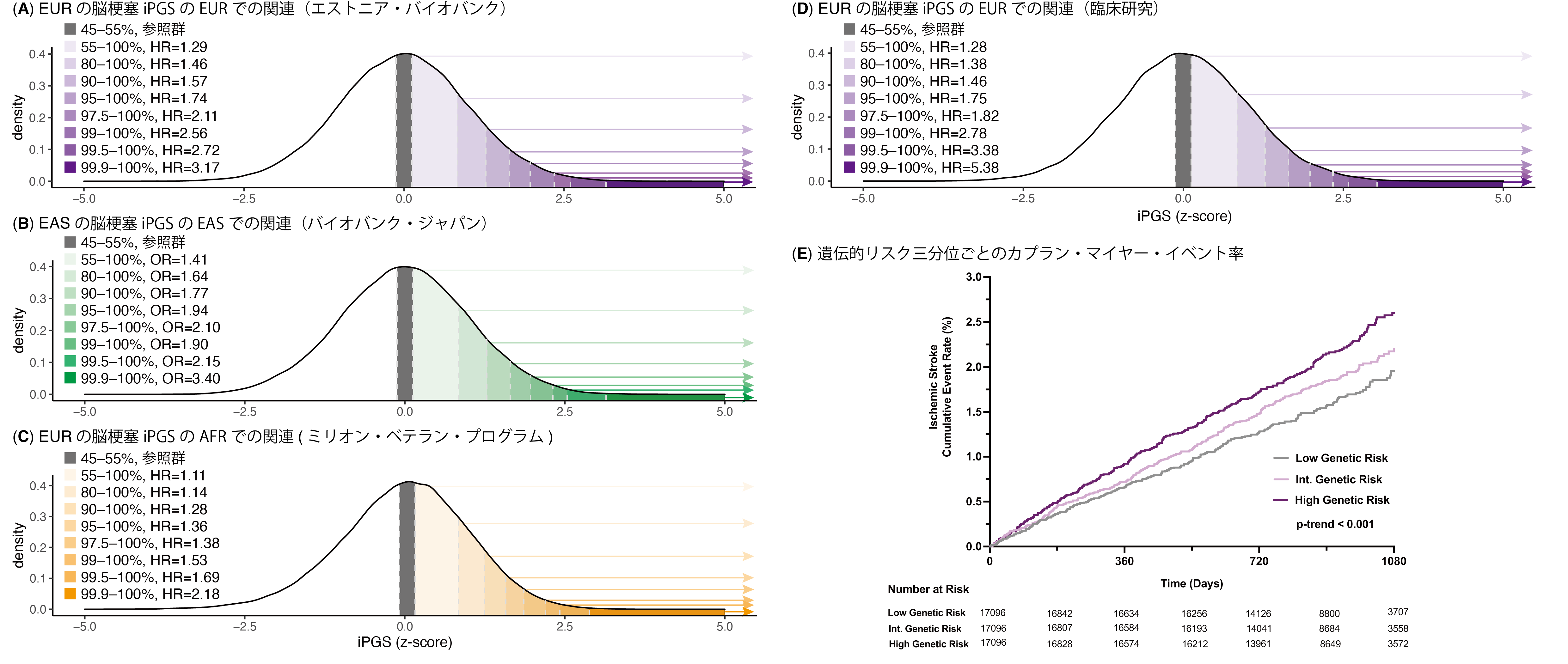
この研究で得られた全ゲノム関連解析結果をもとに数千の遺伝的バリアントの効果を評価する多遺伝子スコア(ポリジェニック・スコア)を作成したところ、そのスコアはヨーロッパ系集団の脳梗塞リスクと強く関連するだけでなく、東アジア人でも関連することを今回初めて明らかにしました。さらに、52,600人の患者を対象とした5つの臨床試験のデータを活用し、同定された脳卒中の遺伝的危険因子を組み合わせると、臨床的危険因子(高血圧、喫煙など)とは独立して脳梗塞を予測し、より少ない遺伝的データセットに基づくこれまでの研究よりもはるかに強く予測できることを示しました(図3)。
図3 (A-D)多遺伝子スコアiPGSのヨーロッパ系集団(A)、東アジア集団(B)、アフリカ系アメリカ人集団(C)とヨーロッパ系集団の臨床研究(D)。ヨーロッパ系集団とアフリカ系アメリカ人集団ではCox比例ハザードモデル、東アジア集団ではロジスティック回帰モデルで、年齢、性別、5つの主成分で調整した10分位点中位(45-55%)の集団を参照群とした時のリスク効果を示した。(E)ヨーロッパ系集団の5つの臨床試験を統合した脳梗塞のカプラン・マイヤー・イベント率を遺伝的リスクの三分位点をもとに3年間追跡した(遺伝的リスクは、集団横断的な全脳卒中GWASをもとにした)。遺伝的リスクが高いと脳梗塞リスクが増えることが観察されている。
現在我が国では、がんの体細胞変異や希少疾患における単一遺伝子変異を対象として実際の医療で役立てるゲノム医療の取り組みが準備段階に入っています。しかしながら、今回の研究対象であり、日本の第3位〜4位の死因を占める脳卒中を含む多くの疾患の診療でゲノム情報を役立てる道筋はまだ立っていません。本研究は、将来の脳卒中の予防や治療における多遺伝子スコアを利用したゲノム医療の構築につながる研究成果であると言えます。このため、アメリカで進んでいるような多遺伝子スコアの有効性を調べるための更なるゲノム研究が我が国でも必要です。
また、本研究はゲノム解析結果とトランスクリプトーム・プロテオームなどの多層オミックス情報(注3)を組み合わせて脳卒中の病態に関わる遺伝子を解明できることを改めて立証しました。これらの脳卒中関連遺伝子について実験することにより、病態解明や創薬に結びつくことが期待されます。
研究リーダーからのコメント
ステファニー・デベット(Stephanie Debette)博士(ボルドー大学疫学科教授、INSERM(同大学公衆衛生研究センター)、同大学病院神経内科医、同大学公衆衛生研究センター所長)
「祖先集団背景の多様性を持つ研究参加者の貢献が最も重要で、新規の遺伝的関連の検出能力を高め、その生物学的基盤に対する理解を深め、大陸を超えて多遺伝子型の脳卒中のリスク予測の転移性を向上することが可能になりました。」
マルティン・ディシュガンス(Martin Dichgans)博士(ルードヴィヒ・マクシミリアン大学(LMU)ミュンヘン神経内科教授、脳卒中・認知症研究所(ISD)所長)
「この研究は、大規模で公的に利用可能な遺伝子発現やタンパク質濃度のリソースと組み合わせたゲノミクスが、創薬に役立つ力を持つことを示しています。本研究で同定された創薬標的候補のうち2つについては、現在すでに脳卒中の一次予防あるいは二次予防を目的とした第2相臨床試験が行われており、このアプローチの有効性を裏付けています。」
鎌谷洋一郎教授(東京大学大学院新領域創成科学研究科メディカル情報生命専攻複雑形質ゲノム解析分野)
「この研究は、脳卒中の公平で精密な予防と薬剤開発のためのアプローチへの新たな一歩となります。」
アニキット・ミスラ(Aniket Mishra)博士(ボルドー大学、INSERM(同大学公衆衛生研究センター))※筆頭著者
「この結果は基礎研究から臨床へと橋を繋ぐトランスレーショナル・リサーチとして役立ち、脳卒中の新規創薬標的を開拓していくための将来の実験における優先順位をつけるための指針を提供するものです」と述べています。「この研究はさらに、すでに市販されて他の疾患に適応のある薬の、脳卒中の予防や治療へのリポジショニング(転用)の裏付けとなります。」
リリ・ミラニ(Lili Milani)博士(タルトゥ大学エストニアゲノムセンター所長)
「今回はアフリカ系集団に特異的な脳卒中の多遺伝子スコアを作成するには参加者が少なかったのですが、ヨーロッパ系集団のスコアが、弱いものの、アフリカ系アメリカ人とアフリカ大陸のアフリカ人の双方で有意に関連することが示されました。これは全く新しい研究です!」
マヨワ・オウォラビ(Mayowa O. Owolabi)博士(イバダン大学医学部ゲノム精密医療センター所長)
「重要なことは、ゲノム研究の参加者の多様性、特にアフリカのような最も参加者の少ない地域からの参加を大幅に増やすことが急務であることも明らかになったことです。これによってトランスレーショナル・ゲノミクス、精密医療、そして精密な公衆衛生の時代に健康格差が拡大するのを防ぐことになるでしょう。」
論文情報
発表雑誌:「Nature」
タイトル:Stroke genetics informs drug discovery and risk prediction across ancestries
著者:Aniket Mishra, Rainer Malik, Tsuyoshi Hachiya, Tuuli Jürgenson, Shinichi Namba, Daniel C. Posner, Frederick K. Kamanu, Masaru Koido, Quentin Le Grand, Mingyang Shi, Yunye He, Marios K. Georgakis, Ilana Caro, Kristi Krebs, Yi-Ching Liaw, Felix C. Vaura, Kuang Lin, Bendik Slagsvold Winsvold, Vinodh Srinivasasainagendra, Livia Parodi, Hee-Joon Bae, Ganesh Chauhan, Michael R. Chong, Liisa Tomppo, Rufus Akinyemi, Gennady V. Roshchupkin, Naomi Habib, Yon Ho Jee, Jesper Qvist Thomassen, Vida Abedi, Jara Cárcel-Márquez, Marianne Nygaard, Hampton L. Leonard, Chaojie Yang, Ekaterina Yonova-Doing, Maria J. Knol, Adam J. Lewis, Renae L . Judy, Tetsuro Ago, Philippe Amouyel, Nicole D Armstrong, Mark K. Bakker, Traci M. Bartz, David A. Bennett, Joshua C. Bis, Constance Bordes, Sigrid Børte, Anael Cain, Paul M. Ridker, Kelly Cho, Zhengming Chen, Carlos Cruchaga, John W. Cole, Phil L. de Jager, Rafael de Cid, Matthias Endres, Leslie E. Ferreira, Mirjam I. Geerlings, Natalie C. Gasca, Vilmundur Gudnason, Jun Hata, Jing He, Alicia K. Heath, Yuk-Lam Ho, Aki S. Havulinna, Jemma C. Hopewell, Hyacinth I. Hyacinth, Michael Inouye, Mina A. Jacob, Christina E. Jeon, Christina Jern, Masahiro Kamouchi, Keith L. Keene, Takanari Kitazono, Steven J. Kittner, Takahiro Konuma, Amit Kumar, Paul Lacaze, Lenore J. Launer, Keon-Joo Lee, Kaido Lepik, Jiang Li, Liming Li, Ani Manichaikul, Hugh S. Markus, Nicholas A. Marston, Thomas Meitinger, Braxton D. Mitchell, Felipe A. Montellano, Takayuki Morisaki, Thomas H. Mosley, Mike A. Nalls, Børge G. Nordestgaard, Martin J. O’Donnell, Yukinori Okada, N. Charlotte Onland-Moret, Bruce Ovbiagele, Annette Peters, Bruce M. Psaty, Stephen S. Rich, Jonathan Rosand, Marc S. Sabatine, Ralph L. Sacco, Danish Saleheen, Else Charlotte Sandset, Veikko Salomaa, Muralidharan Sargurupremraj, Makoto Sasaki, Claudia L. Satizabal, Carsten O. Schmidt, Atsushi Shimizu, Nicholas L. Smith, Kelly Sloane, Yoichi Sutoh, Yan V. Sun, Kozo Tanno, Steffen Tiedt, Turgut Tatlisumak, Nuria P. Torres-Aguila, Hemant K. Tiwari, David-Alexandre Trégouët, Stella Trompet, Anil Man Tuladhar, Anne Tybjærg-Hansen, Marion van Vugt, Riina Vibo, Shefali S. Verma, Kerri L. Wiggins, Patrik Wennberg, Daniel Woo, Peter WF Wilson, Huichun Xu, Qiong Yang, Kyungheon Yoon, the COMPASS Consortium*, the INVENT consortium*, the Dutch Parelsnoer initiative (PSI) Cerebrovascular Disease Study Group*, the Estonian Biobank*, the PRECISE4Q consortium*, the NINDS Stroke Genetics Network (SiGN)*, the MEGASTROKE Consortium*, the SIREN Consortium*, the China Kadoorie Biobank Collaborative Group*, the VA Million Veteran Program*, the International Stroke Genetics Consortium (ISGC)*, the Biobank Japan*, the CHARGE Consortium*, the GIGASTROKE Consortium*, Iona Y. Millwood, Christian Gieger, Toshiharu Ninomiya, Hans J. Grabe, J Wouter Jukema, Ina L. Rissanen, Daniel Strbian, Young Jin Kim, Pei-Hsin Chen, Ernst Mayerhofer, Joanna MM Howson, Marguerite R. Irvin, Hieab Adams, Sylvia Wassertheil-Smoller, Kaare Christensen, Mohammad A. Ikram, Tatjana Rundek, Bradford B. Worrall, G. Mark Lathrop, Moeen Riaz, Eleanor M. Simonsick, Janika Kõrv, Paulo H.C. França, Ramin Zand, Kameshwar Prasad, Ruth Frikke-Schmidt, Frank-Erik de Leeuw, Thomas Liman , Karl Georg Haeusler, Ynte M. Ruigrok, Peter Ulrich Heuschmann, W T. Longstreth, Keum Ji Jung, Lisa Bastarache, Guillaume Paré, Scott Damrauer, Daniel I. Chasman, Jerome I. Rotter, Christopher D. Anderson, John-Anker Zwart, Teemu J. Niiranen, Myriam Fornage, Yung-Po Liaw, Sudha Seshadri, Israel Fernández-Cadenas, Robin G. Walters, Christian T. Ruff, Mayowa O. Owolabi, Jennifer E. Huffman, Lili Milani, Yoichiro Kamatani, Martin Dichgans, and Stephanie Debette.
* Consortium
DOI:10.1038/s41586-022-05165-3
用語解説
(注1)GIGASTROKコンソーシアム:
ISGC(https://www.strokegenetics.org/)は2007年4月に米国ボストンで組織され、脳卒中のゲノム解析の国際連携を目的としたコンソーシアムである。これまですでに脳卒中の遺伝因子を解明するMETASTROKE、MEGASTROKEコンソーシアムが結成されて結果を報告してきたが、今回新たに史上最大となるGIGASTROKEコンソーシアムがISGC内で結成され、本研究報告に至った。
(注2)ゲノムワイド関連解析(Genome-Wide Association Study:GWAS):
ヒトゲノムの全域に分布する遺伝的バリアントと、病気などの形質との因果関係を網羅的に検討する遺伝統計解析手法。単一遺伝子によらない複雑な疾患の解析に用いられる。これまでに、数百を超える形質や病気を対象に実施され、数多くの関連遺伝子が同定されている。
(注3)多層オミックス情報:
オミックスとは、個体(または種)の特定のレベルの生物学的情報の総体と言ったような意味を持つ接尾辞-ome(オーム)に、「…学」という意味の接尾辞-icsをつけたもの(ome+ics=omics)である。例えば、遺伝情報総体の学問ならgene(遺伝子)+-omics = genomics(ゲノミクス)、遺伝子発現情報(mRNA)総体の学問ならtranscript(転写物質)+-omics=transcriptomics(トランスクリプトミクス)、タンパク質総体の学問ならprotein(タンパク質)+-omics=proteomics(プロテオミクス)と呼ぶ。実際にはヒトを含む生物では遺伝子からmRNAが転写され、mRNAからタンパク質が翻訳される、というようにこれらのそれぞれのレベルのオミックス情報が密接に関連しあっている。そこで、これらの多数のレベルのオミックス情報を組み合わせた場合、多層オミックス(またはマルチオミックス)などと呼ぶ。
お問い合わせ
新領域創成科学研究科 広報室