ペンシルベニア州立大学の統計学者が、一般に知られていないグループから遺伝データを収集し、複雑なデータを最大限に活用するための統計手法を改良しています。 Penn State statistician helps gather genetic data from underrepresented groups and improve statistical methods to make the most of the complex data.
2023-02-01 ペンシルベニア州立大学(PennState)
◆心臓病の遺伝学は非常に複雑です。例えば、「心臓発作の遺伝子」は1つだけではありません。その代わり、多くの遺伝子が互いに、また環境因子と相互作用して、その人の心臓病の全体的なリスクに寄与しています。しかし、どの遺伝子がこれらの「多遺伝子」形質に関与しているか、また、それらの相対的な寄与は、個人間や集団間で異なる可能性がある。ペンシルベニア州立大学の統計学者Xiang Zhuは、国際的な共同研究により、できるだけ多様なサンプルでこれらの遺伝子と心臓病におけるその役割を研究し、リスクに関する理解を深め、最終的にはすべての人の心臓の健康を改善することを目指しています。
◆Zhuは、国際的な研究者チームと協力して、心臓病の原因となる2つの要因(冠動脈疾患-心臓発作につながる最も一般的な心臓病の形態-と心臓病の測定可能な危険因子であるコレステロール値)の遺伝学に関する最大かつ最も遺伝的に多様な2つの研究で重要な役割を担ってきました。
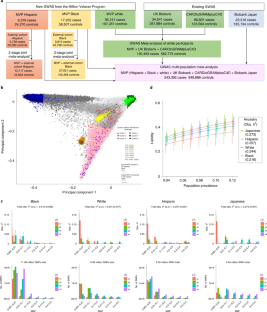
◆最初の研究では、研究チームは、多様な人々を対象とした医療システムを含むMillion Veteran Programから得た冠動脈疾患に関する遺伝子データと、最近発表された研究からのデータを使用しました。その結果、黒人やヒスパニック系などこれまでで最大のサンプルを含む約25万人の冠動脈疾患患者の情報が得られ、研究チームはこれらの集団における疾患の特徴を初めて明らかにすることができました。また、このデータに基づいて作成された多遺伝子リスク・スコアは、主にヨーロッパ人の祖先を持つ集団から得られたデータに基づくこれまでのスコアと同等かそれ以上のものであった。
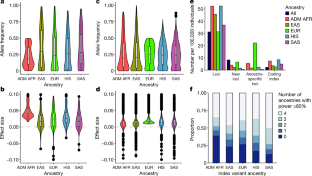
◆次の研究では、ZhuたちはGlobal Lipids Genetics Consortiumと協力して、コレステロールの遺伝学的性質を探った。35カ国から165万人が参加した201の過去の研究データを統合し、コレステロール値に関するこれまでで最大の遺伝学的研究を実施した。コレステロール値は、心臓病、特に心臓発作のリスクと相関の高い低比重リポ蛋白(LDL)コレステロールのレベルを予測するのに非常に有効である。
◆研究者達は、多様なデータに基づく多遺伝子リスクスコアは、ヨーロッパのゲノムデータのみに基づくスコアと比較して、その人がLDLを上昇させるかどうかをより予測することを発見しました。次に、コレステロール関連遺伝子の発現に関与するゲノムの機能要素を調べ、遺伝子と機能要素がどのように相互作用してコレステロールに影響を与えるかを調べ、将来のLDL低下薬のターゲットとなりうるものを同定した。また、特定の遺伝子や遺伝子群が、LDLとアルツハイマー病や肥満などの複数の疾患のリスクを同時に制御している可能性も探った。
<関連情報>
- https://www.psu.edu/news/eberly-college-science/story/improving-heart-health-diversifying-genetic-data/
- https://www.nature.com/articles/s41591-022-01891-3
- https://www.nature.com/articles/s41586-021-04064-3
遺伝的に多様な集団における冠動脈疾患の大規模ゲノムワイド関連研究 Large-scale genome-wide association study of coronary artery disease in genetically diverse populations
Catherine Tcheandjieu, Xiang Zhu, Austin T. Hilliard, Shoa L. Clarke, Valerio Napolioni, Shining Ma, Kyung Min Lee, Huaying Fang, Fei Chen, Yingchang Lu, Noah L. Tsao, Sridharan Raghavan, Satoshi Koyama, Bryan R. Gorman, Marijana Vujkovic, Derek Klarin, Michael G. Levin, Nasa Sinnott-Armstrong, Genevieve L. Wojcik, Mary E. Plomondon, Thomas M. Maddox, Stephen W. Waldo, Alexander G. Bick, Saiju Pyarajan, Regeneron Genetics Center, CARDIoGRAMplusC4D Consortium, Biobank Japan, Million Veteran Program, …Themistocles L. Assimes
Nature Medicine Published:01 August 2022
DOI:https://doi.org/10.1038/s41591-022-01891-3
Abstract
We report a genome-wide association study (GWAS) of coronary artery disease (CAD) incorporating nearly a quarter of a million cases, in which existing studies are integrated with data from cohorts of white, Black and Hispanic individuals from the Million Veteran Program. We document near equivalent heritability of CAD across multiple ancestral groups, identify 95 novel loci, including nine on the X chromosome, detect eight loci of genome-wide significance in Black and Hispanic individuals, and demonstrate that two common haplotypes at the 9p21 locus are responsible for risk stratification in all populations except those of African origin, in which these haplotypes are virtually absent. Moreover, in the largest GWAS for angiographically derived coronary atherosclerosis performed to date, we find 15 loci of genome-wide significance that robustly overlap with established loci for clinical CAD. Phenome-wide association analyses of novel loci and polygenic risk scores (PRSs) augment signals related to insulin resistance, extend pleiotropic associations of these loci to include smoking and family history, and precisely document the markedly reduced transferability of existing PRSs to Black individuals. Downstream integrative analyses reinforce the critical roles of vascular endothelial, fibroblast, and smooth muscle cells in CAD susceptibility, but also point to a shared biology between atherosclerosis and oncogenesis. This study highlights the value of diverse populations in further characterizing the genetic architecture of CAD.
脂質のゲノムワイド関連研究における遺伝的多様性の威力 The power of genetic diversity in genome-wide association studies of lipids
Sarah E. Graham, Shoa L. Clarke, Kuan-Han H. Wu, Stavroula Kanoni, Greg J. M. Zajac, Shweta Ramdas, Ida Surakka, Ioanna Ntalla, Sailaja Vedantam, Thomas W. Winkler, Adam E. Locke, Eirini Marouli, Mi Yeong Hwang, Sohee Han, Akira Narita, Ananyo Choudhury, Amy R. Bentley, Kenneth Ekoru, Anurag Verma, Bhavi Trivedi, Hilary C. Martin, Karen A. Hunt, Qin Hui, Derek Klarin, VA Million Veteran Program, Global Lipids Genetics Consortium*, …Cristen J. Willer
Nature Published:09 December 2021
DOI:https://doi.org/10.1038/s41586-021-04064-3
Abstract
Increased blood lipid levels are heritable risk factors of cardiovascular disease with varied prevalence worldwide owing to different dietary patterns and medication use1. Despite advances in prevention and treatment, in particular through reducing low-density lipoprotein cholesterol levels2, heart disease remains the leading cause of death worldwide3. Genome-wideassociation studies (GWAS) of blood lipid levels have led to important biological and clinical insights, as well as new drug targets, for cardiovascular disease. However, most previous GWAS4,5,6,7,8,9,10,11,12,13,14,15,16,17,18,19,20,21,22,23 have been conducted in European ancestry populations and may have missed genetic variants that contribute to lipid-level variation in other ancestry groups. These include differences in allele frequencies, effect sizes and linkage-disequilibrium patterns24. Here we conduct a multi-ancestry, genome-wide genetic discovery meta-analysis of lipid levels in approximately 1.65 million individuals, including 350,000 of non-European ancestries. We quantify the gain in studying non-European ancestries and provide evidence to support the expansion of recruitment of additional ancestries, even with relatively small sample sizes. We find that increasing diversity rather than studying additional individuals of European ancestry results in substantial improvements in fine-mapping functional variants and portability of polygenic prediction (evaluated in approximately 295,000 individuals from 7 ancestry groupings). Modest gains in the number of discovered loci and ancestry-specific variants were also achieved. As GWAS expand emphasis beyond the identification of genes and fundamental biology towards the use of genetic variants for preventive and precision medicine25, we anticipate that increased diversity of participants will lead to more accurate and equitable26 application of polygenic scores in clinical practice.