2023-08-28 理化学研究所,東京医科歯科大学,バイオ産業情報化コンソーシアム
理化学研究所(理研)生命機能科学研究センター 生体分子動的構造研究チームの嶋田 一夫 チームリーダー(バイオ産業情報化コンソーシアム(JBIC)特別顧問)、今井 駿輔 上級研究員、東京医科歯科大学 高等研究院卓越研究部門 細胞構造生理学研究室の藤吉 好則 特別栄誉教授、鈴木 博視 特任准教授らの共同研究チームは、脳心筋炎ウイルス[1]のゲノムRNAが宿主細胞のタンパク質合成系を乗っ取る過程において、RNAの動的構造平衡[2]が重要な役割を果たすことを明らかにしました。
本研究成果は、ウイルスが感染した細胞内でのウイルスタンパク質の合成を阻害する、新しいタイプの抗ウイルス薬の開発に貢献すると期待できます。
脳心筋炎ウイルスはセンス一本鎖RNA[3]をゲノムとして持ち、感染細胞に放出されたRNAが宿主細胞のタンパク質合成系(翻訳系)を利用して、増殖に必要なウイルスタンパク質を合成します。この機構はウイルスの効率的な増殖に寄与するものですが、ウイルスRNAがどのように宿主細胞の翻訳系を乗っ取り、タンパク質を直接合成させるのかは明らかではありませんでした。
今回、共同研究チームは、溶液核磁気共鳴(NMR)法[4]とクライオ電子顕微鏡法[5]を組み合わせた構造生物学的解析により、脳心筋炎ウイルスのゲノムRNAの5′非翻訳領域[6]が、複数の立体構造を行き来する動的構造平衡にあることを発見しました。日本医療研究開発機構(AMED)の支援により導入されたBruker社製4K 1GHz NMR装置[7]と、日本電子(JEOL)社製第8世代のクライオ電子顕微鏡装置[8]を用いた解析により、この平衡状態には、ゲノムRNAの二つのドメインが宿主細胞の翻訳開始因子[9]と相互作用できるように配向された構造が含まれており、ゲノムRNAが翻訳開始因子への選択的な結合を介して宿主細胞の翻訳系を乗っ取ることが示されました。
本研究は、科学雑誌『Nature communications』オンライン版(8月28日付:日本時間8月28日)に掲載されました。
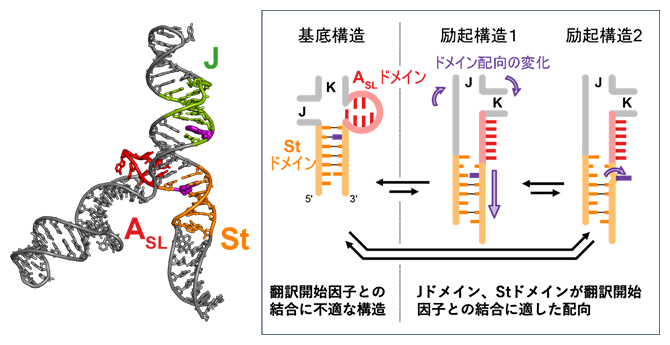
三つの構造を行き来する脳心筋炎ウイルスRNA5’非翻訳領域の動的構造平衡
背景
脳心筋炎ウイルスはセンス一本鎖RNAをゲノムとして持つRNAウイルスの一種であり、哺乳動物(主に齧歯類)に感染すると脳炎や心筋炎を発症することが知られています。このようなRNAウイルスは宿主細胞へ吸着した後、細胞内に放出したセンス一本鎖RNAを宿主細胞のタンパク質合成系(翻訳系)にそのまま送り込むことで、自らの増殖に必要なタンパク質を合成します。
ゲノムとしてアンチセンス一本鎖RNA[3]やDNAを持つウイルスでは、宿主細胞内で逆転写[10]や核への移行といった段階を経て、初めてタンパク質の合成が開始されます。これに対し、ゲノムRNAが宿主細胞内に取り込まれてからすぐにタンパク質合成が開始するセンス一本鎖RNAを持つウイルスの機構は、ウイルスの効率の良い増殖に寄与しています。特に、脳心筋炎ウイルスは極めて高い翻訳効率を示すことが知られており、このRNAが宿主細胞の翻訳系を直接”乗っ取る”機構を理解することは、RNAウイルスの増殖機構の理解を深めるのみならず、抗ウイルス薬としての増殖抑制剤の開発指針を提示することにつながる重要な課題です。
脳心筋炎ウイルスのゲノムRNAが宿主の翻訳系を利用する際には、細胞にもともと存在している二つの翻訳開始因子(eIF4GとeIF4A)を使います。これまでに、脳心筋炎ウイルスのゲノムRNAのうち5′非翻訳領域に存在するJ-K-Stと呼ばれる領域が、eIF4Gタンパク質のHEAT1ドメインに直接結合することが、宿主細胞の翻訳機構の利用に必要であることが分かっています(図1)。eIF4Gは本来、RNAとの相互作用が機能上必須ではありません。にもかかわらず、脳心筋炎ウイルスのゲノムRNAがeIF4Gと選択的に結合する機構は不明でした。
本研究では、溶液核磁気共鳴(NMR)法とクライオ電子顕微鏡法を組み合わせた構造生物学的解析によって、脳心筋炎ウイルスのゲノムRNAが宿主細胞の翻訳開始因子eIF4Gに結合する機構の解明を試みました。
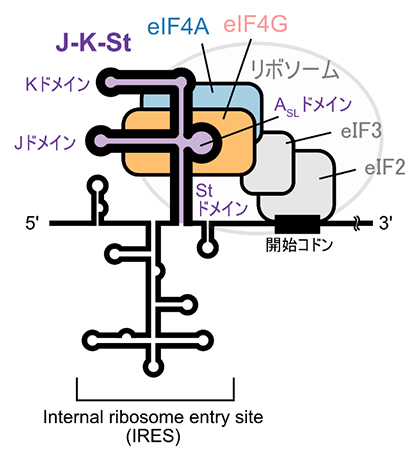
図1 脳心筋炎ウイルスゲノムRNAの模式図
脳心筋炎ウイルスゲノムについて、主にRNAの5′末端側のInternal Ribosome Entry Site(IRES)と呼ばれる非翻訳領域のループやヘアピンなどの構造を示す。IRES RNAは立体構造を組むことで、直接宿主細胞のリボソームを呼び込む機能を持つ。脳心筋炎ウイルスゲノムでは、IRESのJ-K-St領域が翻訳開始因子eIF4Gと結合して、RNA/eIF4G/eIF4Aの3者複合体を形成する。
研究手法と成果
今回、共同研究グループは、脳心筋炎ウイルスゲノムRNAのJ-K-St領域(108塩基)と、ヒト翻訳開始因子eIF4GのHEAT1ドメイン(eIF4GHEAT1、247アミノ酸残基)、eIF4A(406アミノ酸残基)の3者複合体のクライオ電子顕微鏡構造を初めて決定しました(図2)。得られた構造は、Y字型構造を取るJ-K-Stが持つ3本のステム[11](局所的なRNA二本鎖部分)のうち、JドメインとStドメインだけがeIF4GHEAT1と直接相互作用し、Kドメインは結合に関わらないことを示していました(図2B)。
また、翻訳開始因子と結合していない単独状態J-K-StのNMR構造と、クライオ電子顕微鏡法で決定した複合体中のJ-K-Stの構造を比較したところ、Y字型構造の連結部に存在するゲノムRNAのASLドメインの構造が大きく変化し、J、K、Stの3ドメインの配向も変化することが分かりました(図2C)。さらに、単独状態ではステムの内部に遮蔽されている二つの塩基、すなわちJドメインの724番目のアデニン(A724)とStドメインの777番目のグアニン(G777)がステムの外側に飛び出し、eIF4GHEAT1と直接相互作用していることも明らかになりました(図2B)。
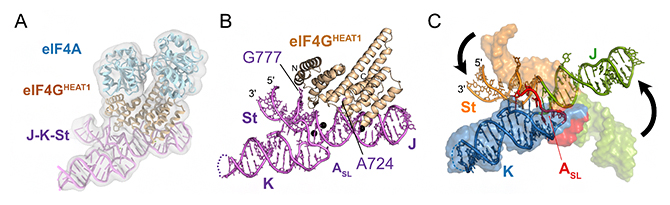
図2 J-K-St/eIF4GHEAT1複合体のクライオ電子顕微鏡構造
A.J-K-St/eIF4GHEAT1/eIF4A複合体のクライオ電子顕微鏡構造。分解能は3.8オングストローム(å、1åは100億分の1メートル)。J-K-StはeIF4GHEAT1に結合し、eIF4Aとは接触しない。
B.複合体構造中におけるeIF4GHEAT1/J-K-Stの拡大図。Jドメイン、StドメインがeIF4GHEAT1と直接相互作用し、Kドメインは相互作用しない。これら3ドメインの連結部に存在するASLドメインもeIF4GHEAT1とは直接相互作用しない。
C.本研究で得られたeIF4GHEAT1との複合体のクライオ電子顕微鏡構造中のJ-K-St構造(リボン表示)と、先行報告における単独状態J-K-StのNMR構造(表面表示)の重ね合わせ。J、K、St、ASL各ドメインを両構造において同じ色で示した。Kドメインの位置を基準に両者を重ね合わせると、JドメインとStドメインの配置が大きく変化している(黒矢印)。
次に、複合体を形成する前のJ-K-StがどのようにeIF4GHEAT1を認識し結合するのかを解明するため、共鳴周波数600メガヘルツ(MHz、1MHzは100万ヘルツ)と1ギガヘルツ(GHz、1GHzは10億ヘルツ)の溶液NMR装置を用いて、運動性の解析を行いました(図3A)。この解析により、J-K-St中のStドメインとASLドメインが、互いに関連しながら複数の構造を行き来する協奏的な構造平衡(動的構造平衡)にあることが初めて明らかになりました。
特に、複合体構造中においてeIF4Gと結合するStドメインのG777がeIF4Gと結合していない単独状態においては、基底構造、励起構造1、励起構造2と名付けた三つの構造間で平衡状態にあることが分かりました(図3B)。このうち励起構造2では、Stドメインの二次構造の組み替え(分子内で二本鎖を形成する相手の入れ替え)とG777のステム外部への突出が起こることが示されました(図4)。このG777のステム外部への突出は、クライオ電子顕微鏡による観察で複合体構造中に見られた構造(図2B)と一致するものです。
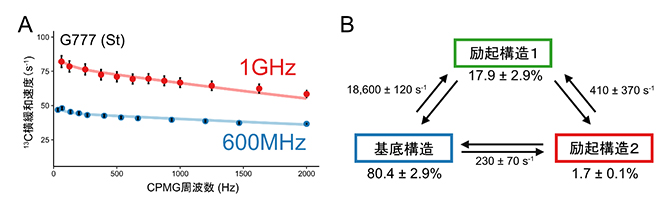
図3 G777の溶液NMR解析による構造平衡解析
A.複数の状態の間を化学交換する核スピンの挙動が磁場強度によって異なることを利用し、その運動性を定量する13C緩和分散実験の結果を示す。本研究では、磁場強度1GHzでの挙動を600MHzでの挙動と比較した。Stドメインの777番目のグアニン(G777)の8位13Cシグナルの横緩和速度のCPMG周波数依存性を、1H共鳴周波数1GHz(赤)と600MHz(青)で測定した。横緩和速度定数がCPMG周波数依存的に変化したことから、G777にマイクロ秒~ミリ秒タイムスケールの運動性があることが示された。
B.13C緩和分散実験から明らかになった、30℃におけるG777の3構造の存在比と交換速度定数。先行研究で示されていたJ-K-Stドメイン単独でのNMR構造に対応する基底構造のほか、存在確率の低い二つの構造(励起構造1、2)が初めて観測された。
さらにASLドメインも、Stドメインの二次構造の組み替えに伴って構造変化し、励起構造1と2では、eIF4GHEAT1/eIF4Aとの複合体で見られる構造に類似した構造を取っていることを見いだしました。このASLドメインの構造変化によって、eIF4GHEAT1が存在しないときでも、J、K、Stドメインの配向がeIF4GHEAT1への結合に適したものになり得ることが示されました。
以上の結果から、脳心筋炎ウイルスゲノムRNAが宿主細胞の翻訳系を乗っ取るための戦略に、J-K-St領域が果たしている役割が見えてきました。J-K-St/eIF4GHEAT1/eIF4A複合体のクライオ電子顕微鏡構造から、JドメインとStドメインがeIF4GHEAT1に直接結合していることが明らかになりましたが、先行研究では、Jドメイン、Stドメインそれぞれ単独ではeIF4GHEAT1に結合できず、ASLドメインによる連結が必要であることが示されていました。
また、eIF4GHEAT1の構造上には、RNA結合に有利な塩基性アミノ酸残基や芳香族性アミノ酸残基から成る広い相互作用面が存在しません。しかし、脳心筋炎ウイルスのゲノムRNAは、それぞれ単独では親和性が低く結合できない2本のステムドメイン(Jドメイン、Stドメイン)を、ASLドメインの構造平衡によって適切な配向に調節することにより、本来はRNAと結合しにくいeIF4GHEAT1への選択的結合を可能にしていることが示されました(図4)。こうして宿主細胞翻訳系への手掛かりを得たウイルスゲノムは、感染細胞内で効率よく自分のタンパク質を合成させるのです。
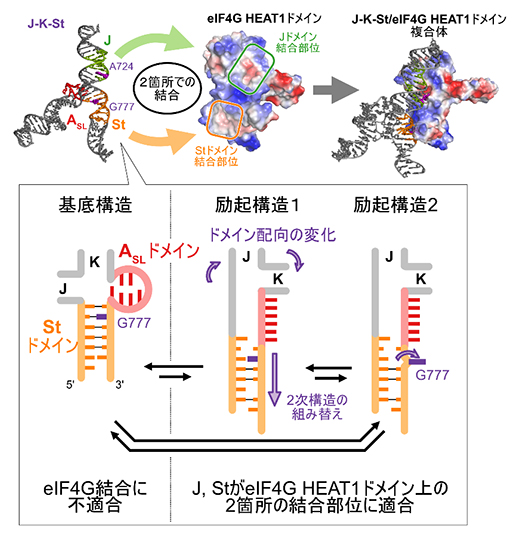
図4 Stドメイン、ASLドメインの構造平衡とeIF4GHEAT1への結合
翻訳開始因子と複合体を形成していない状態で、StドメインとASLドメインは、Stドメインの二次構造の組み替え(分子内で二本鎖を形成する相手の入れ替え)とASLドメインの構造変化を伴う3パターンの構造を行き来する協奏的な構造平衡(動的構造平衡)にある。基底構造は安定な構造だが、eIF4GHEAT1と直接相互する777番目のグアニン(G777)がStドメイン内で塩基対を形成し、外部に露出していない。一方、励起構造1、2においては、Stドメインの二次構造の組み替えによりG777が塩基対を形成しておらず、励起構造2ではステム(局所的なRNAの二本鎖部分)外部に飛び出す。さらに、ASLドメインの構造変化によってJ、K、Stドメイン配向の変化が起こり、eIF4GHEAT1への結合に適したものとなる。
今後の期待
本研究により、センス一本鎖RNAをゲノムに持つ脳心筋炎ウイルスが、宿主細胞の翻訳系を利用して効率よく増殖する機構が明らかとなりました。脳心筋炎ウイルスのゲノムRNAは、RNAとの結合に適した広い界面を持たないタンパク質(翻訳開始因子)に対して、親和性が低くそれぞれ単独では結合できない複数のRNAドメインを組み合わせ、連結部の動的な構造平衡を通じてそれらのドメインを適切に配置することにより、標的タンパク質への選択的な結合を可能にしています。この仕組みを基にした創薬として、ウイルスRNAに結合してその動的構造平衡を抑制し、適切なドメイン配向を取れなくすることでタンパク質合成を抑制するような、新しい作用機序を持つウイルス増殖抑制薬の開発方針が考えられます。
今回のJ-K-St領域の動的構造の検出には、日本医療研究開発機構(AMED)の支援により導入された、4K 1GHz NMR装置としては世界第1号機となる超高磁場NMR装置が用いられました。また、J-K-St/eIF4GHEAT1/eIF4A複合体のクライオ電子顕微鏡解析には、干渉性の良い光源とエネルギー分光装置を備え、損傷低減のために液体ヘリウムでも冷却が可能な装置を備えた300kVクライオ電子顕微鏡が用いられました。今後、存在割合がより低いマイナー構造の高感度検出や、巨大分子の動的構造解析に4K 1GHz NMR装置が活用され、100塩基以下の小さいRNAや脂質膜内に埋まってこれまでに構造解析が困難であった膜タンパク質の構造解析にこのクライオ電子顕微鏡装置が活用されることにより、構造に導かれた合理的創薬が加速するものと期待できます。
さらに本研究成果は、分子の機能に直結する運動性を保った動的構造を解析できる溶液NMR法と、生体分子の静的立体構造を解析できるクライオ電子顕微鏡法の長所を組み合わせた解析によって初めてもたらされました。今後、両手法を統合した構造生物学のさらなる発展も期待できます。
補足説明
1.脳心筋炎ウイルス
マウスやラットなどの齧歯類を主な宿主とする、カルジオウイルスに属するRNAウイルス。
2.動的構造平衡
タンパク質や核酸などの立体構造が一つではなく、複数の異なる構造間の交換状態にあり、見かけ上はそれぞれの構造比が一定の状態にあること。
3.センス一本鎖RNA、アンチセンス一本鎖RNA
センス一本鎖RNAは、一本鎖RNAのうち遺伝情報を直接コードしているもの。タンパク質を作るための遺伝コードと直接対応する配列を持つRNAを指す。これに対し、その逆相補配列を持つRNAはアンチセンス一本鎖RNAと呼ばれる。RNAウイルスには、センス一本鎖RNAを持つものとアンチセンス一本鎖RNAを持つものが存在する。
4.溶液核磁気共鳴(NMR)法
溶液中の生体分子などを、強い磁場中に置くことで生じる原子核の共鳴現象を観測することで、生体分子の構造や運動性を原子レベルで解析できる分光法。磁場の強さは慣習的に、その磁場における1H(プロトン)の共鳴周波数を用いてMHz(100万Hz)単位で表される。NMRはnuclear magnetic resonanceの略。
5.クライオ電子顕微鏡法
タンパク質などの生体試料の構造を決定するため、試料溶液を急速凍結して分子をごく薄い氷の層に閉じ込めた状態で、液体窒素温度(-196℃)で電子線を当てて電子顕微鏡により分子の像を観察する手法。
6.5′非翻訳領域
タンパク質合成の鋳型となるRNA分子において、開始コドンより5′末端側に存在する、タンパク質に翻訳されない領域のこと。
7.4K 1GHz NMR装置
水素原子核の共鳴周波数が1GHz(10億Hz)である最新型の超高磁場NMR装置。NMR装置の感度や分解能は、磁場が高いほど向上する。従来の超高磁場NMR装置ではマグネットを2K(-271°C)まで冷却する必要があったが、この装置では冷却温度4K(-269°C)で1GHzを達成している。このため、装置の総重量は前世代機の3分の1(2,300kg)まで小型化され、漏洩磁場の指標となる5ガウスライン半径は2分の1(1.9m)に、冷却に用いるヘリウムの消費量は3分の1(80mL/hr)に抑えられている。
8.第8世代のクライオ電子顕微鏡
液体ヘリウムで試料を冷却することで電子線損傷を低減させることができるクライオ電子顕微鏡が、藤吉博士と日本電子との共同で開発されてきた。第8世代と呼ばれるクライオ電子顕微鏡では、液体ヘリウム冷却でなく液体窒素冷却条件でも撮影が可能になったことに加えて、冷陰極の電子銃を備えて干渉性の高い電子線を用いることにより分解能が向上した。さらに、試料を包埋する氷などから発生する非弾性散乱電子を除いて、ノイズの少ない像を撮影することができる最新鋭のクライオ電子顕微鏡である。
9.翻訳開始因子
細胞内でタンパク質合成(翻訳)を行うリボソームが合成を開始する際に、協調的に働くタンパク質の総称。真核生物の翻訳開始因子は、真核生物型開始因子(eukaryotic initiation factor: eIF)と呼ばれ、eIF1、eIF1Aなど少なくとも12種の翻訳開始因子が知られている。
10.逆転写
逆転写酵素と呼ばれる酵素の働きによって、RNAを鋳型として逆相補配列を持つDNAを合成する反応のこと。
11.ステム
RNAの二次構造の一種。一本鎖RNAは、分子内で塩基同士(AとU、CとG)が水素結合することで局所的に二本鎖を形成する。この二本鎖の部分をステムと呼び、ステムを構成するRNAが折れ曲がって環状の一本鎖となっている構造をステムループと呼ぶ。
共同研究チーム
理化学研究所 生命機能科学研究センター 生体分子動的構造研究チーム
チームリーダー 嶋田 一夫(シマダ・イチオ)
(バイオ産業情報化コンソーシアム 特別顧問、広島大学 副学長)
上級研究員 今井 駿輔(イマイ・シュンスケ)
東京医科歯科大学 高等研究院卓越研究部門 細胞構造生理学研究室
特別栄誉教授 藤吉 好則(フジヨシ・ヨシノリ)
特任准教授 鈴木 博視(スズキ・ヒロシ)
研究支援
本研究は、理化学研究所運営費交付金(生命機能科学研究)で実施し、日本医療研究開発機構(AMED)「次世代治療・診断実現のための創薬基盤技術開発事業」(RNA標的創薬技術開発)による助成を受けて行われました。
原論文情報
Shunsuke Imai, Hiroshi Suzuki, Yoshinori Fujiyoshi, Ichio Shimada, “Dynamically regulated two-site interaction of viral RNA to capture host translation initiation factor”, Nature communications, 10.1038/s41467-023-40582-6
発表者
理化学研究所
生命機能科学研究センター 生体分子動的構造研究チーム
チームリーダー 嶋田 一夫(シマダ・イチオ)
(バイオ産業情報化コンソーシアム 特別顧問)
上級研究員 今井 駿輔(イマイ・シュンスケ)
東京医科歯科大学 高等研究院 卓越研究部門細胞構造生理学研究室
特別栄誉教授 藤吉 好則(フジヨシ・ヨシノリ)
特任准教授 鈴木 博視(スズキ・ヒロシ)
報道担当
理化学研究所 広報室 報道担当
東京医科歯科大学 総務部総務秘書課広報係
バイオ産業情報化コンソーシアム問い合わせ窓口