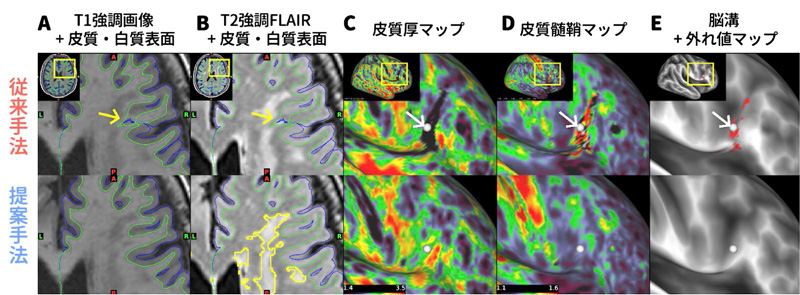
2023-10-03 理化学研究所,名古屋大学
理化学研究所(理研)生命機能科学研究センター エピジェネティクス制御研究チーム(研究当時)の梅原 崇史 チームリーダー(研究当時、現 創薬タンパク質解析基盤ユニット 上級研究員)、ナンド・ダス 研究員(研究当時)、生命医科学研究センター エピゲノム技術研究チーム(研究当時)の蓑田 亜希子 チームリーダー(研究当時、現 ラドバウド大学 准教授)、古関 明彦 副センター長(免疫器官形成研究チーム チームリーダー)、名古屋大学大学院 医学系研究科 腫瘍生物学の近藤 豊 教授らの共同研究グループは、がん遺伝子などの発現を強く活性化するスーパーエンハンサー[1]を、ヒストン[2]H4の高アセチル化[3]状態を指標として再定義することにより、がん細胞の幹細胞性[4]の制御に関わるスーパーエンハンサーを見いだしました。
がんなどの疾患細胞では特定の遺伝子の発現が暴走した状態が見られますが、これは細胞増殖などに関わる遺伝子の制御領域がスーパーエンハンサーを形成することが一因と考えられています。スーパーエンハンサーはこれまで、がん細胞で高発現しているBRD4[5]などのタンパク質や、エピゲノム[6]の構成タンパク質であるヒストンH3の27番目のリシン残基のアセチル化(H3K27ac)が濃縮しているエピゲノムの領域として見いだされてきました。今回、共同研究グループは、ヒトのがん細胞株である膠芽腫(こうがしゅ)細胞[4]を用いて、ヒストンH4の5番目と8番目のリシン残基のアセチル化(H4K5acK8ac)を指標としてスーパーエンハンサーを再定義することにより、がんなどの疾患細胞で特異的に機能している遺伝子を同定する新手法を開発しました。
本研究は、科学雑誌『BMC Genomics』オンライン版(9月27日付)に掲載されました。
背景
私たちの細胞の一つ一つに含まれるヒトゲノムDNAには約2万種類の遺伝子が存在します。これらの遺伝子は一斉には働かず、細胞ごとに決められた遺伝子をいつどこで発現させるかを調節する仕組みがあります。その一つがヒストンと呼ばれるタンパク質による調節です。ヒストンは、4種類のコアタンパク質(H2A、H2B、H3、H4)が二つずつ組み合わさった8量体として存在し、糸巻きのようにDNAを巻き付けることで、長いDNAを核内に収納しています(図1上、クロマチン[2]構造)。ヒストンは、アセチル化やメチル化[7]などの修飾を受けてクロマチン構造を変化させ、ゲノムDNAの特定の領域に存在する遺伝子の発現を制御しています。ヒストンに対する化学修飾は元に戻すことも可能(可逆的)であり、DNAに対する可逆的な化学修飾であるメチル化とともに「エピジェネティクス制御」と呼ばれています。エピジェネティクス制御は、個体の一生を通じて不変であるDNAの塩基配列と異なり、可逆的に遺伝情報を制御する仕組みです。
ヒトのエピジェネティクス制御には、ヒストンのH3とH4に対するアセチル化修飾が重要な役割を担うことが知られています。特に、H4のN末端側に存在する二つのリシン残基[8](K5とK8)が同時にアセチル化された高アセチル化状態(H4K5acK8ac)のヒストンには、がん細胞で高発現しているBRD4などのタンパク質が結合し、がん遺伝子の発現を活性化します(図1下)。
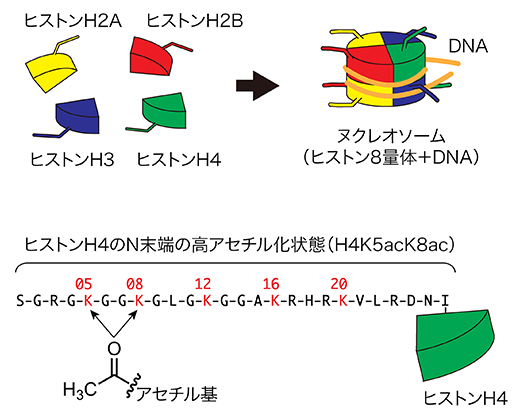
図1 クロマチン構造とヒストンの高アセチル化
4種類のコアタンパク質(H2A、H2B、H3、H4)が二つずつ組み合わさった8量体にDNAが巻き付いたものをヌクレオソームと呼び、これがクロマチンの単位構造である(上)。各ヒストンはアセチル化やメチル化などの修飾を受け、特にH4のN末端側に存在する二つのリシン残基(K5とK8)が同時にアセチル化されたものを高アセチル化状態(H4K5acK8ac)と呼ぶ(下)。
特定の細胞で特に発現が多い遺伝子は、その発現を制御するDNA領域にエンハンサー[1]と呼ばれる配列を複数持つという特徴があります。この長い制御配列を持つDNA領域はスーパーエンハンサーと呼ばれ、細胞の分化や機能に重要な遺伝子の近傍に多く見られ、がん遺伝子の活性化にも関わることが分かりつつあります。スーパーエンハンサーのヒストンは化学修飾されていることが多いと考えられていたため、従来の研究では、スーパーエンハンサーは、アセチル化リシンに結合するタンパク質であるBRD4の局在や、活性化クロマチンの指標であるヒストンH3の27番目のリシン残基のアセチル化(H3K27ac)などが濃縮しているゲノム領域として見いだされてきました。
しかし最近になって、H3K27acは必ずしも遺伝子発現の活性化に必須ではないという報告がありました。つまり、従来の手法で見いだされたスーパーエンハンサーには機能しないものが含まれている可能性が考えられます。一方、BRD4が結合するH4K5acK8acはスーパーエンハンサーの目印になり得ますが、BRD4の局在だけではH4K5acK8acを特異的に検出できなかったため、調べたい細胞のエピゲノムのどこにスーパーエンハンサーが形成されているのかを精密に特定することが困難でした。
研究手法と成果
共同研究グループはこれまでの研究において、ヒストンH4のさまざまなアセチル化状態を試験管内で再現する技術を開発し、その技術を応用してH4K5acK8acを選択的に認識するモノクローナル抗体[9]を開発しました注1、2)。本研究ではこの抗体や、H3K27acを認識する抗体、BRD4を認識する抗体などを用いて、難治性のがんとして知られる膠芽腫の細胞株を用いて、ヒト膠芽腫細胞のエピゲノムのどこにスーパーエンハンサーが形成されているのかを精密に特定することを目指しました。
最初に、膠芽腫の幹細胞株(GSC)、膠芽腫細胞株(U87)、ミクログリア細胞株(C13)においてどのようなヒストン修飾パターンが形成されているのかを、上記の抗体を用いたクロマチン免疫沈降・塩基配列決定(ChIP-seq)法[10]で調べました(図2)。その結果、H4K5acK8acが検出されたエピゲノム領域の多くは、H3K27acや、ヒストンH3のK4のトリメチル化(H3K4me3)、BRD4の局在とそれぞれ重なるものの、H4K5acK8acしか検出されないエピゲノム領域や、H3K27acしか検出されないエピゲノム領域も多数検出されました。この傾向は3種類の細胞株で共通に見られました。さらに、特定のヒストン修飾パターンが見られるエピゲノム領域の数が細胞株の間で大きく違うことから、H4K5acK8acをはじめとするヒストン修飾のエピゲノムにおける位置は細胞株ごとに大きく異なることが分かりました。
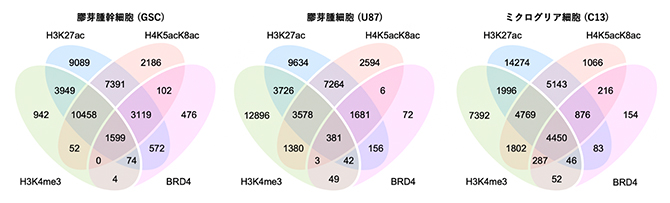
図2 膠芽腫関連細胞におけるヒストンH4の高アセチル化と他のヒストン修飾の分布
膠芽腫幹細胞株(GSC、左)、膠芽腫細胞株(U87、中央)、ミクログリア細胞株(C13、右)において3種類のヒストン修飾が見られたエピゲノム領域と、BRD4が結合したエピゲノム領域の重なりをベン図で示した。3種類のヒストン修飾(H3K27ac、H4K5acK8ac、H3K4me3)、およびBRD4をChIP-seq解析で検出したエピゲノム領域の数をそれぞれの重なりの位置に示している。
近年、がんが発生する仕組みについて、少数の幹細胞が大量のがん細胞を作り出す「がん幹細胞」の存在が注目されています。そこで、膠芽腫の幹細胞株におけるH4K5acK8acとH3K27acの役割の違いを見いだすことを目指しました。まず、これら両方のアセチル化修飾を持つエピゲノム領域をH4K5acK8acの比率が低いものから高いものに沿って6種類のグループに分類し、がん幹細胞株で高発現するBRD4との共局在の割合がグループごとに変化しているかを検討した結果、H4K5acK8acの割合が高まるにつれてBRD4との共局在の割合が高まることが分かりました(図3A)。これは試験管内における直接結合の解析結果と一致しており、細胞内においてもBRD4はH3K27acに比べてH4K5acK8acとより結合していることを示唆する結果です。
BRD4とエピゲノム領域の結合を阻害することで、がん細胞の遺伝子発現パターンが正常細胞の遺伝子発現パターンに近づくケースが知られています。そこで、エピゲノム結合の阻害剤であり、抗がん剤としての効果が期待されている化合物であるJQ1[11]を添加したときの膠芽腫幹細胞株のエピゲノムを解析しました。その結果、遺伝子発現の制御に関わるプロモーター[1]とエンハンサーにおいて、JQ1の添加によってBRD4はほぼ完全にエピゲノムから解離する一方、H4K5acK8acは極めて頑強にエピゲノムに維持されることが分かりました(図3B)。この結果は、共同研究グループが肺がん細胞株で以前に報告した結果注1)と一貫性があり、H4K5acK8acの頑強性は多くのがん細胞のエピゲノムに共通して見られる特徴であると考えられます。
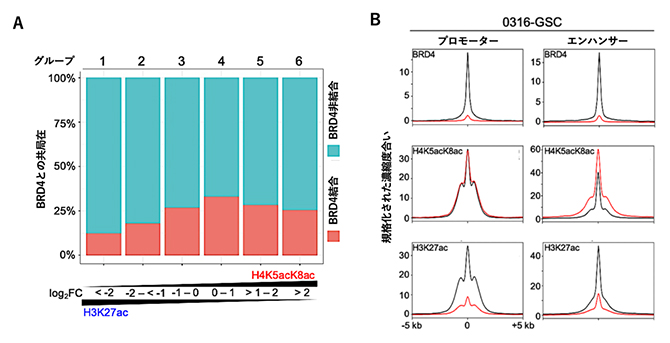
図3 膠芽腫幹細胞株におけるH4K5acK8acとBRD4の共局在と結合阻害剤の影響
A)H3K27acに対するH4K5acK8acの割合とBRD4との共局在性の相関解析。横軸のlog2FC(fold change)は、H3K27acのChIP-seqピーク強度に対するH4K5acK8acのChIP-seqピーク強度の対数比を示す。グループ1から6のうち、右側のグループほどH3K27acに対するH4K5acK8acの割合が高い。H3K27acに富んだグループ1と比べるとH4K5acK8acに富んだグループ4から6の方がBRD4との共局在性が高いことがわかる。
B)BRD4阻害剤のJQ1の添加によるプロモーターおよびエンハンサー領域のChIP-seqピークの変化。ChIP-seqで用いた検出抗体は各パネルの左上に示す。グラフはピークの位置を揃えて平均化したChIP-seqデータを示し、黒色はJQ1無し、赤色はJQ1ありのデータを示す。ピークが高いほど検出抗体に対応するアセチル化修飾またはタンパク質の存在頻度が高いことを示す。ピークの中心を0、ピークから上流または下流に5kb(キロベース、1bは一つの塩基対を示す)離れた位置を±5kbで示す。JQ1を添加すると、BRD4抗体で得られるChIP-seqピークは著しく減少した。これは、BRD4とエピゲノムの結合がほぼ無くなったことを意味する。一方、H4K5acK8ac抗体で得られるピークはJQ1を添加しても減少しなかった。これらの結果から、BRD4の結合が無くなってもH4K5acK8acは維持されることが分かる。
さらに、H4K5acK8acがスーパーエンハンサーの指標になるかを検証するため、エンハンサー領域をH4K5acK8acとH3K27acのそれぞれの数が多い順に順位付けしました(図4)。これらのアセチル化修飾が特に多い領域は、遺伝子発現を著しく高めるスーパーエンハンサーであると考えられます。このスーパーエンハンサーをH4K5acK8acとH3K27acのそれぞれの数を指標として判定し、スーパーエンハンサーとして判定された領域が制御している標的遺伝子を調べたところ(図4A、C)、約半数のスーパーエンハンサー領域と制御標的遺伝子が重複しましたが、およそ4割のスーパーエンハンサー領域や制御標的遺伝子はH4K5acK8acまたはH3K27acのいずれかの指標のみで見いだされました(図4B)。
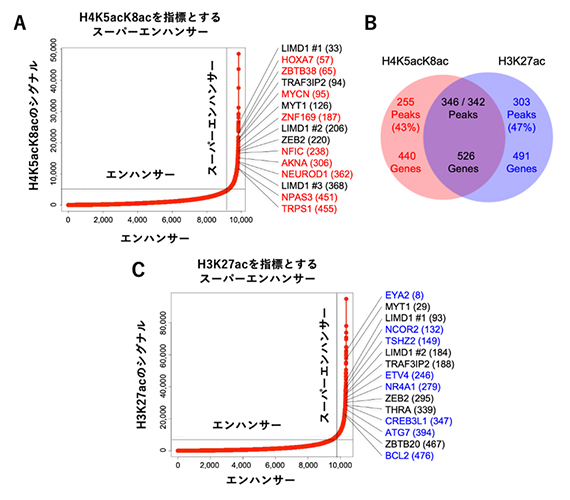
図4 H4K5acK8acを指標とした順位付けによるスーパーエンハンサーの再定義
膠芽腫幹細胞のエンハンサーについて、H4K5acK8ac(A)またはH3K27ac(C)のシグナルを指標として順位付けした。特にシグナルの数が多いスーパーエンハンサーとその制御標的遺伝子をそれぞれの図中に示した。横軸は、検出された縦軸のシグナルの数を少ない方から並べたときの順位を示す。図の右側に、それぞれのスーパーエンハンサーが制御する代表的な遺伝子を示す。遺伝子の右側の括弧内の数字は、縦軸のシグナルの数を多い方から並べたときの順位を示す。遺伝子の色は、赤色はH4K5acK8acの順位付けでのみスーパーエンハンサーが見いだされた制御標的遺伝子、青色はH3K27acの順位付けでのみスーパーエンハンサーが見いだされた制御標的遺伝子、黒色は両者の順位付けで共通にスーパーエンハンサーが見いだされた制御標的遺伝子を示す。ベン図(B)はH4K5acK8acまたはH3K27acで判定したスーパーエンハンサー(ピーク)の数と制御標的遺伝子の数を示す。
H4K5acK8acとH3K27acの指標で判定したスーパーエンハンサーの半数近くが異なっていたことから、両者のそれぞれの指標でのみスーパーエンハンサーと判定された領域の役割を検証しました。H4K5acK8acの指標で判定されたスーパーエンハンサーのうち、がん遺伝子(MYCNおよびNFIC)を制御する、H4K5acK8acの数において上位に位置するスーパーエンハンサーをCRISPR/Cas9法によるゲノム編集[12]で欠失させ、その影響を検証しました。
その結果、スーパーエンハンサーの欠失によってMYCNとNFICのそれぞれの発現が有意に減少するだけでなく、膠芽腫幹細胞の細胞増殖能が有意に低下し、膠芽腫の幹細胞性に関連する他の遺伝子の発現も有意に減少することが分かりました。また、膠芽腫の幹細胞は細胞同士が接着した細胞塊を形成しますが、MYCNやNFICを制御するスーパーエンハンサーを欠失すると細胞塊の形成能も有意に低下しました(図5)。一方、H3K27acの指標によって特異的にスーパーエンハンサーと判定された複数のゲノムDNA領域についても同様の検討を行いましたが、本研究で調べた限りではH4K5acK8ac指標で判定したスーパーエンハンサーと同様の傾向は見られませんでした。

図5 H4K5acK8acを指標として見いだしたスーパーエンハンサーの膠芽腫幹細胞性への関与
ゲノム編集によりがん遺伝子のスーパーエンハンサーを欠失させた膠芽腫幹細胞株の位相差画像。スケールバーは50マイクロメートル(µm、1µmは100万分の1メートル)を示す。ゲノム編集を行っていない細胞株(左)が培養幹細胞の特徴である細胞塊を形成しているのに対し、MYCN(中)やNFIC(右)を制御するスーパーエンハンサーをゲノム編集で欠失させると、細胞塊の形成能が低下した。
これらの解析結果から、従来のエピゲノム研究においてスーパーエンハンサーの判定基準の一つとして用いられてきているH3K27acに加えて、H4K5acK8acの検出がスーパーエンハンサーを精密に判定するために有効な手法であることが分かりました(図6)。
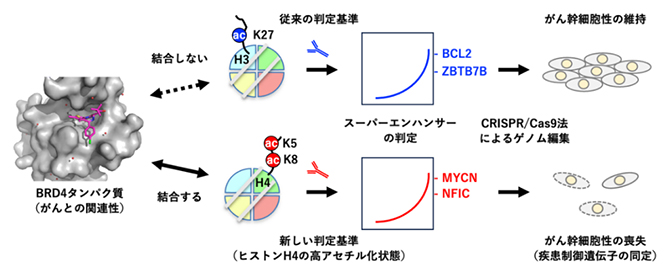
図6 ヒストンH4の高アセチル化状態を指標とするスーパーエンハンサーの判定法
活性化クロマチンの指標であるH3K27acで判定されるスーパーエンハンサーは、がんとの関連が深いBRD4とは結合せず、またゲノム編集で欠失させてもがん幹細胞性は維持されたままだった。一方、H4K5acK8acで判定されるスーパーエンハンサーは、BRD4が結合する標的であり、ゲノム編集で欠失させるとがん幹細胞性が喪失する。
注1)2018年8月8日プレスリリース「『がんエピゲノム』を検出する新手法」
注2)2020年11月26日プレスリリース「エピゲノムの制御を受けた転写の方程式」
今後の期待
スーパーエンハンサーは、がん細胞や炎症などの疾患において疾患関連遺伝子の発現を暴走させる一因として近年注目を集めています。スーパーエンハンサーの形成には、多くのがん細胞に共通して発現するBRD4などのタンパク質がエピゲノムのアセチル化領域に結合することが鍵となっています。しかし、細胞内のエピゲノム研究を先導する国際コンソーシアム注3、4)が解析を推奨しているヒストンのアセチル化修飾はH3K27acやヒストンH3の9番目のリシン残基のアセチル化(H3K9ac)に限られています。ヒストンのアセチル化修飾はH3K27acやH3K9ac以外にも数多くのヒストンや残基で起こることが知られており、特にヒストンH4の高アセチル化修飾は親細胞のエピゲノム情報を娘細胞に継承したり、遺伝子転写を活性化したりする上で重要な修飾と考えられています注5)。本研究から、ヒストンH4の高アセチル化修飾であるH4K5acK8acを指標とするスーパーエンハンサーの判定法は、従来のスーパーエンハンサーの判定法よりも優れた特徴を有していることが明らかになりました。
今回の研究では、難治性疾患の一種である膠芽腫細胞をモデル系として選択しましたが、本手法は細胞種を問わずに利用できます。今後は、さまざまな疾患細胞に存在するスーパーエンハンサーを今回開発した手法で再定義することによって、これまで見いだされていなかった疾患制御遺伝子の発見や疾患状態の精密な診断技術の開発につながることが期待されます。
注3)NIH Roadmap Epigenomics Mapping Consortium![]()
注4)International Human Epigenome Consortium(IHEC)![]()
注5)2023年7月17日プレスリリース「遺伝子発現を制御するエピゲノムの複製と転写」
補足説明
1.スーパーエンハンサー、エンハンサー、プロモーター
遺伝子発現を制御する機能を持つDNA配列。主に遺伝子の本体部分から離れた上流や下流に位置し、遺伝子の転写効率を変化させる特定のDNA配列のうち、転写効率を高める部分をエンハンサー領域(配列)という。複数のエンハンサー領域から構成された長いDNA領域で転写効率を強く高める部分をスーパーエンハンサーという。一方、ゲノムDNA上でRNAを転写開始する位置の近くにあり、遺伝子を発現させる機能を持つ部分をプロモーター領域(配列)という。
2.ヒストン、クロマチン
DNAを巻き付けることで長大なDNAを核内に収納するタンパク質をヒストンという。代表的なヒストンはH1、H2A、H2B、H3、H4の5種類があり、H2A、H2B、H3、H4の4種類(コアヒストン)が二つずつ集まってヒストン8量体を形成する。ヒストン8量体の周りにDNAが巻き付いた構造をヌクレオソームと呼ぶ。ヌクレオソームを基本単位とするゲノムDNAとタンパク質の高次複合体をクロマチンと呼ぶ。
3.アセチル化
分子にアセチル基(CH3CO-)を結合する反応。ヒストンはアセチル基転移酵素と脱アセチル化酵素により、リシン残基の側鎖に可逆的なアセチル化修飾を受ける。ヒストンがアセチル化修飾を受けたクロマチンによって制御される遺伝子は通常、転写が活性化している。
4.幹細胞性、膠芽腫細胞
膠芽腫は脳内に生じる悪性度が高い腫瘍の一種。膠芽腫組織の内部には未分化状態の膠芽腫幹細胞が存在している。この細胞は細胞増殖を繰り返しても未分化状態を維持する幹細胞性と呼ばれる性質を備えている。膠芽腫をはじめとする多くのがん組織で幹細胞性を備えたがん幹細胞が見いだされており、これががんの発症や再発と結びついていると考えられている。
5.BRD4
ヒトの核内に存在する一部のタンパク質に共通して含まれるブロモドメインと余剰末端(Bromodomain and Extra-Terminal domain:BETドメイン)を持つタンパク質。ヒトでは4種類(BRD2、BRD3、BRD4、BRDT)のBETドメイン含有タンパク質が知られている。BRD4は多くのがん細胞で高発現して腫瘍促進遺伝子の発現調節に関与することから、NUT中線がん(NMC)や急性骨髄性白血病(AML)をはじめとするがんの治療標的と見なされている。
6.エピゲノム
細胞内の全DNAの塩基配列情報を指す「ゲノム」に対し、DNAやヒストンの化学修飾などで細胞の個性を記憶する情報を「エピゲノム」と呼ぶ。エピゲノムのパターンは細胞ごとに特徴がある。本研究では、ヒストンH4の高アセチル化修飾(H4K5acK8ac)の情報を、ヒストンH3の27番目のリシンのアセチル化(H3K27ac)をはじめとする他のヒストン修飾などの情報と比較して解析を行った。
7.メチル化
分子にメチル基(CH3-)を結合する反応。ヒストンはメチル基転移酵素と脱メチル化酵素により、リシン残基とアルギニン残基の側鎖に可逆的なメチル化修飾を受ける。ヒストンがメチル化修飾を受けたクロマチンによって制御される遺伝子は、メチル化される残基の種類、位置、度合いに依存して転写が活性化または抑制化される。
8.リシン残基
タンパク質を構成するアミノ酸の一つ。1文字略称はK。側鎖にアミノ基を持つため、タンパク質中の残基としてアミノ基へのアセチル化やメチル化修飾の標的となる。
9.モノクローナル抗体
タンパク質などの特定の生体分子に特異的に結合する抗体のうち、抗体産生細胞とがん細胞が融合したハイブリドーマから産生された抗体のこと。抗体産生細胞は主として1種類の抗体を作るため、特定の種類の抗体を大量に得ることができる。
10.クロマチン免疫沈降・塩基配列決定(ChIP-seq)法
クロマチンに含まれる分子を抗体で免疫沈降し、その領域のDNA配列を解読する方法。次世代シーケンサーを用いて、共沈降した数千万本から数億本のDNA配列の断片を並列処理で解読・分析する方法をChIP-seq法と呼ぶ。ChIP-seq法をエピゲノム解析に応用すると、特定のエピゲノム修飾が存在するゲノム領域を高分解能で検出できる。ChIPはクロマチン免疫沈降を意味するChromatin immunoprecipitationの略。
11.JQ1
BRD4などのBETドメイン含有タンパク質のアセチル化リシン認識部位に結合してヒストンとBETドメイン含有タンパク質との結合を競合的に阻害する低分子。JQ1やその類縁化合物は多くのがんや炎症などに対する治療効果が期待されている。
12.CRISPR/Cas9法によるゲノム編集
ゲノムDNA中の任意のDNA領域を切断することにより、ゲノムDNAを局所的に改変する手法。ゲノム中の標的配列を認識するガイドRNAをDNA切断酵素の一種であるCas9タンパク質と複合体化させた酵素を用いる。本研究ではスーパーエンハンサーを構成するDNA配列を部分的に欠失して細胞内での機能を検証した。
共同研究グループ
理化学研究所
生命機能科学研究センター
エピジェネティクス制御研究チーム(研究当時)
チームリーダー(研究当時)梅原 崇史(ウメハラ・タカシ)
(現 創薬タンパク質解析基盤ユニット 上級研究員)
研究員(研究当時)Nando D. Das(ナンド・ダス)
技師(研究当時)渡邉 寿美(ワタナベ・ヒサミ)
生命医科学研究センター
エピゲノム技術研究チーム(研究当時)
チームリーダー(研究当時)蓑田 亜希子(ミノダ・アキコ)
(現 ラドバウド大学(オランダ)准教授)
研究員(研究当時)Jen-Chien Chang(レンチェン・チャン)
特別研究員(研究当時)S. Tomas Kelly(トーマス・ケリー)
ゲノム情報解析チーム
チームリーダー Chung-Chau Hon(チョウヂョン・ホン)
特別研究員(研究当時)Marina Lizio(マリーナ・リツィオ)
ゲノム制御ネットワーク研究チーム
研究員 Bogumil Kaczkowski(ボグミル・カチコフスキー)
生命医科学研究センター
副センター長 古関 明彦(コセキ・ハルヒコ)
(免疫器官形成研究チーム チームリーダー)
免疫器官形成研究チーム
研究員 伊藤 伸介(イトウ・シンスケ)
名古屋大学
大学院医学系研究科 腫瘍生物学
教授 近藤 豊(コンドウ・ユタカ)
助教(研究当時)勝島 啓佑(カツシマ・ケイスケ)
未来社会創造機構
特任教授 夏目 敦至(ナツメ・アツシ)
研究支援
本研究は、日本学術振興会(JSPS)科学研究費助成事業学術変革領域(A)「DNAの物性から理解するゲノムモダリティ(領域代表者:西山朋子)」、同基盤研究(B)「がん細胞で頑強に維持される超アセチル化エピゲノムを操作する(研究代表者:梅原崇史)」、同基盤研究(C)「高アセチル化ヒストンH4を指標にした活性化エンハンサーの再定義(研究代表者:ナンド・ダス)」、日本医療研究開発機構(AMED)次世代がん医療創生研究事業「がん細胞の分化制御に関わるエピゲノムを標的とした革新的治療法の開発(研究代表者:近藤豊)」、AMED革新的先端研究開発支援事業(AMED-CREST)「エピゲノム研究に基づく診断・治療へ向けた新技術の創出(研究総括:山本雅之)」の研究課題「エピジェネティクスによるエンハンサー動態制御メカニズムの解明と細胞機能制御への応用(研究代表者:古関明彦)」、科学技術振興機構(JST)戦略的創造研究推進事業個人型研究(PRESTO、さきがけ)「細胞機能の構成的な理解と制御(研究領域総括:上田泰己)」の研究課題「『エピヌクレオソーム』の精密な再構成による遺伝子発現制御解析(研究者:梅原崇史)」などによる助成を受けて行われました。
原論文情報
Nando D. Das, Jen-Chien Chang, Chung-Chau Hon, S. Thomas Kelly, Shinsuke Ito, Marina Lizio, Bogumil Kaczkowski, Hisami Watanabe, Keisuke Katsushima, Atsushi Natsume, Haruhiko Koseki, Yutaka Kondo, Aki Minoda and Takashi Umehara*, “Defining super-enhancers by highly ranked histone H4 multi-acetylation levels identifies transcription factors associated with glioblastoma stem-like properties”, BMC Genomics, 10.1186/s12864-023-09659-w
発表者
理化学研究所
生命機能科学研究センター
エピジェネティクス制御研究チーム(研究当時)
チームリーダー(研究当時)梅原 崇史(ウメハラ・タカシ)
(現 創薬タンパク質解析基盤ユニット 上級研究員)
研究員(研究当時)Nando D. Das(ナンド・ダス)
生命医科学研究センター
エピゲノム技術研究チーム(研究当時)
チームリーダー(研究当時)蓑田 亜希子(ミノダ・アキコ)
(現 ラドバウド大学(オランダ)准教授)
生命医科学研究センター
副センター長 古関 明彦(コセキ・ハルヒコ)
(免疫器官形成研究チーム チームリーダー)
名古屋大学
大学院医学系研究科 腫瘍生物学
教授 近藤 豊(コンドウ・ユタカ)
報道担当
理化学研究所 広報室 報道担当
名古屋大学 医学部・医学系研究科 総務課総務係