2023-12-13 カリフォルニア大学サンディエゴ校(UCSD)
◆この研究結果は、新しい薬を創造するためのカスタムプロテインの設計に寄与し、DNAの持つ情報量を拡張する可能性があるとされています。研究は、AEGIS(Artificially Expanded Genetic Information System)と呼ばれる標準の遺伝子アルファベットの新バージョンを使用し、二つの新しい塩基対を組み合わせています。これにより、合成塩基対が自然な塩基対と同じ構造を持つことが確認され、転写過程で区別されないことが示されました。
◆研究者たちは今後、他の合成塩基対と細胞酵素の組み合わせにおいても同様の効果が見られるかどうかを検証することに興味を示しています。
<関連情報>
- https://today.ucsd.edu/story/enzymes-cant-tell-artificial-dna-from-the-real-thing
- https://www.nature.com/articles/s41467-023-43735-9
大腸菌RNAポリメラーゼによる6文字の拡張DNAアルファベットの転写は、統一されたワトソン・クリック幾何学によって駆動される A unified Watson-Crick geometry drives transcription of six-letter expanded DNA alphabets by E. coli RNA polymerase
Juntaek Oh,Zelin Shan,Shuichi Hoshika,Jun Xu,Jenny Chong,Steven A. Benner,Dmitry Lyumkis & Dong Wang
Nature Communications Published:12 December 2023
DOI:https://doi.org/10.1038/s41467-023-43735-9
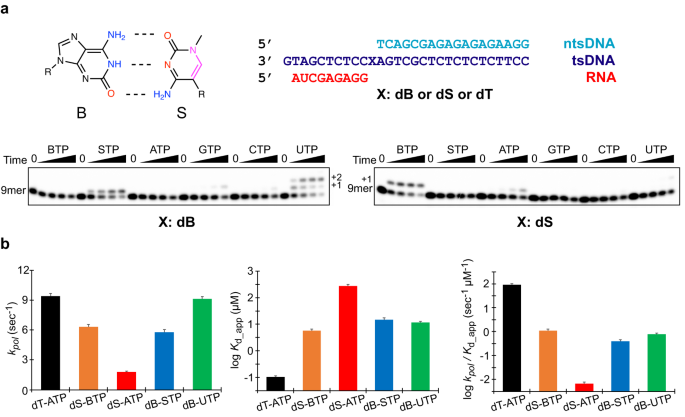
Abstract
Artificially Expanded Genetic Information Systems (AEGIS) add independently replicable unnatural nucleotide pairs to the natural G:C and A:T/U pairs found in native DNA, joining the unnatural pairs through alternative modes of hydrogen bonding. Whether and how AEGIS pairs are recognized and processed by multi-subunit cellular RNA polymerases (RNAPs) remains unknown. Here, we show that E. coli RNAP selectively recognizes unnatural nucleobases in a six-letter expanded genetic system. High-resolution cryo-EM structures of three RNAP elongation complexes containing template-substrate UBPs reveal the shared principles behind the recognition of AEGIS and natural base pairs. In these structures, RNAPs are captured in an active state, poised to perform the chemistry step. At this point, the unnatural base pair adopts a Watson-Crick geometry, and the trigger loop is folded into an active conformation, indicating that the mechanistic principles underlying recognition and incorporation of natural base pairs also apply to AEGIS unnatural base pairs. These data validate the design philosophy of AEGIS unnatural basepairs. Further, we provide structural evidence supporting a long-standing hypothesis that pair mismatch during transcription occurs via tautomerization. Together, our work highlights the importance of Watson-Crick complementarity underlying the design principles of AEGIS base pair recognition.


