2024-02-22 東京医科歯科大学,科学技術振興機構
ポイント
- 深層生成モデルを活用して空間トランスクリプトームデータから一細胞レベルでの共局在解析を行う手法、DeepCOLOR(ディープカラー)を開発。
- 多様な組織内で近接する細胞集団の同定や細胞間コミュニケーションの分子機構の推定が可能。
- 細胞間コミュニケーションの包括的な全体像の解明により、疾患の超早期段階からの予測、創薬標的の探索や治療法開発への応用が期待。
国立がん研究センター研究所 計算生命科学ユニットの小嶋 泰弘 独立ユニット長(東京医科歯科大学 難治疾患研究所 計算システム生物学分野 連携研究員)、東京医科歯科大学 難治疾患研究所 計算システム生物学分野の島村 徹平 教授(名古屋大学 大学院医学系研究科 システム生物学分野 特任教授)、名古屋大学 大学院医学系研究科 腫瘍病理学・分子病理学分野の三井 伸二 准教授、榎本 篤 教授、同 大学院医学系研究科 皮膚科学分野の秋山 真志 教授らの研究グループは、生体組織内の細胞間共局在関係を解析するための画期的な情報解析手法「DeepCOLOR」を開発しました。
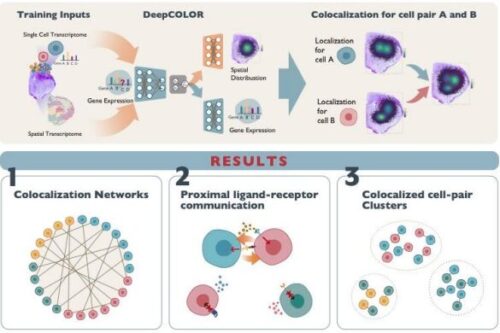
この手法は、一細胞トランスクリプトームデータと空間トランスクリプトームデータを統合し、深層生成モデルの枠組みを利用して、生体組織内の細胞間ネットワークを一細胞解像度で分析することを可能にする技術です。本解析手法をマウスの脳組織、ヒトの扁平上皮がんサンプル、SARS-CoV-2に感染したヒトの肺組織のデータに適用し、細胞間の共局在関係を網羅的に解析することにより、組織内で近接する細胞集団の同定や、細胞間コミュニケーションの分子機構の推定が可能となりました。
細胞間コミュニケーションの理解は、細胞応答や疾患、組織の生物学的機能研究において不可欠です。DeepCOLORは、一細胞トランスクリプトームデータを用いて、細胞の組織内空間分布を復元し、一細胞レベルの共局在解析を実現することに成功しました。本解析手法は、細胞間コミュニケーションの分子メカニズムに関する網羅的なデータに基づく仮説の提案を可能にし、疾患の超早期段階からの予測や新規の創薬標的の探索に役立つと期待されます。
本研究成果は、国際学術誌「Cell Systems」に2024年2月21日(米国東部時間)にオンライン掲載されます。
本研究は、日本学術振興会(JSPS) (課題番号20H04281、20K22839、22H04839、22H04925、23H04938c)、日本医療研究開発機構(AMED) (課題番号JP22ama221215、JP22ama221501、JP22ek0109488、JP23wm0325068、JP22wm0425007、JP23tm0424226)、科学技術振興機構(JST) ムーンショット型研究開発事業(目標2)(課題番号JPMJMS2025)および戦略的創造研究推進事業(課題番号JPMJAX20AB)、産業技術総合研究所(AIST) AI橋渡しクラウド(ABCI)、東京大学 ヒトゲノム解析センターのスーパーコンピュータシステム SHIROKANEなどの支援を受けて行いました。
<プレスリリース資料>
- 本文 PDF(295KB)
<論文タイトル>
- “Single-cell colocalization analysis using a deep generative model”
<お問い合わせ先>
<研究に関すること>
島村 徹平(シマムラ テッペイ)
東京医科歯科大学 難治疾患研究所 計算システム生物学分野
<JST事業に関すること>
犬飼 孔(イヌカイ コウ)
科学技術振興機構 ムーンショット型研究開発事業部
<報道担当>
東京医科歯科大学 総務部 総務秘書課 広報係
科学技術振興機構 広報課


