2024-02-29 京都大学アイセムス
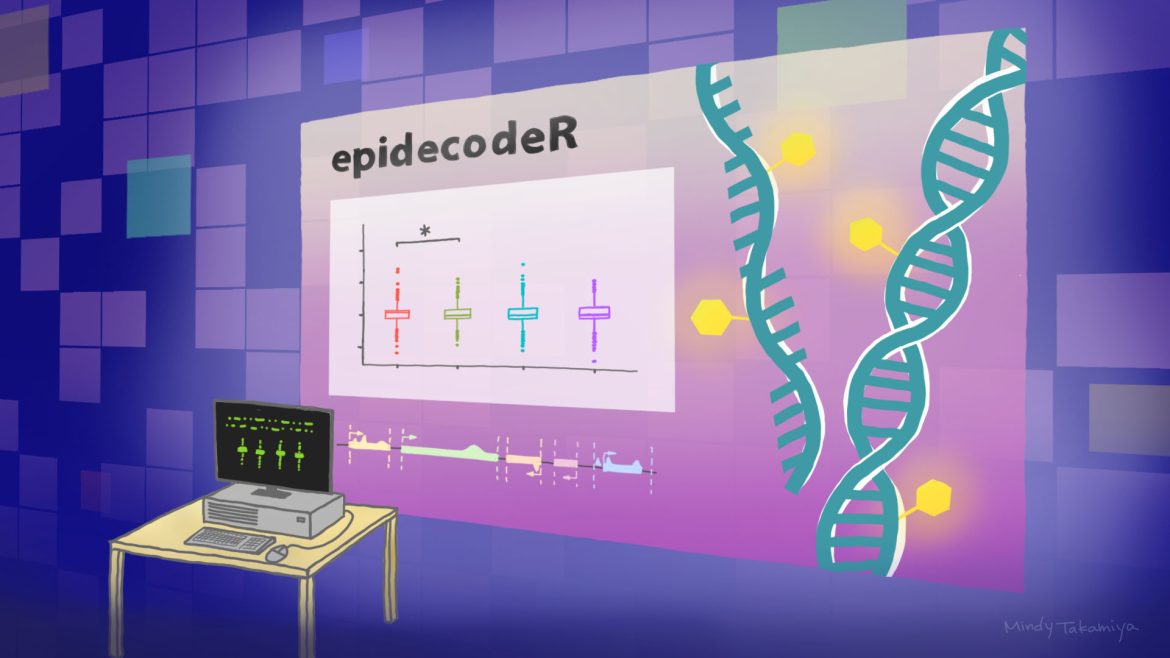
「epidecodeR」は複雑なエピゲノムとエピトランスクリプトームデータの解析を効率化できるツールで、遺伝子発現に及ぼすエピマークの影響を迅速かつ正確に予測することができます(©️高宮ミンディ/京都大学アイセムス)
現在、DNA および RNA 修飾の次世代解読技術によって、エピゲノムおよびエピトランスクリプトミクスの膨大なデータセットが生成されています。しかし、データに隠されている生命科学のメッセージを解明するツールが圧倒的に不足しているのが現状です。個々の核酸修飾(“エピマーク”)部位の同定、位置づけ、定量的比較を可能にするインフォーマティクスツールが存在する一方、特定のエピマークの働く仕組みとして、細胞における特定のプロセス文脈における遺伝子発現制御に結びつけるという本質的な課題はまだ達成されていません。その探索プロセスをより迅速なものにするために、京都大学アイセムス(高等研究院 物質―細胞統合システム拠点)の王丹客員教授らはプログラミング言語 R による epidecodeRを作成しました。epidecodeR を用いて、我々はヒストン脱アセチル化酵素や、アデノシンメチル化酵素・脱メチル酵素の操作による遺伝子発現変化との関係性を示すことに成功し、また脱メチル化酵素阻害剤の効果評価や、特定の脳部位におきる薬物依存により起きるヒストンタンパク質の関連修飾と遺伝子発現変化との結びつきを明らかにしました。
このような計算分析ツールはエピゲノムとエピトランスクリプトミクス制御を介した細胞制御メカニズムの解明や、病態評価、薬物機能の評価など、様々な生命や医療分野においてデータ解析ツールとして役に立つことが期待できます。
本研究は、Briefings In Bioinformatics に 1 月 23 日にオンライン公開されました。
詳しい研究成果について
エピゲノムとエピトランスクリプトームの 新たな解析ツール「epidecodeR」の開発
書誌情報
論文タイトル:“epidecodeR: A Functional Exploration Tool for Epigenetic and Epitranscriptomic Regulation”
著者:Kandarp Joshi, Dan Ohtan Wang