2024-09-20 東京大学
発表のポイント
- コレラ菌や緑膿菌を含む一部の病原性細菌と植物のオルガネラから、tRNAの新たなシチジン修飾、2-アミノバレラミジジン(ava2C)を発見しました。ava2C修飾はtRNAへのアミノ酸付加と、mRNA上のコドン認識に必須であり、タンパク質合成に欠かせない働きをしていることがわかりました。
- AUAコドンの認識に関わるシチジン修飾は他の生物種において二種類見つかっていますが、今回見つかったava2Cはそれらとは異なる修飾です。これは、生命活動の根幹を成す遺伝情報の読み取り方が生物ごとに微調整されていることを示す重要な発見です。
- 本成果は将来的に、病原菌のもつtRNA修飾を阻害することで増殖を抑えるような抗生物質の開発にも応用が期待できます。

植物オルガネラや一部のバクテリアではava2C修飾がmRNAのAUAコドンを認識する
発表概要
東京大学大学院工学系研究科の宮内健常特任研究員、秋山奈穂大学院生、鈴木勉教授らの研究グループと、Brigham and Women’s Hospitalの木村聡講師(研究当時、現:コーネル大学准教授)、Matthew K. Waldor教授の研究グループは、東京大学大学院工学系研究科の岡本晃充教授、理化学研究所生命機能科学研究センターの白水美香子チームリーダー、千葉大学大学院園芸学 研究院の相馬亜希子講師らと共同で、tRNA(注1)のアンチコドン(注2)から新規シチジン修飾である2-アミノバレラミジジン(ava2C)を発見し、クライオ電子顕微鏡(注3)を用いた構造解析により、リボソーム(注4)上でこの修飾がAUAコドン(注5)を解読するしくみを明らかにしました。
tRNAはタンパク質合成においてコドン(注6)を解読する重要な役割を担う短いRNA分子であり、多様な化学修飾が数多く施されることが知られています。特に、アンチコドン領域にはさまざまな修飾が見られ、tRNAがアミノ酸を受容するアミノアシル化(注7)や、リボソーム上において、mRNAのコドンを読み取る際に重要な役割を担うことで、正確で効率の良いタンパク質合成を可能にしています。
大腸菌を代表とする多くの細菌において、AUAコドンをイソロイシン(Ile)に解読するtRNA(tRNAIle2)のアンチコドン1字目(34位)にシチジン(C)の修飾体であるライシジン(L)が見つかっています(図1)。Lは、tRNAIle2がIleを受容し、AUAコドンを認識するための必須の修飾であることがわかっています。Lの修飾酵素はほぼすべての細菌に存在することから、Lは細菌に共通のtRNA修飾であると考えられてきました。一方で、アーキア(古細菌)ではtRNAIle2にアグマチジン(agm2C)(図1)と呼ばれる別のシチジン修飾が存在し、Lと同様の働きをしています。また、植物のオルガネラ(注8)であるミトコンドリアと葉緑体のtRNAIle2も何らかのシチジン修飾を利用してAUAコドンをIleに解読すると推定されてきましたが、その化学構造は不明でした。
本研究では、ホウレンソウの葉緑体およびミトコンドリアから単離したtRNAIle2に含まれる塩基修飾をRNA質量分析法(注9)により調べ、アンチコドンの34位に新たな修飾体を発見しました。この修飾はシチジンに5-アミノバレラミド(注10)という化合物が結合した化学構造をもつことがわかり、2-アミノバレラミジジン(ava2C)と命名しました(図1)。ava2Cは、長い側鎖をもつ点でLに類似していますが、側鎖の末端は異なる化学構造をもっています。

図1:AUAコドン解読に関わるtRNA 34位のシチジン修飾
コドン-アンチコドン対合の概要(左)とtRNAIle2の34位に見られるシチジン修飾の化学構造(右)。
他の生物についてもtRNAIle2の修飾を調べたところ、シロイヌナズナやタバコなどの高等植物、原始的な藻類である紅藻のほか、意外なことに緑膿菌やコレラ菌など一部の細菌にもava2Cが見つかりました。L同様、ava2CはtRNAIle2がIleを受容するために必要であり、さらにAUAコドンの解読に欠かせない修飾であることがわかりました。さらに、ava2CがAUAコドンの読み取りにどのように関わるかを調べるために、クライオ電子顕微鏡によりコドン-アンチコドン相互作用の構造を可視化しました。その結果、AUAコドン3字目のアデニンとava2Cが1本の水素結合を介した特殊な配向で対合すること、ava2Cの側鎖がこの対合の安定化に寄与することが示されました。生物種により異なるtRNA修飾を用いてコドン解読が微調整されていることが示唆されました。
さまざまなtRNA修飾によってタンパク質合成の精度や効率が制御されることが知られている一方、生物種ごとにどの修飾を用いてどのようにタンパク質合成の精度や効率を制御しているかについては未解明な部分が多いのが現状です。本研究は未知のRNA修飾レパートリーによるタンパク質合成制御機構の一端を明らかにした成果と言えます。また、コレラ菌や緑膿菌などの病原性細菌がava2Cをもつことから、新しい抗生物質の開発など創薬のターゲットとしても期待できます。
本研究成果は、2024年9月19日(英国夏時間)に「Nature Chemical Biology」に公開されました。
発表内容
〈研究の背景〉
すべての生物はゲノムDNAに塩基配列としてタンパク質のアミノ酸配列が記録されており、mRNAへの転写、タンパク質への翻訳という過程を経てタンパク質が合成されます。その際、mRNA上のコドンを読み取り、対応するアミノ酸を合成途中のタンパク質へ運ぶアダプター分子がtRNAです。tRNAは転写後に多くの化学修飾が施され、tRNA分子の安定性や機能が適切に調節されます。特に、mRNAのコドンと対合するアンチコドン領域にはさまざまな修飾が見られ、mRNA上のコドン解読能やアミノ酸の受容能を制御することで、正確で効率のよいタンパク質合成を可能にしています。近年の研究から、生物は種によって異なる修飾塩基をtRNAに付与することによりmRNA上のコドンを解読していることが明らかとなってきています。それぞれの生物種がどのような修飾塩基を用いてコドンを解読しているかを明らかにすることは生命の根幹を担う遺伝暗号解読の進化を考える上で非常に重要です。
大腸菌や枯草菌を含む多くの細菌においてAUAコドンをIleに解読するtRNA(tRNAIle2)のアンチコドン1字目(34位)には、シチジンの2位にリジンが結合した修飾体、ライシジン(L)(図1)が存在します。L修飾は、tRNAIle2のアミノ酸受容能をメチオニン(Met)からIleに変化させるとともに、コドン認識能をAUGからAUAに変えることで、AUAコドンの解読に必須な修飾であることがわかっています。当研究グループにより発見されたL修飾酵素であるTilS(注11)は、大腸菌や枯草菌において必須遺伝子にコードされており、L修飾は細菌の生存に欠かせないtRNA修飾であることがわかっています。
他のさまざまな細菌についてもtRNAIle2がL修飾を受けていると考えられてきましたが、枯草菌やマイコプラズマなどいくつかの細菌を除き、多くの生物種では、tRNAの修飾状態はほとんど調べられていませんでした。また、植物のオルガネラ、すなわち葉緑体とミトコンドリアにおいても、未知のtRNA修飾がAUAコドンをIleに解読すると考えられてきましたが、その化学構造は同定されていませんでした。アーキア(古細菌)では、tRNAIle2にアグマチジン(agm2C)(図1)と呼ばれる別のシチジン修飾が当研究グループにより発見され、Lと同様の働きをしていることが明らかとなっています。
そこで本研究では植物の葉緑体、およびミトコンドリアにおけるtRNAIle2の修飾塩基の構造および機能を明らかにすることを目指しました。
〈研究の内容〉
宮内健常特任研究員らは、ホウレンソウから葉緑体tRNAIle2を単離精製し、RNA質量分析法により修飾を調べたところ、分子量341の未知の修飾体N341を発見しました。同じN341修飾が、ホウレンソウ、シロイヌナズナの葉緑体とミトコンドリアそれぞれのtRNAIle2、原始的な藻類である紅藻シアニディオシゾンの葉緑体tRNAIle2にもあることが判明しました。紅藻の培養は相馬亜希子講師らと共同で行われました。
木村聡講師(当時)らは独立にコレラ菌tRNAの解析において分子量341の修飾体を見つけ、両研究グループは共同で、この修飾は植物で見つかったN341と同一であることを確認しました。安定同位体のリジンを用いた代謝ラベル実験や、単離したN341ヌクレオシドを用いた重水素置換実験などからN341の化学構造をシチジンの2位に5-アミノバレラミドが結合した構造と推定しました。この推定候補化合物の化学合成を岡本晃充教授らのグループが行い、植物で見つかったN341と比較したところ、天然のN341と推定化合物が同一であることが判明し、N341を2-アミノバレラミジジン(ava2C)と命名しました(図1)。ava2Cは、長い側鎖をもつ点でLに似ていますが、側鎖の末端構造が異なっています。
進化的に離れた生物種である植物と細菌において同一のtRNA修飾が見出されたことは興味深く、ava2C修飾がこれら生物種において共通の役割を担っていることを示唆しています。
次に、AUAコドンをIleに解読する際のava2C修飾の役割を明らかにしました。まず、未修飾シチジン、L、ava2Cをそれぞれ導入したtRNAIle2を用意し、tRNAがイソロイシルtRNA合成酵素(注12)によってIleを受容する効率を測ったところ、ava2Cを導入したtRNAは、Lを導入したtRNAと同程度の高い効率でIleを受容することが判明しました(図2A)。次に、これら修飾状態の異なるtRNAIle2がリボソーム上でAUAコドンを認識する効率を試験管内で測定したところ、ava2Cを導入することでtRNAIle2はAUGコドンに結合しづらくなり、AUAコドンに特異的かつ効率よく結合するようになりました(図2B)。さらに、細胞内tRNAIle2のava2C修飾を培地中のアラビノース濃度で制御できるように設計したコレラ菌を用い、連続したAUAコドンを含むレポーター遺伝子を翻訳させました。その結果、アラビノース濃度を下げ、ava2C修飾率を低下させた際に、AUAコドンを含むレポーター遺伝子の発現量が顕著に低下しました(図2C)。
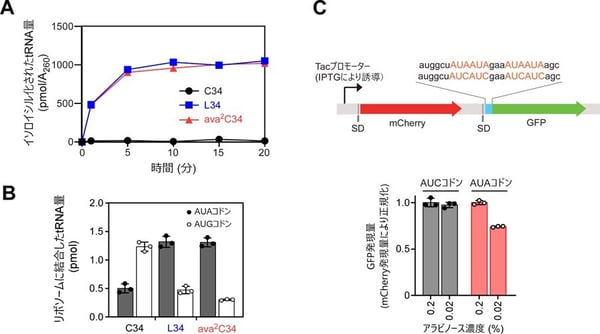
図2:ava2CはAUAコドンの正確かつ安定な読み取りを支えている
(A)異なる34位修飾をもつtRNAの試験管内におけるイソロイシル化効率。
(B)異なる34位修飾をもつtRNAのリボソーム上におけるAUA/AUGコドンへの結合量(n=3)。
(C)レポーター遺伝子のデザイン(上)とコレラ菌でのAUA/AUCコドンの翻訳効率(下)。AUAコドンまたはAUCコドンを遺伝子配列の上流に含むGFPの発現量を、mCherryの発現量で正規化した(n=3)。
これらの試験管内および細胞内の実験により、ava2C修飾はtRNAIle2のアミノアシル化およびAUAコドンの解読に必須であることが明らかになりました。
さらにクライオ電子顕微鏡を用いた構造解析により、ava2Cを含むtRNAIle2のアンチコドンがmRNA上のAUAコドンとどのように対合するかを原子レベルで解明しました(図3)。

図3:AUAコドンを読み取るava2Cの構造
(A)tRNAIle234位のava2CとAUAコドン3字目のアデニン(A3)の塩基対合。水素結合を赤点線、その距離を赤字で示した。Å(読み:オングストローム)は10-10m。
(B)ava2C34を取り巻くmRNA(グレー)と16SリボソームRNA(黄)の配置。
(C)ava2C側鎖がmRNAとリボソームRNAの狭間にはまり込む様子。
その結果、tRNAIle2 34位のava2CはAUAコドン3字目のアデニン塩基とたった1本の水素結合を介して対合していることがわかりました(図3A)。一般的に、RNAの塩基対合は2本または3本の水素結合を形成しますが、ava2C34-A3塩基対はそれらと異なる特徴的な対合様式です。さらにava2C34の周辺の構造を詳しく見てみると、ava2C側鎖はmRNAの下流(3’方向)に伸び、AUAコドンの3字目に隣接した4番目のmRNA残基と追加の水素結合を作ることが示されました(図3B)。また、ava2Cの側鎖がmRNAとリボソームRNAの作る溝にぴったりと収まることで、疎水性相互作用(注13)やファン・デル・ワールス相互作用(注14)が生じることが示唆されました(図3C)。こうしてava2C側鎖がAUAコドン以外の残基とのさまざまな相互作用を仲立ちすることで、1本の水素結合しか持たない不安定なava2C34-A3塩基対を安定化し、AUAコドンを効率よく読めるようにしていると考えられます。
今回、ava2C側鎖の全長にわたって密度を観察することができました(図3A)。AUAコドンを認識するLの構造を明らかにした当グループの先行研究[Akiyama et al., Nature Struct Mol Biol., 31, 817–825 (2024)]ではL側鎖の全長を観察できなかったことから、Lよりもava2Cの側鎖の方が、周囲との相互作用でより強く固定されていると言えます。また、ava2C側鎖末端は電荷を持たないアミド基であり、L側鎖より化学的に不活性かつ疎水的です。したがって、ava2Cは、Lよりも安定かつAUAコドンの解読をより強くサポートする修飾塩基として、一部の生物が進化の過程で獲得したものと考えられます。
〈今後の展望〉
本研究は、それぞれの生物がさまざまな化学修飾を駆使することで正しい遺伝暗号解読を実現していることを示しています。多くの生物種におけるtRNAの化学修飾を用いた遺伝暗号の解読機構には未知なことが多く、さらなる研究が必要です。ava2C修飾はコレラ菌や緑膿菌などの病原性細菌からも見つかったことから、tRNA修飾を阻害することでコレラ菌や緑膿菌の増殖を抑えるような新たな抗生物質の開発にも応用が期待できます。また、tRNAは新たな創薬モダリティとしても脚光を浴びており、本研究成果は将来的に、tRNAの機能を調節するためのツールとして、創薬研究にも利用できる可能性を秘めています。
発表者・研究者等情報
東京大学大学院工学系研究科 化学生命工学専攻
宮内 健常 特任研究員
秋山 奈穂 博士課程/日本学術振興会特別研究員
鈴木 勉 教授
Brigham and Women’s Hospital
木村 聡 研究当時:講師、現:コーネル大学准教授
Matthew K. Waldor 教授
論文情報
雑誌名:Nature Chemical Biology
題 名:A tRNA modification with aminovaleramide facilitates AUA decoding in protein synthesis
著者名:Kenjyo Miyauchi+, Satoshi Kimura+, Naho Akiyama+, Kazuki Inoue, Kensuke Ishiguro, Thien-Son Vu, Veerasak Srisuknimit, Kenta Koyama, Akiko Soma, Asuteka Nagao, Mikako Shirouzu, Akimitsu Okamoto, Matthew K. Waldor*, Tsutomu Suzuki*
+ Equal contributions, * Corresponding authors
DOI:10.1038/s41589-024-01726-x
研究助成
本研究は、日本学術振興会JSPSの基盤研究(S)「RNAエピジェネティックスと高次生命現象」(代表:鈴木勉、26220205)、基盤研究(S)「RNA修飾の変動と生命現象」(代表:鈴木勉、18H05272)、特別研究員奨励費「原核生物におけるAUAコドン解読の分子基盤」(代表:秋山奈穂、23KJ0409)、および科学技術振興機構(JST) 戦略的創造研究推進事業 ERATO「鈴木RNA修飾生命機能プロジェクト」(研究総括:鈴木勉、JPMJER2002)などの支援を受けて実施されました。
用語解説
(注1)tRNA
Transfer RNA(転移RNA)。タンパク質合成において、コドン(特定の3つの塩基配列)とアミノ酸を1対1で対応させるアダプター分子として働く。70〜90塩基長の短い一本鎖RNAで、特徴的なクローバー葉様の二次構造をとり、それが折りたたまれてL字型の立体構造をとる。tRNAは3’末端に自身に対応した特定のアミノ酸を受容し、20種類のアミノ酸それぞれに、対応するtRNAが複数種類存在する。tRNAはコドンに対応した3塩基からなるアンチコドンを持ち、リボソーム上でmRNA(伝令RNA)上のコドンと結合し、そのコドンに対応するアミノ酸を伸長中のタンパク質へと導入する。
(注 2)アンチコドン
tRNAの34~36位の3つの塩基から構成され、リボソーム上で対応するコドンと対合することで遺伝暗号を解読し、対応するアミノ酸を導入する。
(注 3)クライオ電子顕微鏡
生体分子の試料に低温下(約-200℃)で電子線を照射し、その構造を観察できる電子顕微鏡。試料を水溶液中で瞬間凍結することで、生体内に近い環境で目的分子の構造解析を行うことができる。
(注 4)リボソーム
タンパク質合成の場である、3ないし4種のRNAと多数のタンパク質から構成される巨大な複合体。大小2つのサブユニットからなり、大サブユニットはペプチジル転移反応を、小サブユニットはmRNAとtRNA間のコドンーアンチコドン対合を監視するといった重要な役割をもつ。
(注 5)AUAコドン
コドンの一種。この記事で登場する細菌、アーキア、植物オルガネラのAUAコドンはIleを指定する。一方、隣のコドンであるAUGはMet(メチオニン)を指定する。
(注 6)コドン
遺伝暗号の基本単位。RNAの4種類ある塩基(アデニン、グアニン、ウラシル、シトシン)が3つずつの組となり、64通りのコドンが定義される。標準的な遺伝暗号では、このうち61個が20種類のアミノ酸に対応し、3個のコドンがタンパク質合成の終結を指定する。
(注 7)アミノアシル化
tRNAの末端にアミノ酸を結合させる生体反応。アミノ酸の種類ごとに専属のアミノアシルtRNA合成酵素が存在し、特定のtRNAに対して正しい種類のアミノ酸を結合させることで、コドンとアミノ酸の対応づけが厳密に制御されている。
(注 8)オルガネラ
細胞内小器官。核、ミトコンドリア、葉緑体、小胞体、ゴルジ体など、細胞内の構造物のこと。
(注9)RNA質量分析法
質量分析によりRNA分子を解析する手法。さまざまな核酸消化酵素を用いてRNAをヌクレオシドモノマー、あるいは短い断片に分解し、液体クロマトグラフィーで分離しつつ質量分析を行う。得られた質量電荷比(m/z)から精密な分子量がわかり、修飾構造の決定や修飾率の推定を行うことができる。
(注10)5-アミノバレラミド
分子式C5H12N2Oで示される物質で、5-アミノペンタンアミド、5-アミノ吉草酸アミドとも呼ばれる。さまざまな生物から微量な代謝物として検出された例が知られるが、生成、代謝経路については一部の細菌でしか判明していない。
(注11)TilS
tRNA(Ile)ライシジン合成酵素のこと。2003年に当研究グループが以下の論文で発見したイソロイシンtRNAにL修飾を導入する酵素。ほぼすべての細菌が持ち、大腸菌や枯草菌では必須遺伝子にコードされている。Soma et al., Mol Cell. 2003 Sep;12(3):689-98. doi:10.1016/s1097-2765(03)00346-0.
(注12)イソロイシルtRNA合成酵素
tRNAIleを特異的に認識し、その3’末端にIleをアシル結合させる反応を触媒する。
(注13)疎水性相互作用
水溶液中に存在する疎水性分子が水を避けて互いに引き合うことで、溶媒との接触面を少なくし安定化する作用。
(注14)ファン・デル・ワールス相互作用
分子同士の相互作用のうち、双極子同士の間に生じる引力性の力。
プレスリリース本文:PDFファイル
Nature Chemical Biology:https://www.nature.com/articles/s41589-024-01726-x