2025-10-29 ロックフェラー大学
<関連情報>
- https://www.rockefeller.edu/news/38513-major-milestone-achieved-in-capturing-ribosome-assembly/
- https://www.nature.com/articles/s41586-025-09688-3
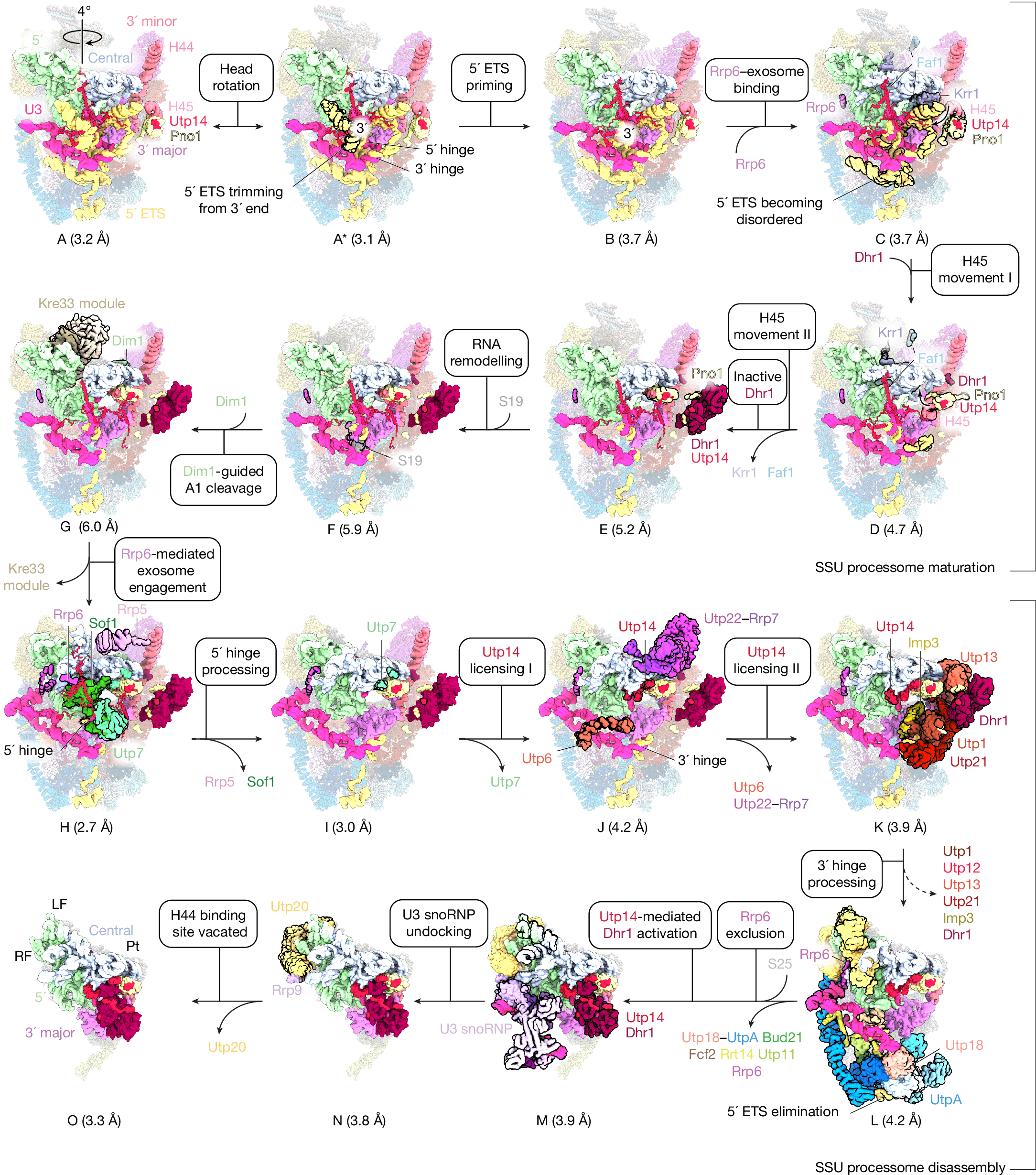
ヘリカーゼを介したSSUプロセソームの成熟と分解のメカニズム Helicase-mediated mechanism of SSU processome maturation and disassembly
Olga Buzovetsky & Sebastian Klinge
Nature Published:29 October 2025
DOI:https://doi.org/10.1038/s41586-025-09688-3
Abstract
Eukaryotic ribosomal small subunit (SSU) assembly requires the SSU processome, a nucleolar precursor containing the RNA chaperone U3 small nucleolar RNA (snoRNA). The underlying molecular mechanisms of SSU processome maturation, remodelling, disassembly and RNA quality control, and the transitions between states remain unknown owing to a paucity of intermediates1,2,3. Here we report 16 native SSU processome structures alongside genetic data, revealing how two helicases, the Mtr4-exosome and Dhr1, are controlled for accurate and unidirectional ribosome biogenesis. Our data show how irreversible pre-ribosomal RNA degradation by the redundantly tethered RNA exosome couples the transformation of the SSU processome into a pre-40S particle, during which Utp14 can probe evolving surfaces, ultimately positioning and activating Dhr1 to unwind the U3 snoRNA and initiate nucleolar pre-40S release. This study highlights a paradigm for large dynamic RNA–protein complexes in which irreversible RNA degradation drives compositional changes and communicates these changes to govern enzyme activity while maintaining overall quality control.

