2018-06-06 理化学研究所,東京大学,京都大学,科学技術振興機構,内閣府政策統括官(科学技術・イノベーション担当)
ポイント
理化学研究所(理研)開拓研究本部新宅マイクロ流体工学理研白眉研究チームの新宅博文理研白眉研究チームリーダー、マハメッド・ナディ・アブデルモエズ研修生、東京大学大学院理学系研究科の小口祐伴特任助教、上村想太郎教授、京都大学大学院医学研究科飯田慶特定助教らの共同研究グループ※は、一つの細胞から核RNA[1]と細胞質RNA[1]を分画して、それぞれの遺伝子発現を解析できるマイクロ流体技術を基盤とする「1細胞RNA分画解読法(SINC-seq法注1))」を開発しました。
本研究成果は、遺伝子発現制御の理解を通じて細胞生物学の研究を加速し、将来的には、遺伝子治療や創薬、微生物産業などへの応用展開が期待できます。 近年、細胞の多様性を理解するために「1細胞RNA-seq法[2]」が用いられています。 RNAは核内で発現した後、細胞質に移動してタンパク質に翻訳されるまでにさまざまな修飾を受けますが、これまで、1細胞から核RNAと細胞質RNAに分離して、網羅的に遺伝子発現を解析する技術はありませんでした。
共同研究グループは、マイクロ流路における電場と流れを制御して、1細胞から核RNAと細胞質RNAを分離して並列に解読解析するSINC-seq法を開発しました。そして、本手法を用いて一つの細胞内のRNAの局在や遺伝子発現の相関を解析できることを実証しました。さらに、これらが、細胞周期[3]やRNAスプライシング[4]などの生命機能と密接に関わっていることを示しました。
本研究成果は、英国の科学雑誌『Genome Biology』のオンライン版(6月6日付け:日本時間6月6日)に掲載されます。
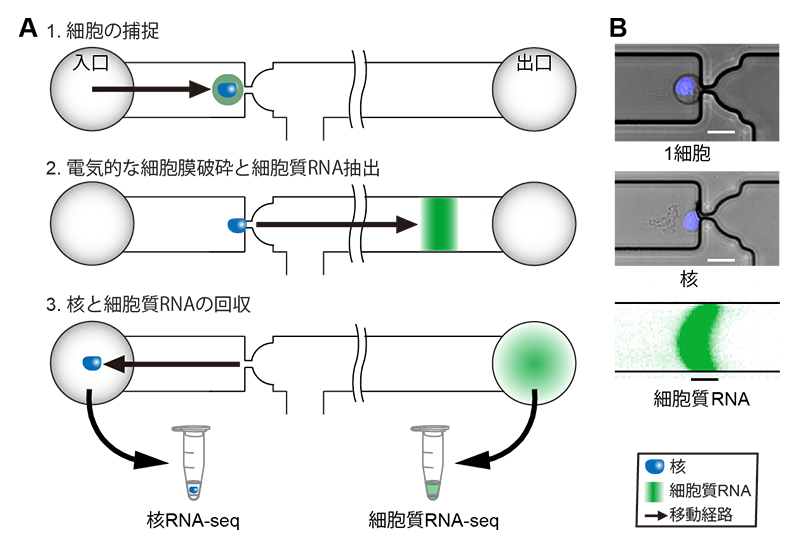
図 SINC-seq法の概要
※共同研究グループ
理化学研究所 新宅マイクロ流体工学理研白眉研究チーム
理研白眉研究チームリーダー 新宅 博文(しんたく ひろふみ)
研修生 マハメッド・ナディ・アブデルモエズ(MahmoudNadyAbdelmoez)
(京都大学大学院工学研究科博士後期課程)
東京大学大学院理学系研究科 生物科学専攻
特任助教 小口 祐伴(おぐち ゆうすけ)
教授 上村 想太郎(うえむら そうたろう)
京都大学大学院医学研究科
特定助教 飯田 慶(いいだ けい)
筑波大学医学医療系血液内科
准教授 錦井 秀和(にしきい ひでかず)
スタンフォード大学
教授 ワン・ジー・サンティアゴ(JuanG.Santiago)
京都大学大学院工学研究科
准教授 横川 隆司(よこかわ りゅうじ)
教授 小寺 秀俊(こてら ひでとし)
※研究支援
本研究は、内閣府革新的研究開発推進プログラム(ImPACT)「セレンディピティの計画的創出による新価値創造(合田圭介プログラム・マネージャー)」の支援を受けて行われました。また、本研究の一部は日本学術振興会(JSPS)科学研究費補助金 挑戦的萌芽研究「単一細胞のRNAおよびDNA同時抽出・解析技術の開発(研究代表者:新宅博文)」および基盤研究(B)「マイクロ・ナノ電気穿孔法による細胞核への分子導入法の開発(研究代表者:新宅博文)」の支援を受けて行われました。
注1) Single cell Integrated Nuclear and Cytoplasmic RNA-seq法
背景
近年、「1細胞RNA-seq法」により一つの細胞の中に存在するRNAを網羅的に計数することが可能になり、細胞ごとの遺伝子発現を詳細に理解できるようになりました。しかし、1細胞RNA-seq法では、解析対象となる1細胞を単離することが必要不可欠であるため、単離が難しい脳神経細胞などでは、細胞の代わりに単離した核を用いる「核RNA-seq法」が使われ始めています。
核RNAと細胞質RNAは、RNAの局在などにより異なる発現パターンを持つ可能性が複数の細胞を利用した解析から知られています。そのため、核RNA-seq法を1細胞RNA-seq法の代用として利用する方法の妥当性は不明でした。また、核RNAと細胞質RNAの発現を1細胞レベルかつ網羅的に比較することで、RNAスプライシングなどの遺伝子発現調節機構に関して新たな知見が得られると期待されていました。
しかしこれまで、1細胞から核RNAと細胞質RNAを分離して網羅的に遺伝子発現を解析する技術はありませんでした。加えて、1細胞に含まれるRNA量は10ピコグラム(pg、1pgは1兆分の1グラム)程度と非常に少量であることも、核RNAと細胞質RNAを分離して別々に解析するための技術的障壁の一つでした。
研究手法と成果
共同研究グループは、マイクロ流体工学と生命科学の融合により、核と細胞質分子を正確に分離するシステムを構築し注2)、一つの細胞から核RNAと細胞質RNAを並列に解読解析することを可能にしました(図1)。
このマイクロ流体システムは、マイクロ流路内に1細胞を捕捉するオリフィス構造(流体を流す小さな穴)を持っています。細胞補足後に外部電場を加えることで、オリフィス構造の近くに形成される集中電場により細胞膜が選択的に破砕され、細胞質RNAが効率的に抽出され、核と細胞質RNAが正確に分画されます。本手法は新宅理研白眉研究チームリーダーが2014年に開発した手法注3、4)と比較すると、加える電圧をおよそ20分の1程度に下げることができます。このことにより、装置の簡略化、電気伝導性の高い細胞懸濁液の使用などの選択肢が広がりました。また、分離した核と細胞質RNAはマイクロ流体システムから別々に回収し、これらを独立した分析にかけ、遺伝子の局在を網羅的に知ることができます(図2A)。
さらに、1例として核RNAと細胞質RNAの並列解読解析を実施し、それぞれの遺伝子発現パターンに見られる相関性の全貌を1細胞レベルで調べました。その結果、遺伝子発現データから、核RNAと細胞質RNAの発現はおおむね相関しており、核RNA-seq法による1細胞解析の妥当性が示されました。さらに、相関性が比較的高い群は細胞周期に関わる遺伝子を多く含むことが分かりました。一方で、明確な相関性が観察されない、あるいは逆相関する遺伝子群も一定数存在し、それらがRNAスプライシングなどの特徴的な機能を持つ可能性が示されました(図2B)。
注2)新宅博文,藁谷卓哉,上村想太郎,小口祐伴,生体高分子分画用チップ、それを用いた生体高分子の分画方法、および生体高分子の分析方法, 特許第6338262号
注3)Juan G. Santiago, Hirofumi Shintaku, Simultaneous Extraction and Separation of RNA and DNA from Single Cells Using Electrophoretic Techniques, US 14/593,925 (2015/1/9), PCT/US2015/010884(2016/4/7)
注4)Hirofumi Shintaku, Hidekazu Nishikii, Lewis A. Marshall, Hidetoshi Kotera, and Juan G. Santiago, On-Chip Separation and Analysis of RNA and DNA from Single Cells, Analytical Chemistry, Vol. 86, No. 4 (2014), pp 1953–1957.
今後の期待
従来の1細胞RNA-seq法に加えて今回開発したSINC-seq法を利用することにより、核と細胞質間でのRNA輸送を含む遺伝子発現の転写後制御の理解が進み、将来的には、それらの知見をもとに遺伝子発現を効果的に制御することで、遺伝子治療や創薬、微生物産業などへ応用展開されることが期待できます(図3)。また、本技術による分離対象の必要要件は、溶液中で電荷を持つことだけであるため、細胞内のRNA以外の分子や細胞内小器官の分離・分析技術の基盤となる可能性があります。
合田圭介プログラム・マネージャーのコメント
本成果は、ImPACT「セレンディピティの計画的創出による新価値創造」に参画する新宅チームによるものです。本チームでは、膨大な細胞集団から分取した目的の細胞一つ一つを、より詳細に解析するための技術を開発しています。今回の成果は、世界で初めて、一つの細胞を複数の分画に分けて遺伝子発現を解析することに成功したものです。本技術は、1細胞を捉えて細胞内の機能、特に遺伝子の発現制御メカニズムを理解するための新たな情報を得ることができるため、本プログラムが目指す希少で貴重な細胞の分取と解析において大きな役割を果たすものと期待されます。
原論文情報
Mahmoud N. Abdelmoez, Kei Iida, Yusuke Oguchi, Hidekazu Nishikii, Ryuji Yokokawa, Hidetoshi Kotera, Sotaro Uemura, Juan G. Santiago, and Hirofumi Shintaku, “SINC-seq: correlation of transient gene expressions between nucleus and cytoplasm reflects single-cell physiology”, Genome Biology, 10.1186/s13059-018-1446-9
発表者
理化学研究所
主任研究員研究室 新宅マイクロ流体工学理研白眉研究チーム
理研白眉研究チームリーダー 新宅 博文(しんたく ひろふみ)
研修生 マハメッド・ナディ・アブデルモエズ(Mahmoud Nady Abdelmoez)
(京都大学大学院工学研究科博士後期課程)
東京大学大学院理学系研究科 生物科学専攻
特任助教 小口 祐伴(おぐち ゆうすけ)
教授 上村 想太郎(うえむら そうたろう)
京都大学大学院医学研究科
特定助教 飯田 慶(いいだ けい)
報道担当
理化学研究所 広報室 報道担当
東京大学 大学院理学系研究科・理学部 広報室
京都大学 総務部 広報課 国際広報室
科学技術振興機構 広報課
ImPACT事業に関すること
内閣府 革新的研究開発推進プログラム担当室
ImPACTプログラム内容およびPMに関すること
科学技術振興機構 革新的研究開発推進室
補足説明
-
- RNA
- ゲノムは細胞が持つ全遺伝情報であり、DNA(デオキシリボ核酸)という生体高分子に4種類の塩基の配列として記録されている。ゲノムの中の遺伝情報が記録(コード)された領域が遺伝子である。遺伝子領域の情報は、DNAを鋳型としてRNA(リボ核酸)が合成(転写)されることで読み出される。
-
- 1細胞RNA-seq法
- 1細胞中に含まれるRNAを、ハイスループットDNAシーケンサーを用いて解読(塩基配列決定)し、網羅的かつ定量的にその量や種類を決定する方法。
-
- 細胞周期
- 細胞は、分裂を繰り返して増殖するが、この細胞分裂のサイクルを細胞周期と呼ぶ。細胞周期は、間期とM期に分けられる。分裂が起こるM(Mitosis)期と、DNAの複製が起こるS(Synthesis)期、それぞれの間をつなぐG1(Gap1)期、G2(Gap2)期からなり、サイクルはG1→S→G2→M→G1→…の順に進む。
-
- RNAスプライシング
- ゲノム上の遺伝子をメッセンジャーRNA(mRNA)に転写する過程の一つ。ゲノム上の遺伝子はエクソンとイントロンからなるが、mRNAが形成される過程でイントロン配列が削除され、エクソン配列のみがmRNAとして残る。

図1 SINC-seq法の概要
A)本研究で開発したSINC-seqの概要。マイクロ流体技術で1細胞を核と細胞質RNAに分画し、それぞれをRNA-seqで解析。マイクロ流路(幅50マイクロメートル(μm、1μmは100万分の1m)、深さ25μm)に設けたオリフィス構造に細胞を捕捉し、電気的な細胞膜の破砕と細胞質RNAの抽出、核との分画を可能にする。分画後はそれぞれのサンプルを回収してさまざまな分析に利用できる。
B)上から順に、オリフィスに捕捉した1細胞、細胞質RNA抽出後の核、等速電気泳動法により抽出された細胞質RNA。スケールバーは20μm。

図2 SINC-seq法により得られた並列RNA-seqの結果の1例
A)核(左)と細胞質(右)における遺伝子局在解析。ミトコンドリア遺伝子群は細胞質に局在していることから、分画が成功していることを示している。
B)核と細胞質における遺伝子発現変動の相関解析。相関の強さに従って遺伝子を並び替え(順位をGene rankとして表記)、相関の強い遺伝子群(縦軸1~0.3)と逆相関の強い遺伝子群(-0.4~ -0.3)を抽出した。pは帰無仮説の有位確率。多くの遺伝子で相関係数が正であることから核RNAと細胞質RNAの発現がおおむね相関していることが分かる。
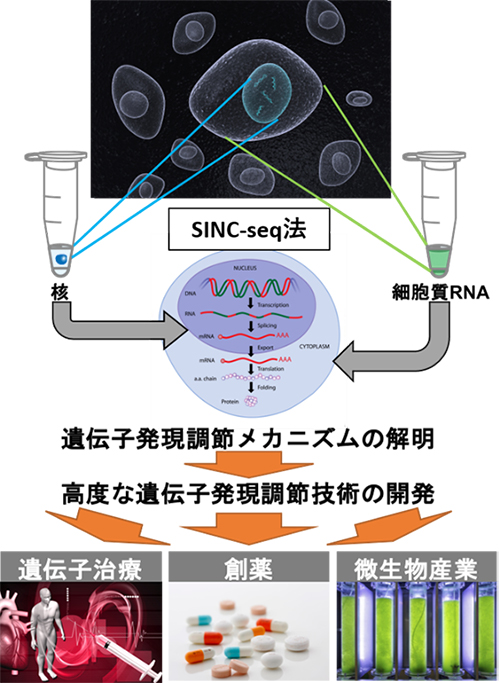
図3 SINC-seq法に利用による今後の進展
遺伝子発現制御の理解を通じて細胞生物学の研究を加速し、将来的には、遺伝子治療や創薬、微生物産業などへの応用展開が期待できる。