心房細動の詳細な発症メカニズムの解明に貢献
2018-06-12 理化学研究所,東京医科歯科大学,日本医療研究開発機構
要旨
理化学研究所(理研)統合生命医科学研究センターの久保充明副センター長(研究当時)、統計解析研究チームの鎌谷洋一郎チームリーダー(研究当時)、ロー・シュー・キー客員研究員(研究当時)、循環器疾患研究チームの伊藤薫チームリーダー(研究当時)、東京医科歯科大学大学院の田中敏博教授らの国際共同研究グループ※は、大規模ゲノムワイド関連解析(GWAS)[1]を通じて、心房細動の70個の新しい疾患感受性座位[2]の同定に成功しました。
心房細動は、重症な脳梗塞の原因となる重要な不整脈です。心房細動の環境要因については、疫学研究で十分に検討されてきましたが、遺伝学的要因については、2017年に理研を含む国際共同研究グループにより21個の感受性座位が発表され、合計30個の感受性座位が判明していました注1、2)。しかし、他の循環器疾患である虚血性心疾患(心筋梗塞など)では100個以上の疾患感受性座位が同定されており、それに比べて研究が進んでいませんでした。
それを克服するため、国際共同研究グループは日本人を含む多人種の心房細動患者65,446人と対照者50万人に対してGWASを行い、多数の新しい心房細動の感受性座位を発見しました。その後、左心房のトランスクリプトーム解析[3]を含むオミクス解析[4]を行うことにより、これら感受性座位の遺伝子多型[5]が作用する57個の遺伝子を同定しました。これら遺伝子群は、心臓の分化形成や電気生理学的機能、心筋収縮・形態形成に関わるものであり、心房細動発症の分子メカニズムの全体像を示しています。
今後、本成果を手掛かりに心房細動発症の詳細なメカニズムの解明や、メカニズムに関連した分子ターゲットの発見によって疾患に効果的な創薬につながるものと期待できます。本研究成果は、国際科学雑誌『Nature Genetics』オンライン版(6月11日付け:日本時間6月12日)に掲載されます。
本研究で使用した日本人集団のジェノタイプデータは、科学技術振興機構(JST)バイオサイエンスデータベースセンター(NBDC)を通じて既に公開されています注3)。
なお本研究は、文部科学省「オーダーメイド医療実現化プロジェクト」および日本医療研究開発機構(AMED)「オーダーメイド医療の実現プログラム」の支援を受けて行われました。
- 注1)
- 2017年4月18日プレスリリース「心房細動に関わる新しい遺伝子マーカーを同定」
- 注2)
- Christophersen IE, Rienstra M, Roselli C et al. Large-scale analyses of common and rare variants identify 12 new loci associated with atrial fibrillation. Nat Genet. 49:946-952, 2017
- 注3)
- バイオサイエンスデータベースセンター(NBDC)
※国際共同研究グループ
- 理化学研究所 統合生命医科学研究センター
- 副センター長(研究当時)久保 充明(くぼ みちあき)
- 統計解析研究チーム
- チームリーダー(研究当時)鎌谷 洋一郎(かまたに よういちろう)
(現 理化学研究所 生命医科学研究センター 統計解析研究チーム チームリーダー、京都大学大学院医学研究科 ゲノム医学センター 准教授) - 客員研究員(研究当時)ロー・シュー・キー(Low Siew Kee)
(現 理化学研究所 生命医科学研究センター 統計解析研究チーム 客員研究員、がん研究会 がん研究所 がんプレシジョン医療研究センター グループリーダー) - 循環器疾患研究チーム
- チームリーダー(研究当時)伊藤 薫(いとう かおる)
(現 理化学研究所 生命医科学研究センター 循環器疾患研究チーム チームリーダー) - 東京医科歯科大学大学院 医歯学総合研究科 疾患多様性遺伝学分野
- 教授 田中 敏博(たなか としひろ)
- ブロード研究所/マサチューセッツ総合病院
- ディレクター パトリック・エリナー(Patrick T. Ellinor)
- 研究員 カロリナ・ロセリニ(Carolina Roselli)
背景
心房細動は最も一般的に認められる不整脈であり、心不全や脳梗塞など重篤な病態を引き起こす場合もある重要な疾患です。心房細動発症の環境要因については、疫学研究によって十分検討されてきました。
一方、遺伝学的要因については、2012年に理研を含む国際共同研究グループにより大規模な研究が行われ、6個の疾患感受性座位が同定され注4)、 2017年までに合計30個の感受性座位が報告されました 。しかし、他の循環器疾患である虚血性心疾患(心筋梗塞など)では、100以上の感受性座位が同定されています。それに比べて、心房細動ではその疾患の重要性にもかかわらず研究が進んでいませんでした。
そこで、国際共同研究グループはこれまでの多数の心房細動研究を集め、50万人以上のサンプルを用いた大規模解析を試みました。
研究手法と成果
国際共同研究グループは、これまで世界中で行われた50以上の心房細動研究を集めることにより、日本人を含む多人種の心房細動患者65,446人(欧米人84.2%、日本人12.5%、アフリカ系アメリカ人2%、ブラジル人とヒスパニック系1.3%)と対照者約50万人に対して、大規模ゲノムワイド関連解析(GWAS)を行いました。このとき、過去の研究の多くが対象としてきたアレル頻度[6]5%以上の8,328,530 の頻度が多く一般的な一塩基多型(SNP)[5]だけでなく、アレル頻度1~5%の2,884,670 SNP、また疾患発症に与える影響がより大きいと考えられるアレル頻度1%以下の936,779 SNPも解析しました。
この多人種メタ解析[7]の結果、67個の感受性座位を同定しました。ほとんどのマーカーは、アレル頻度5%以上で相対危険度[8]が1.04から1.49の間にありました。アレル頻度5%以下の二つの低頻度マーカーをC1orf185遺伝子、UBE4B遺伝子領域内に発見しました。さらに、GSEA解析[9]の結果から55の生物学的パスウェイ[10]が心房細動発症に関与していることが明らかになりました。これらのパスウェイは心臓の分化形成、電気生理学的機能、心筋細胞収縮、心臓形態形成に関係するものでした(図1)。
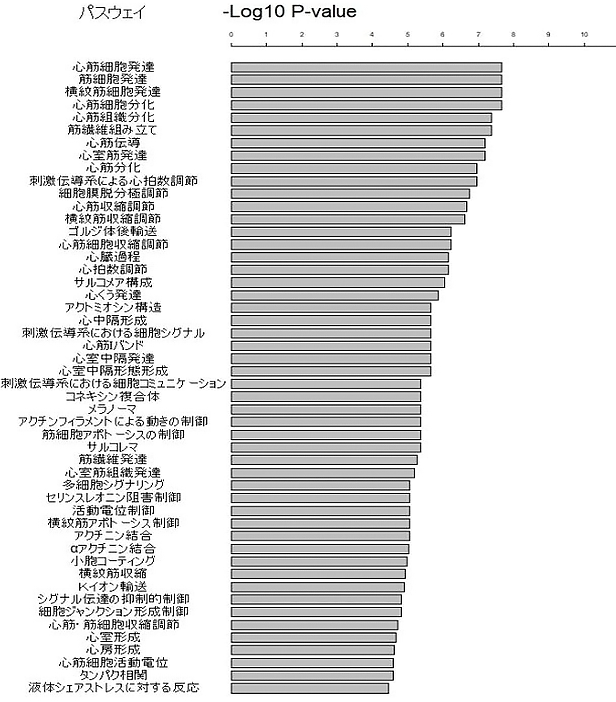
図1 有意となった生物学的パスウェイ
今回検出された感受性座位を用いた解析で、55の生物学的パスウェイが心臓細動発症に関与していることが明らかになった。これらは心臓の形成分化、電気生理学的機能、心筋細胞収縮、心臓形態形成に関係するものであった。
次に、心房細動の遺伝的人種差を検討するために、人種特異的メタ解析を行いました。この結果、欧米人のメタ解析でCDK6 遺伝子領域、EPHA3遺伝子領域、GOSR2遺伝子領域の3個の感受性座位を新たに発見しました。これらは多人種のメタ解析では有意にはならず、欧米人特有のものと考えられました。また、日本人と欧米人では、染色体4q25のPITX2遺伝子の上流領域が最も強く心房細動と相関し、これはアフリカ系アメリカ人でも同様の結果を得ました。その他の多人種メタ解析で得られた重要な感受性座位のほとんどは、人種間で有意な不均質性が認められず、これらは人種の違いによらず心房細動の発症に関与していること示しています。
多人種メタ解析で発見したマーカー67個のうち、64個は遺伝子調節領域[11]上に存在し、さらに54個は心臓組織のエンハンサー領域[12]に存在していました。そのため、今回発見された感受性座位が直接作用する遺伝子を決定するために、左心房組織のトランスクリプトーム[3]情報や組織特異的遺伝子発現データベースを用いたeQTL解析[13]を行いました。その結果、心房細動の感受性座位での多型が有意に発現を変化させている遺伝子を57個同定しました。
最後に、UKバイオバンク[14]の臨床データを用いた解析により、今回発見した心房細動マーカーは、身長、肥満の指標であるBMI[15]、高血圧症と遺伝的相関を示しました。また、PITX2遺伝子領域は、心不全、脳梗塞、徐脈への、CEP68遺伝子領域は肺血管病、高脂血症、高血圧、冠動脈疾患、2型糖尿病への多相遺伝(Pleiotrophy)[16]が示しました。
今後の期待
本研究により、多人種間で共通、または人種特異的な心房細動の感受性座位を明らかにしました。今後これらを手掛かりに心房細動発症の詳細なメカニズムの解明や、メカニズムに関連した分子ターゲットの発見によって疾患に効果的な創薬につながるものと期待できます。
なお、本研究で用いた日本人集団におけるジェノタイプデータ[17]は、科学技術振興機構(JST)バイオサイエンスデータベースセンター(NBDC)を通じて公開しています。
論文情報
- タイトル
- Multi-Ethnic Genome-wide Association Study for Atrial Fibrillation
- 著者名
- Carolina Roselli, Mark D. Chaffin, Lu-Chen Weng, Stefanie Aeschbacher, Gustav Ahlberg, Christine M. Albert, Peter Almgren, Alvaro Alonso, Christopher D. Anderson, M Krishna G. Aragam, Dan E. Arking, John Barnard, Traci M. Bartz, Emelia J. Benjamin, Nathan A. Bihlmeyer, Joshua C. Bis, Heather L. Bloom, Eric Boerwinkle, Erwin B. Bottinger, Jennifer A. Brody, Hugh Calkins, Archie Campbell, Thomas P. Cappola, John Carlquist, Daniel I. Chasman, Lin Y. Chen, Yii-Der Ida Chen, Eue-Keun Choi, Seung Hoan Choi, Ingrid E. Christophersen, Mina K. Chung, John W. Cole, David Conen, James Cook, Harry J. Crijns, Michael J. Cutler, Scott amrauer, Brian R. Daniels, Dawood Darbar, Graciela Delgado, Joshua C. Denny, Martin Dichgans, Marcus Dörr, Elton A. Dudink, Samuel C. Dudley, Nada Esa, Tonu Esko, Markku Eskola, Diane Fatkin, Stephan B. Felix, Ian Ford, Oscar H. Franco, Bastiaan Geelhoed, Raji Grewal, Vilmundur Gudnason, Xiuqing Guo, Namrata Gupta, Stefan Gustafsson, Rebecca Gutmann, Anders Hamsten, Tamara B. Harris, Caroline Hayward, Susan R. Heckbert, Jussi Hernesniemi, Lynne J. Hocking, Albert Hofman, Andrea R. V. R. Horimoto, Jie Huang, Paul L. Huang, Jennifer Huffman, Erik Ingelsson, Esra Gucuk Ipek, Kaoru Ito, Jordi Jimenez-Conde Renee Johnson, J. Wouter Jukema, Stefan Kääb, Mika Kähönen, Yoichiro Kamatani, John P. Kane, Adnan Kastrati, Sekar Kathiresan, Petra Katschnig-Winter, Maryam Kavousi, Thorsten Kessler, Bas L. Kietselaer, Paulus Kirchhof, Marcus E. Kleber, Stacey Knight, MStat Jose E. Krieger, Michiaki Kubo, Lenore J. Launer, Jari Laurikka, Terho Lehtimäki, Kirsten Leineweber, Rozenn N. Lemaitre, Man Li, Hong Euy Lim, Henry J. Lin, Honghuang Lin, Lars Lind, Cecilia M. Lindgren, Marja-Liisa Lokki, Barry London, Ruth J. F. Loos, Siew-Kee Low, Yingchang Lu, Leo-Pekka Lyytikäinen, Peter W. Macfarlane, Patrik K. Magnusson, Anubha Mahajan, Rainer Malik, Alfredo J. Mansur, Gregory M. Marcus, Lauren Margolin, Kenneth B. Margulies, Winfried März, David D. McManus, Olle Melander, Sanghamitra Mohanty, Jay A. Montgomery, Michael P. Morley, Andrew P. Morris, Martina Müller-Nurasyid, Andrea Natale, Saman Nazarian, Benjamin Neumann, Christopher Newton-Cheh, Maartje N. Niemeijer, Kjell Nikus, Peter Nilsson, Raymond Noordam, Heidi Oellers, Morten S. Olesen, Marju Orho-Melander, Sandosh Padmanabhan, Hui-Nam Pak, Guillaume Paré, Nancy L. Pedersen, Joanna Pera, Alexandre Pereira, David Porteous, Bruce M. Psaty, Sara L. Pulit, Clive R. Pullinger, Daniel J. Rader, Lena Refsgaard, Marta Ribasés, Paul M. Ridker, Michiel Rienstra, Lorenz Risch, Man Roden, Jonathan Rosand, Michael A. Rosenberg, Natalia Rost, FAAN Jerome I. Rotter, Samir Saba, Roopinder K. Sandhu, Renate B. Schnabel, Katharina Schramm, Heribert Schunkert, Claudia Schurman, Stuart A. Scott, Ilkka Seppälä, Christian Shaffer, Svati Shah, Alaa A. Shalaby, Jaemin Shim, M. Benjamin Shoemaker, Joylene E. Siland, Juha Sinisalo, Moritz F. Sinner, Agnieszka Slowik, Albert V. Smith, Blair H. Smith, J. Gustav Smith, Jonathan D. Smith, Nicholas L. Smith, Elsayed Z. Soliman, Nona Sotoodehnia, Bruno H. Stricker, Albert Sun, Han Sun, Jesper H. Svendsen, Toshihiro Tanaka, Kahraman Tanriverdi, Kent D. Taylor, Maris Teder-Laving, Alexander Teumer, Sébastien Thériault, Stella Trompet, Nathan R. Tucker, Arnljot Tveit, Andre G. Uitterlinden, Pim Van Der Harst, Isabelle C. Van Gelder, David R. Van Wagoner, Niek Verweij, Efthymia Vlachopoulou, Uwe Völker, Biqi Wang, Peter E. Weeke, Bob Weijs, Raul Weiss, Stefan Weiss, Quinn Wells, Kerri L. Wiggins, Jorge Wong, Daniel Woo, Bradford B. Worrall, Pil-Sung Yang, Jie Yao, Zachary T. Yoneda, Tanja Zeller, Lingyao Zeng, Steven A. Lubitz , Kathryn L. Lunetta, Patrick T. Ellinor.
- 雑誌
- Nature genetics
- DOI
- 10.1038/s41588-018-0133-9
補足説明
- [1] ゲノムワイド関連解析(GWAS)
- GWASは、Genome-Wide Association Studyの略。疾患の感受性遺伝子を見つける代表的な方法。ヒトゲノムを網羅した数百万~1,000万の一塩基多型を対象に、対象サンプル群における疾患との因果関係を評価できる。2002年に世界で初めて理化学研究所で実施された手法であり、以後世界中で利用されている。
- [2] 疾患感受性遺伝子
- 単一遺伝子病の原因遺伝子のように、遺伝子に変異があると必ず発症するというものではなく、変異があると発症しやすくなったり、逆に発症しにくくなったりする遺伝子。
- [3] トランスクリプトーム解析、トランスクリプトーム
- トランスクリプトームとは、一つのゲノム、または特定の細胞・組織・器官の中で生産される転写産物(転写によって合成されたRNA)全体を指す。トランスクリプトーム解析は、特定の遺伝子発現に着目するのではなく、全ての発現遺伝子に関して網羅的に解析する手法。マイクロアレイや次世代シークエンサーを用いたRNAシークエンスなどの方法がある。
- [4] オミクス解析
- ゲノム情報やエピゲノム情報など、生体内や細胞内の情報を網羅的に解析し、生命現象を調べる解析手段。
- [5] アレル頻度
- 個々のヒトゲノムを比較するとその塩基配列に違いがある。例えば、ある染色体上の位置において、個人によりAA/AG/GGのどれかの塩基配列を持つ。集団の中でのA(アデニン)やG(グアニン)の頻度をアレル頻度という。
- [6] 遺伝子多型、一塩基多型(SNP)
- ヒトゲノムの個人間の違いのうち、集団での頻度が1%以上のものを遺伝子多型と呼ぶ。代表的なものとして、ヒトゲノム塩基配列上の1カ所が変化して生じる一塩基多型がある。SNPはSingle Nucleotide Polymorphismの略。
- [7] メタ解析
- 二つ以上の統計解析結果を合わせる際に、それぞれの解析結果でばらつきのある面を統計学的に排除し、偏りのない合算をする手法。
- [8] 相対危険度
- リスクアレル(疾患発症に寄与する方のアレル)を持っている場合にそれを持っていない場合に比べて何倍疾患が発症しやすいかを表す尺度。
- [9] GSEA解析
- 過去の実験データより得られた特定の生物学的機能に関係する遺伝子群を遺伝子セット(Gene Set)としてあらかじめ準備しておき、変化した遺伝子群がどの遺伝子セットに多く含まれているかを調べることにより、その遺伝子変化の生物学的意味を解析する手法。GSEAはGene-set Enrichment Analysisの略。
- [10] パスウェイ
- シグナル伝達系、遺伝子の制御関係、代謝経路、タンパク質間相互作用などの情報をもとにグループ化された遺伝子やタンパク質の集合とその経路情報。
- [11] 遺伝子調節領域
- 遺伝子の転写を調節するエンハンサー、プロモーター、サイレンサーなどのゲノム領域のこと。
- [12] エンハンサー領域
- 遺伝子の発現を調節するようなDNA領域。この領域に転写因子などのタンパク質が結合することで、遺伝子の発現が調節される。遺伝子の転写開始点から近い転写調節DNA領域がプロモーター領域で、転写開始点からは隔たっている転写調節DNA領域がエンハンサー領域。
- [13] eQTL解析
- QTLとはQuantitative trait locus(量的形質遺伝子座)の略であり、量的形質がどのように生物に表現されるかに影響を与える染色体上のDNA領域のことである。eQTL解析は遺伝子発現プロファイリングとQTL解析を組み合わせた手法であり、疾病など形質に関連する遺伝子そのもののほか、その発現に影響する調節因子(遺伝子近傍にある調節配列などのシス因子、および遺伝子に結合する転写因子の遺伝子などのトランス因子)を明らかにすることが可能である。
- [14] UKバイオバンク
- 英国で構築されているバイオバンクで、50万人規模の疾患罹患情報、臨床情報、遺伝情報などからなる。
- [15] BMI
- ヒトの肥満の程度を示す指標。「BMI = 体重(kg)/身長(m)2」の計算式に基づき体重と身長の関係から算出される。BMIはBody Mass Indexの略。
- [16] 多相遺伝(Pleiotrophy)
- ある一つの遺伝素因が特定の疾患だけでなく、複数の疾患に作用する多面的作用のこと。
- [17] ジェノタイプデータ
- 実際のサンプルから得られたヒトゲノム配列データの総称。個人間で異なるヒトゲノム配列部位を対象にデータ化されている。
お問い合わせ先
発表者
- 理化学研究所 統合生命医科学研究センター
- 副センター長(研究当時)久保 充明(くぼ みちあき)
- 統計解析研究チーム
- チームリーダー(研究当時)鎌谷 洋一郎(かまたに よういちろう)
(現 理化学研究所 生命医科学研究センター 統計解析研究チーム チームリーダー、京都大学大学院医学研究科 ゲノム医学センター 准教授) - 客員研究員(研究当時)ロー・シュー・キー(Low Siew Kee)
(現 理化学研究所 生命医科学研究センター 統計解析研究チーム 客員研究員、がん研究会 がん研究所 がんプレシジョン医療研究センター グループリーダー) - 循環器疾患研究チーム
- チームリーダー(研究当時)伊藤 薫(いとう かおる)
(現 理化学研究所 生命医科学研究センター 循環器疾患研究チーム チームリーダー) - 東京医科歯科大学大学院 医歯学総合研究科 疾患多様性遺伝学分野
- 教授 田中 敏博(たなか としひろ)
機関窓口
理化学研究所 広報室 報道担当
東京医科歯科大学 総務部総務秘書課広報係
AMED事業に関するお問い合わせ先
日本医療研究開発機構(AMED)
基盤研究事業部 バイオバンク課