スプライシング調節化合物による抗がんメカニズムを解明
2021-09-14 理化学研究所,東京大学
理化学研究所(理研)開拓研究本部岩崎RNAシステム生化学研究室の岩崎信太郎主任研究員、水戸麻理テクニカルスタッフⅠ、環境資源科学研究センターケミカルゲノミクス研究グループの吉田稔グループディレクター(東京大学大学院農学生命科学研究科応用生命工学専攻教授)、チピ・シュレスタ・ジャガット特別研究員、ティルマン・シュナイダー・ポッチュ研究員、カーン・カリッド研修生(研究当時)、同生命分子解析ユニットの堂前直ユニットリーダー、鈴木健裕専任技師らの研究グループは、スプライシング[1]調節化合物の「スプライソスタチンA(SSA)」がイントロン[2]からの翻訳を誘導し、異常タンパク質の凝集体の形成を介して、タンパク質合成の全体を抑えるという、一連の仕組みを解明しました。
本研究成果は、スプライシング調節化合物の抗がんメカニズムの一端を明らかにしたものであり、今後の抗がん剤開発に貢献するものと期待できます。
スプライシング調節化合物の中には、がんの治療薬としての可能性を持つものがあります。しかし、スプライシング調節化合物が持つスプライシング調節活性と抗がん活性との関係はまだ完全には解明されていません。
今回、研究グループは、スプライシング調節化合物であるスプライソスタチンAを処理した細胞から、イントロン配列に由来する短縮型異常タンパク質を網羅的に同定しました。そして、これら短縮型異常タンパク質が凝集体を形成しやすい性質を持つこと、mTORC1[3]と呼ばれるシグナル伝達をつかさどる複合体を不活性化することなどを突き止めました。
本研究は、科学雑誌『Cell Chemical Biology』オンライン版(9月13日付:日本時間9月14日)に掲載されます。
背景
「スプライシング」は、DNAから転写されたメッセンジャーRNA(mRNA)前駆体に含まれるタンパク質合成に不必要な部分(イントロン)を除き、必要な部分(エクソン)を連結する反応です。スプライシングは酵母からヒトまで高度に保存されており、遺伝子が正しく機能する上で最も重要な反応の一つと見なされています。一方、イントロン配列の変異やスプライシング因子の変異が原因によるスプライシングの異常は、がんや精神・神経疾患の原因となることから、スプライシングを標的とする遺伝子治療が注目されています。
理研を中心とする共同研究チームは2007年に、スプライシング調節作用を持つ抗がん活性物質の一つである「スプライソスタチンA(Spliceostatin A:SSA)」を世界に先駆けて発見しました注1)。スプライソスタチンAは、スプライシング因子U2と呼ばれる低分子核内リボヌクレオタンパク質[4]のサブユニットSF3bに特異的に結合し、スプライシング反応を阻害します。この発見を契機に複数のSF3bを標的とするスプライシング調節化合物が開発されてきました。
これらのスプライシング調節化合物の中には、骨髄異形成症候群や急性骨髄性白血病の治療薬としての可能性を持つものがあり、臨床試験が行われています。しかし、スプライシング調節化合物が持つスプライシング調節活性と抗がん活性との関係は、まだ完全には解明されていません。
注1)Kaida D et al., Spliceostatin A targets SF3b and inhibits both splicing and nuclear retention of pre-mRNA.Nat Chem Biol 3, 576-583 (2007)
研究手法と成果
スプライソスタチンAによってスプライシングが阻害されると、mRNA前駆体にイントロンが保持されることが分かっています。通常イントロンは除去されるためタンパク質に翻訳されることはありませんが、もし翻訳されれば、頻繁に翻訳終止コドン[5]が出現して、途中までしか翻訳されない短縮型異常タンパク質が出現するはずです。
研究グループは、RNA-Seq法[6]、リボソームプロファイリング法[7]を用いてイントロンが保持されたmRNAが翻訳を受け、エクソン-イントロンキメラに由来する短縮型異常タンパク質が多数合成されていることを見出しました。さらにタンパク質質量分析[8]を組み合わせることで、スプライソスタチンAを処理した細胞からイントロン配列由来の翻訳産物を含む短縮型異常タンパク質を網羅的に同定しました(図1)。
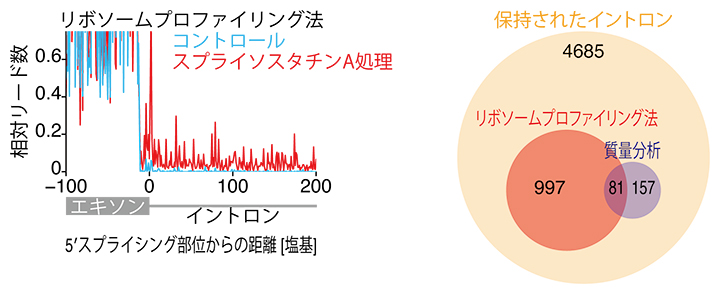
図1 スプライソスタチンAによって誘導される短縮型異常タンパク質の網羅的同定
(左)リボソームプロファイリング法によって、イントロンから翻訳が生じていることを同定した。
(右)RNA-Seq法、リボソームプロファイリング法、タンパク質の質量分析から同定したイントロン由来の短縮型異常タンパク質のまとめ。RNA-Seq法により4,685個をRNAレベルで同定し、そのうちリボソームプロファイリング法により1,078個を翻訳レベルで、質量分析により238個をタンパク質レベルで同定した。
驚くべきことに、これらの短縮型異常タンパク質のイントロン由来配列は、セリン、プロリン、グリシン、およびアルギニンなどの特定のアミノ酸に富んでおり、low complexity region(LCR)と呼ばれる配列を持つことが分かりました。LCRは、液液相分離[9]などのタンパク質凝集体を形成する配列であることが知られています。実際に、多くのイントロン由来配列を含む短縮型異常タンパク質が沈殿し、凝集体を形成することが明らかになりました。
多くの凝集体が形成されると、細胞内のタンパク質恒常性が乱され、細胞ストレスを誘導することが知られています。実際に、細胞にスプライソスタチンA処理を施すと、細胞ストレス応答が誘導され、mTORC1と呼ばれるシグナル伝達をつかさどる複合体が不活性化されることを発見しました。
mTORC1の不活性化は下流のリン酸化経路を阻害し、特に翻訳阻害タンパク質4EBPの脱リン酸化を引き起こし、脱リン酸化型4EBPにより、タンパク質の合成が阻害されることが知られています。今回、スプライソスタチンAによっても同様の4EBPの脱リン酸化および翻訳阻害が観察されました。
実際にmTORC1を標的とした化合物が抗がん作用を持つことが知られており、本研究で明らかになったスプライシング阻害によるイントロン保持、イントロン翻訳、ストレス応答、mTORC1阻害、および翻訳阻害という一連の経路が、実際のスプライシング調節化合物の抗がん作用の原因であると推察されます(図2)。
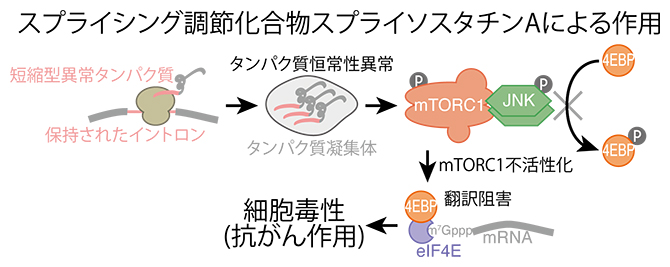
図2 スプライソスタチンAによって誘導される一連の反応
スプライシング調節化合物によって多数のmRNAのスプライシングが阻害され、mRNAにイントロンが保持される。この場合、元来mRNAにコードされたタンパク質より短く、かつイントロン配列由来のキメラタンパク質が合成される。この短縮型異常タンパク質はタンパク質凝集体を形成しやすい。タンパク質凝集体が蓄積すると細胞ストレスとなり、JNKと呼ばれるキナーゼ(リン酸化酵素)がリン酸化され活性化し、さらにmTORC1と呼ばれるキナーゼ複合体がリン酸化され不活性化する。通常、mTORC1は翻訳阻害タンパク質である4EBPをリン酸化させ不活性化させているが、mTORC1の不活性化により4EBPが脱リン酸化され、翻訳抑制能を発揮する。このような経路により、最終的に細胞内のタンパク質合成が減少し、細胞毒性(抗がん作用)を発揮すると考えられる。
今後の期待
スプライシング機構を標的とした多数の化合物が抗がん剤として注目される中、その抗がん作用機構の詳細の解明が望まれていました。今回、スプライシング調節化合物の作用機序の一端が明らかになったことから、本研究の知見を基にした抗がん剤のさらなる開発が期待できます。
また、遺伝子配列中にイントロンが存在することの意義はよく分かっていませんが、イントロンが翻訳されるような状況が発生すると、それ以上の翻訳を抑制して毒性を回避するフィードバック機構がイントロン配列中に隠されている可能性も示されました。今後、イントロンの役割についても理解が深まるものと期待できます。
補足説明
1.スプライシング
遺伝子発現のためには、最初に遺伝子がタンパク質翻訳の鋳型であるmRNAへと転写される。転写直後のmRNAは前駆体の状態にある。真核生物の場合、多くの遺伝子はエクソンとイントロンと呼ばれる2種類の領域を含んでいるが、このうちイントロンはタンパク質配列を指定しない領域で、最終的にはmRNAから取り除かれる必要がある。このイントロン除去過程をスプライシングと呼ぶ。スプライシングは核内で起こり、イントロンが除去された成熟mRNAは核外へ運ばれ、タンパク質に翻訳される。
2.イントロン
mRNA前駆体に存在するタンパク質に翻訳されない領域。スプライシングの結果、イントロン配列は、それぞれ投げ縄構造を形成し、mRNA前駆体から除去される。
3.mTORC1
細胞内外の環境情報に応答するキナーゼ(リン酸化酵素)複合体。細胞が適切な環境にない場合、不活性化される。mTORC1はmechanistic target of rapamycin complex 1の略。
4.低分子核内リボヌクレオタンパク質
低分子核内RNA(snRNA)とタンパク質が形成する複合体。複数種の低分子核内リボヌクレオタンパク質が複雑に連携し、スプライシング反応を担う。
5.終止コドン
遺伝暗号を構成し、タンパク質のアミノ酸配列を定義するコドン配列のうち、タンパク質合成の終わりを示すコドン。
6.RNA-Seq法
いわゆる「次世代シークエンサー」を用いて、RNAの配列断片データを網羅的に取得する方法。これを基に、遺伝子などの転写産物の発現量や配列、スプライスバリアントなどが解析できる。
7.リボソームプロファイリング法
組織からリボソームを抽出し、リボソームと結合しているRNA配列を同定することにより、どの遺伝子がどの程度の効率で翻訳されているかを知る解析法。リボソームは大きな複合体であるため、一定のmRNA領域を覆うように結合する。これらのリボソームとmRNAの複合体に対してRNA分解酵素で処理すれば、リボソームが保護するmRNA断片だけが分解されず回収できる。
8.質量分析
物質をイオン化し、そのイオンを検出することにより、原子や分子の質量を推定する分析法。
9.液液相分離
溶液が二つの液相に分離する現象(例えば、水の中の油が分離するなど)を指す。細胞の中のタンパク質や核酸が液液相分離を引き起こし、細胞内で特殊な環境を形成することが近年の研究で明らかになっている。
研究支援
本研究は、日本学術振興会(JSPS)科学研究費補助金基盤研究(S)「革新的化学遺伝学による内在性代謝物の新機能の解明と応用(研究代表者:吉田稔)」、同新学術領域研究(研究領域提案型)「ケモテクノロジーが拓くユビキチンニューフロンティア(領域代表者:佐伯泰)」、同若手研究A「翻訳開始因子パラログによる選択的翻訳の網羅的解析(研究代表者:岩崎信太郎)」、同新学術領域研究「Chasing translation along the time by novel ribosome profiling(研究代表者:岩崎信太郎)」、同挑戦的研究(萌芽)「重力による翻訳制御の網羅的解析研究(代表者:岩崎信太郎)」、同学術変革領域研究(B)「新規Disome-Seq法: パラメトリックなリボソーム渋滞の網羅的探索(代表者:岩崎信太郎)」、同若手研究「Study on molecular mechanism of anti-tumor activity by splicing modulation (代表者:チピ・シュレスタ・ジャガット)」などの支援を受けて行われました。
原論文情報
Jagat K. Chhipi-Shrestha, Tilman Schneider-Poetsch, Takehiro Suzuki, Mari Mito, Khalid Khan, Naoshi Dohmae, Shintaro Iwasaki, and Minoru Yoshida, “Splicing modulators elicit global translational repression by condensate-prone proteins translated from introns”, Cell Chemical Biology, 10.1016/j.chembiol.2021.07.015
発表者
理化学研究所
開拓研究本部 岩崎RNAシステム生化学研究室
主任研究員 岩崎 信太郎(いわさき しんたろう)
テクニカルスタッフⅠ 水戸 麻理(みと まり)
環境資源科学研究センター
ケミカルゲノミクス研究グループ
グループディレクター 吉田 稔(よしだ みのる)
(東京大学大学院農学生命科学研究科応用生命工学専攻 教授)
特別研究員 チピ・シュレスタ・ジャガット
(Jagat K. Chhipi-Shrestha)
研究員 ティルマン・シュナイダー・ポッチュ
(Tilman Schneider-Poetsch)
研修生(研究当時) カーン・カリッド(Khalid Khan)
生命分子解析ユニット
ユニットリーダー 堂前 直(どうまえ なおし)
専任技師 鈴木 健裕(すずき たけひろ)
報道担当
理化学研究所 広報室 報道担当
東京大学大学院農学生命科学研究科
事務部 総務課 総務チーム 総務・広報情報担当