2022-02-22 理化学研究所
理化学研究所(理研)生命機能科学研究センター細胞システム動態予測研究チームの金坚石研究員、山本れいこテクニカルスタッフ、城口克之チームリーダーらの共同研究グループは、多種・多数の細菌で構成される細菌叢[1]中の個々の細菌を一つ一つ区別して極めて正確に細菌の種類と数を計測する手法を開発し、ビタミンA欠乏によるマウス腸内細菌叢の微少な変化を捉えることに成功しました。
本研究成果は、腸内細菌に関連する病態の理解や診断に貢献するとともに、皮膚、口腔、植物、土壌、海洋、大気などのさまざまな器官や環境に存在する細菌叢の高精度計測へ発展させることで、幅広い科学分野への応用が期待できます。
ヒトの腸には約100兆個、500~1,000種類の細菌が存在し、腸内細菌叢を構成しています。腸内細菌叢のバランスは腸管免疫[2]に関与するなど健康との関係が注目されていますが、その影響を理解するには、どの種類の細菌が何個存在するかを正確に計測することが重要です。
今回、共同研究グループは、細菌一つ一つを液滴に封じ込め、細菌ごとに異なる目印(細胞バーコード[3])を付けることで、一度に10万個以上の細菌を区別し、細菌の種類と各種類の細菌数を正確に計測する「BarBIQ(Barcoding Bacteria for Identification and Quantification)法」を開発しました。このBarBIQ法を用いて、ビタミンAを除去した食餌により生じたマウス盲腸内の腸内細菌数の微少な変化を定量することに成功しました。
本研究は、オンライン科学雑誌『Nature Communications』(2月22日付:日本時間2月22日)に掲載されました。
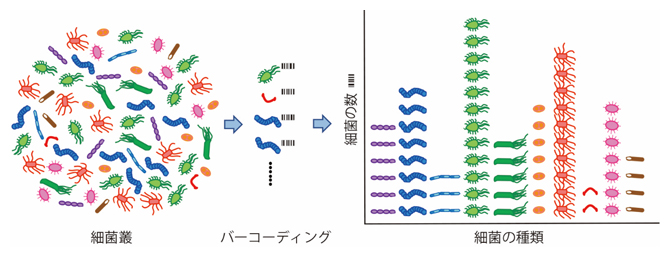
細菌一つを見分けて細菌叢全体を解析するBarBIQ法
背景
細菌は地球のさまざまな場所に生息し、他種の生物と共生関係になる細菌も多く見られます。例えば、ヒトの腸には100兆個程度の腸内細菌が存在し、その種類は500~1,000程度とされています。このような多数・多種の細菌が構成する生態系を「細菌叢」と呼び、腸内細菌叢のバランスが乱れるとヒトの健康に悪影響を与えることが報告されています。特に、腸管免疫には腸内細菌叢が密接に関与すると考えられますが、腸内細菌叢とヒトの健康の関係についてはまだ不明な点が多く、その影響を正確に理解するためには、どの種類の細菌が何個存在するかを正確に計測することが重要です。
細菌の種類を決めるとき、特に多数の細菌を一度に計数する場合は細菌のDNAを調べます。よく使われるのは、16SリボソームRNA(16S rRNA)[4]をコードするDNA配列をシークエンサーで解析する手法です。しかし、従来法では、16S rRNAの配列を決める過程で、多くの細菌種が含まれる試料からDNAを抽出して16S rRNA配列を持つDNA分子を増幅することが多く、このときに異なる16S rRNA配列が部分的につながってしまい、細菌の種類を正確に決定できないという課題がありした。また、細菌の計数値は増幅された16S rRNA配列の数により推定されてきましたが、細菌の種類により16S rRNA配列の数が1~15まで幅のあることが分かっており、細菌数の実測も極めて重要な課題となっていました。
細菌叢の計測におけるこれらの技術的な課題は、腸内細菌叢の微少な変化を捉えることを困難にしています。例えば、食餌に含まれるビタミンやミネラルの欠乏により腸内細菌叢の組成が変化することが分かっています。なかでもビタミンAは視覚や成長などとともに腸管免疫にも関与しており、発展途上国におけるビタミンA欠乏症は小児失明の主因であり、かつ下痢や麻疹などの小児感染症の罹患率や死亡率に影響を与えることが知られています。しかし、ビタミンAの欠乏で腸内細菌叢内の細菌の種類と数がどのように変化するかは正確に測定されていませんでした。
研究手法と成果
共同研究グループは、細菌叢の解析においてこれまで不可能であった、一つ一つの細菌の種類を正確に決定し、細菌数を高精度に計数する革新的な手法を開発しました(図1)。本手法は、城口克之チームリーダーらが2012年と2017年に報告したDNA分子バーコード法[5]を利用することから注1, 2)、「BarBIQ(Barcoding Bacteria for Identification and Quantification)法」と名付けました。BarBIQ法では、細菌叢に含まれる細菌を一つ一つ液滴に封じ込め、それらを区別するためのDNA配列(細胞バーコード)を細菌DNAに付与しながらDNAを増幅します(図1)。一連の手順は、以下のようになります。
①採取した細菌叢に細胞バーコードを加えた反応液を調製し、一つの細菌と1分子の細胞バーコードを含む液滴が多くなる条件の下で、液滴を作製します(図2)。
②液滴の状態のまま、細菌に由来する16S rRNAのDNA分子と、細胞バーコードのDNA分子を同時に増幅します。これら二つのDNA分子は、増幅中に双方の配列が1本につながるように設計されています。
③液滴を破砕して増幅したDNA分子をまとめて回収し、回収したDNAの配列(16S rRNAと細胞バーコード)を次世代シークエンサー[6]で一挙に決定します。
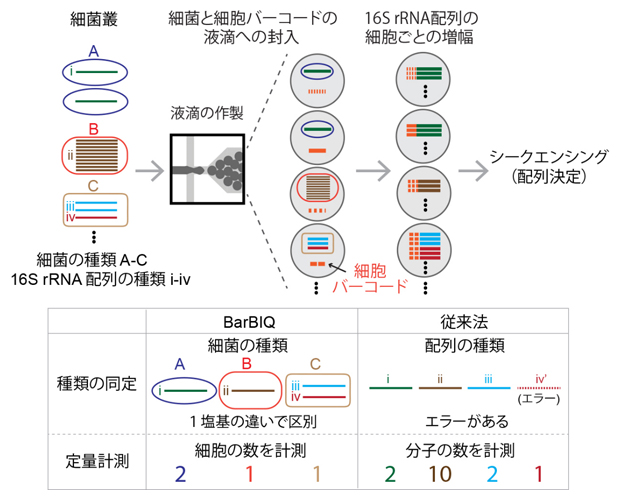
図1 BarBIQ法の手順および従来法との比較
細菌叢にA、B、Cの3種類の細菌がそれぞれ2細胞、1細胞、1細胞存在した場合の解析例を示す。細菌Aは16S rRNA配列を1種類1個、細菌Bは1種類10個、細菌Cは一つの配列2個と別の配列1個を持つものとする。BarBIQ法は各細菌の数(細胞数)を計数し、細胞バーコードにより遺伝子配列のわずかな違いも正確に区別して細菌の種類を同定する。従来法では16S rRNA配列のコピー数(分子数)を計数しており、配列決定エラーも相対的に多い。

図2 細菌を封じ込めた液滴
左)作製直後の液滴の明視野顕微鏡像。
右)液滴中の細菌DNAを蛍光染色で光らせたときの蛍光像。矢印の先に細菌が観察されている。
BarBIQ法では、各細菌を液滴で1細胞ずつ分画することにより、DNAの増幅を行なっても複数の細菌の16S rRNA配列がつながらないようにしました。また、細菌ごとに異なる細胞バーコードが付与されていることを利用して1塩基しか違わない16S rRNA配列を区別し、同じ種類の細菌が持つ似た配列や、違う種類の細菌に由来する似た配列を見分けることができました。さらに、細胞バーコードの種類を計数することで、16S rRNAの数ではなく細胞の数の計数を実現しました。例えば、同一の16S rRNA配列に異なるバーコードが付いたDNAが2パターン見つかれば、同じ種類の細菌が入っていた液滴が2個あったことになります。これらにより、一度に10万個以上の細菌を正確に計測することが可能になりました。
次に実証実験として、ビタミンAの欠乏が腸内細菌叢に与える影響を詳しく調べました。ビタミンAを含む飼料、含まない飼料をそれぞれ与えたマウスの盲腸2カ所(小腸-盲腸-大腸結合部に近い部位と遠い部位)から腸内細菌叢を採取し、BarBIQ法で計測しました(図3左)。その結果、検出された細菌の種類はビタミンAの有無や盲腸内の違う部位でもあまり変わらず、それぞれで200種類を超える細菌の存在が確認されました。
一方、個々の細菌において飼料中のビタミンA無しの場合を有りの場合と比較したところ、結合部に近い部位では8種類、遠い部位では1種類の細菌の数が変化しており、最も減少した細菌で約1/40倍、最も増加したもので約600倍変化したことが分かりました(図3右)。さらに、16S rRNA配列を正確に決定することにより、1,000以上の未登録の16S rRNAの配列を発見しました。このようにして、BarBIQ法により、少数の種類の細菌叢の微少な変化を捉えることができました。
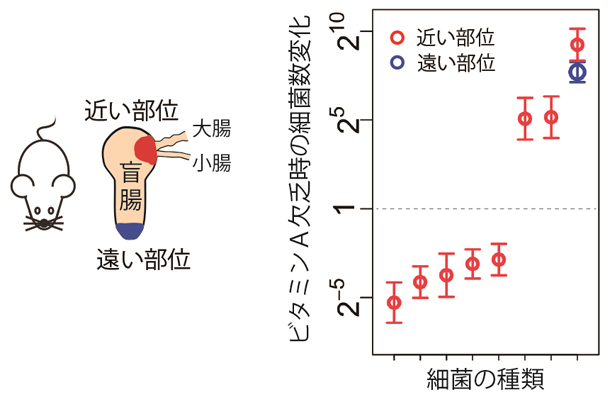
図3 ビタミンA欠乏が腸内細菌叢に与える影響
左)マウスの食餌をビタミンA有り/無しの2群に分け、それぞれの盲腸の小腸-盲腸-大腸結合部に近い部位(赤)と遠い部位(青)から細菌叢を採取し、BarBIQ法で解析した。
右)ビタミンA有り群に対して、ビタミンA無し群で細菌数が統計的に有意に変化した8種類の細菌を示した。小腸-盲腸-大腸結合部に近い部位(赤)において、5種類が減少(最大で約40分の1[約2-5.3])、3種類が増加(最大で約600倍[約29.2])し、遠い部位(青)において1種類が増加した。
注1)Shiroguchi K., Jia T.Z., Sims P.A., Xie X.S.”Digital RNA sequencing minimizes sequence-dependent bias and amplification noise with optimized single molecule barcodes”Proc. Natl. Acad. Sci. USA, 109, 1347-1352 (2012).
注2)2017年10月19日プレスリリース「DNA分子バーコード法の新機能」
今後の期待
細菌叢研究の技術的な課題を解決し、世界で初めて一度に10万個以上の細菌の正確な計測を実現したBarBIQ法は、”木”(個々の細菌)と”森”(細菌叢)を同時に”見る”画期的な手法です。本手法を皮膚、口腔などの器官や、植物、土壌、海洋、大気などさまざまな環境に存在する細菌叢の高精度計測へ発展させることで、幅広い科学分野に貢献することが期待できます。
また、腸内細菌叢におけるビタミンAの影響を解析した例のように、ビタミンやミネラル欠乏症の詳細な機序の理解を含め、腸内細菌叢を介した新たな診断、治療、予防などの一助になることも期待できます。
補足説明
1.細菌叢
多種多様な細菌が集まって存在するときの細菌集団全体の呼称。例えば、腸内細菌叢は宿主の免疫系の構築や栄養素の供給、消化の補助などの役割を担っている。近年の研究から、腸内細菌叢のバランスが崩れるとさまざまな疾患につながることが報告されており、病気の予防や治療の重要なターゲットとして注目されている。
2.腸管免疫
腸管の内腔は、食物とともに摂取される微生物などに常にさらされている。これらに対して、必要に応じて免疫応答を誘導し、病原体を排除したり腸内細菌バランスを維持したりする生体防御システムを腸管免疫という。
3.細胞バーコード
細胞を区別するために付与される、それぞれ異なる配列を持つ核酸分子や核酸分子群。同じ種類の細胞でも、それぞれの細胞に異なる細胞バーコードを付与することにより、各細胞を区別できる。
4.16SリボソームRNA(16S rRNA)
タンパク質合成装置であるリボソームを構成するRNAのうちの一つ。リボソームは全ての生物が持つとされ、構成要素をコードするDNA配列が生物の種間比較や系統分類によく用いられる。16SリボソームRNAは、原核生物のリボソーム小サブユニットの構成要素であるRNAの名称。
5.DNA分子バーコード法
それぞれ異なる配列を持つDNA分子(DNA分子バーコード)を、目的の核酸分子(DNAやRNA)の一つ一つに付加することで、目的の核酸分子の一つ一つを区別し、その数をデジタル的に正確に数えることができる方法。
6.次世代シークエンサー
サンガー法を利用したシークエンサーである「第一世代シークエンサー」と対比させて使われる用語。機種によるが、一度に1,000万以上のDNA分子の配列を決定できる。
共同研究グループ
理化学研究所
生命機能科学研究センター 細胞システム動態予測研究チーム
チームリーダー 城口 克之(しろぐち かつゆき)
研究員 金 坚石(じん じゃんし、Jin Jianshi)
テクニカルスタッフ 山本 れいこ(やまもと れいこ)
基礎科学特別研究員 北條 望(ほうじょう のぞみ)
生命医科学研究センター 粘膜システム研究チーム
チームリーダー 大野 博司(おおの ひろし)
研修生/研究パートタイマーⅠ 竹内 直志(たけうち ただし)
上級研究員 宮内 栄治(みやうち えいじ)
京都大学 ウイルス・再生医科学研究所 免疫制御分野
教授 生田 宏一(いくた こういち)
助教 崔 广為(さい こうい、Cui Guangwei)
研究支援
本研究は、科学技術振興機構(JST)戦略的創造研究推進事業個人型研究(さきがけ)「統合1細胞解析のための革新的技術基盤(研究総括:浜地格)」の研究課題「生体システム理解・医科学応用を実現する1細胞核酸計測技術の開発(研究者:城口克之)」、中谷医工計測技術振興財団技術開発研究助成(特別研究)の研究課題「菌叢ネットワークの理解・コントロールへ向けた”次世代”菌叢解析法の開発(研究者:城口克之)」、理化学研究所共生生物学プロジェクトなどの支援を受けて行われました。
原論文情報
Jianshi Jin, Reiko Yamamoto, Tadashi Takeuchi, Guangwei Cui, Eiji Miyauchi, Nozomi Hojo, Koichi Ikuta, Hiroshi Ohno, Katsuyuki Shiroguchi, “High-throughput identification and quantification of single bacterial cells in the microbiota”, Nature Communications, 10.1038/s41467-022-28426-1
発表者
理化学研究所
生命機能科学研究センター 細胞システム動態予測研究チーム
チームリーダー 城口 克之(しろぐち かつゆき)
研究員 金 坚石(じん じゃんし、Jin Jianshi)
テクニカルスタッフ 山本 れいこ(やまもと れいこ)
報道担当
理化学研究所 広報室 報道担当