2023-03-01 東京大学,琉球大学
発表のポイント
◆魚類のミトコンドリアDNAデータプラットフォーム「MitoFish Suite」は、現在、約3,500魚種のミトコンドリアゲノムデータ、約10,000種の環境DNAデータを有しており、魚種の遺伝的多様性の推定や環境DNAを解析するための情報源として活用されています。
◆今回、これまでよりも高速で正確な解析機能、強化された分析機能や改善されたインターフェースを実装し、機能強化を行いました。
◆魚類の分布調査や資源保全、希少種・新種の発見や保護など、生物資源の持続的発展に関連する幅広い研究分野で活用されると期待されます。

魚類ミトコンドリアDNAデータプラットフォーム「MitoFish Suite」コンセプト図
発表概要
東京大学 大学院新領域創成科学研究科先端生命科学専攻/大気海洋研究所附属地球表層圏変動研究センターの岩崎渉教授を中心とする共同研究チームは、10年以上開発を継続している魚類のミトコンドリアDNAデータに特化したデータプラットフォーム「MitoFish Suite」の大幅な機能強化を行いました。
魚類は世界に34,000種以上生息するとされ、ヒトを含む全脊椎動物の約半数を占めます。赤道域から両極域、川から深海まで、地球上のあらゆる水圏に進出した生物群であり、その中には、水産資源として人間の生活と密接に関連する種や、人間活動の影響で絶滅の危機にある希少種・地域固有種が多く存在します。このことから、魚類の遺伝的多様性を分析・評価することや、環境DNA(注1)に基づいて魚類相を網羅的に分析することは、基礎科学的にも社会的にも重要です。魚類の遺伝的多様性を分析する情報源としては、従来からミトコンドリアゲノム(注2)が多用されており、現在まで多くのデータや知見が蓄積されてきました。
本共同研究チームは、MitoFish Suiteを開発・構築し、10年以上にわたって機能強化を行いながら、世界のデファクトスタンダードの地位を築いてきました。本論文では、最新のアップデートおよび機能強化の成果を報告しています。現在、ミトコンドリアゲノムデータベースMitoFishは魚類約3,500種のミトコンドリアゲノム情報を、魚類環境DNA解析パイプラインMiFish Pipelineは魚類約10,000種のMiFish Primer(注3)領域の配列データを含みます。さらに、ミトコンドリアゲノム解析機能や環境DNAデータ解析機能を高速化し、デノイジング機能(注4)などによる高精度な解析機能を実装したほか、サンプル間の比較分析機能や、インターフェースの改善なども行いました。特にMiFish Pipelineについては、デノイジングを行う解析プログラムを新たに追加することで、解析結果の感度を増強することに成功しました。これにより、MiFish Pipelineが提供しているサンプルデータから、新たに2種の魚類を検出することに成功し、うち1種は絶滅危惧Ⅱ類に指定される希少種でした。
今後、MitoFish Suiteは、ミトコンドリアゲノム情報に立脚した魚類の生態や遺伝的多様性に関する研究を、生命情報科学の側面から今後もさらに強力に推進することが期待されます。生物学がビッグデータ時代を迎えるなか、計算機の使用に親しみのない研究者や潜在的ユーザーにとっては、データ解析のハードルが高くなりつつあります。MitoFish Suiteは、コマンド入力操作を必要としないウェブツールとしてデータ解析やデータベース検索を可能にすることから、生物学や水産学のみならず、環境生態学や水利工学など様々な分野における魚類ミトコンドリアゲノムデータや環境DNA解析の活用をサポートし、さらに促進すると期待されます。
この研究成果は、2023年2月28日付で、学術雑誌『Molecular Biology and Evolution』に掲載されました。
発表内容
〈研究の背景〉
魚類(主として真骨類)は、ヒトを含む脊椎動物の中で最も種数が多い分類群であり、全世界で約34,000種以上が生息すると推定されています。その生息域は、海洋から陸上の河川・湖沼、赤道域から南極・北極域、潮だまりやサンゴ礁などの浅海から海溝深部の超深海にまで及び、地球上のあらゆる水圏に進出した生物群であると言えます。
魚類には、マグロやウナギなど、水産資源として人間の持続的発展と密接に関連する種や、タナゴ類やドジョウ類など、人間活動の影響で絶滅の危機に瀕する希少種や地域固有種が多く存在します。このことから、魚類の遺伝的多様性を分析・評価することや、環境DNAに基づいて魚類相を網羅的に分析することは、基礎科学的にも社会的にも重要です。魚類の遺伝的多様性を分析するための情報源としては、細胞内のコピー数が多いために比較的容易に分析できるミトコンドリアゲノムデータが多用されてきました。
〈研究の内容〉
東京大学大学院新領域創成科学研究科先端生命科学専攻の朱涛(シュ トウ)特任研究員と岩崎渉教授(同大気海洋研究所附属地球表層圏変動研究センター教授兼務)、琉球大学医学部の佐藤行人講師、千葉県立博物館の佐土哲也研究員、宮正樹主任上席研究員は、魚類のミトコンドリアゲノムデータに特化したデータプラットフォームMitoFish Suiteを開発・構築し、10年以上にわたって機能強化を行いながら、世界のデファクトスタンダードの地位を築いてきました。MitoFish Suiteの前身にあたるMitoFishデータベースは、旧東京大学海洋研究所における科学研究費補助金の研究プロジェクトとして2007年頃から作成がスタートしており、当時から数えると15年以上の歴史を持ちます。
岩崎教授らによる2013年の論文公表以降、全世界からのアクセス数は増加を続けています。2018年に琉球大学の佐藤講師らによって2報目の論文が発表された際には、年間被閲覧数が4万回を超えるまでに成長しました。また、同論文の発表時には、MiFish Primerによって増幅された環境DNAデータの自動解析機能MiFish Pipelineを世界に先駆けて提供し、さらに利用が加速しました。両論文の被引用回数は合計で750回を超えており(Google Scholar調べ)、本研究分野で日本を代表する文献となっています。MitoFishは2023年2月時点での収蔵数が全長配列で合計3,562件、部分配列で合計42,398件にのぼります。
今回、これまで問題となっていた魚類の慣用名(common name)および異名(synonym)からのデータベース検索も行えるように改良し、利便性を高めました。また、魚類環境DNA解析パイプラインMiFish Pipelineについても、魚類約10,000種のMiFish Primer領域の配列データを含むように強化しました。ミトコンドリアゲノム解析機能や環境DNAデータ解析機能はより高速化され、高精度な解析機能が実装されました。さらに、サンプル間の比較分析機能や、インターフェースの改善なども行いました。
特にMiFish Pipeline(図1)については、デノイジングを行う解析プログラムを新たに追加することで、解析結果の感度を増強することに成功しました(図2)。従来のMiFish Pipelineでは、ある閾値よりも検出度数が低い配列を一律で除去していましたが、今回の改良によって、そのような検出度数の低い配列についても解析可能となりました。これにより、MiFish Pipelineが提供しているサンプルデータ(沖縄県浦添市・宇地泊川の河川水サンプル)から、新たに2種の魚類、Caranx tille ミナミギンガメアジ(配列頻度12/6714 = 0.18%)とLuciogobius ryukyuensis ミナミヒメミミズハゼ(配列頻度17/6714 = 0.25%)を検出することができました。後者は琉球諸島固有のミミズハゼ属魚類であり、環境省レッドリストで絶滅危惧Ⅱ類に指定されています。今回の結果は、改良版MiFish Pipelineを使った環境DNA解析が、希少種の探索や同定に役立つことを示唆しています。
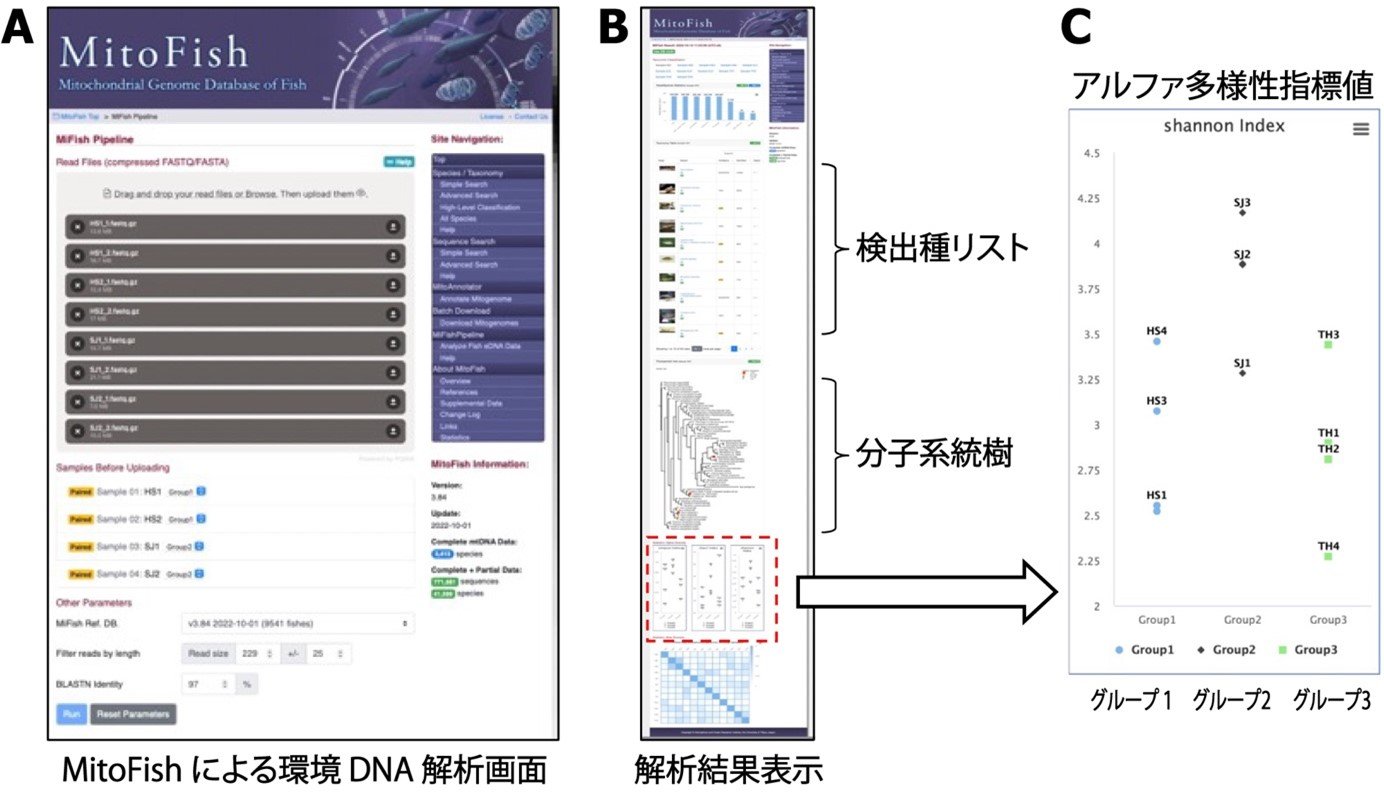
図1:MitoFishを用いた環境DNAデータの自動解析
A:MitoFishが提供する環境DNA自動解析プログラムMiFish Pipelineの実行ページ。高速DNAシークエンサーの出力ファイルfastq.zipをドラッグ&ドロップでデータ転送し、オンライン解析を実行することができる。
B:環境DNAデータ解析の結果表示画面。縦スクロール表示の画面に、検出された種と配列数、魚類種の写真、解析された配列の分子系統樹、入力サンプルのアルファ多様性指標値(注5)などを出力し表示する。
C:推定されたアルファ多様性指標値(Shannon指数H′)の分布プロット図。複数のサンプルをまとめたグループごとに色分けしたプロットを出力し表示する。
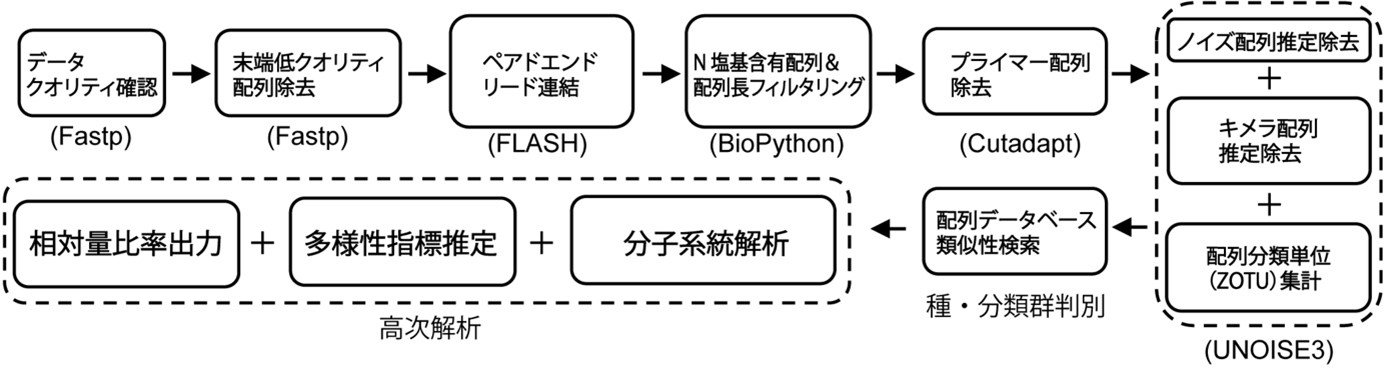
図2:環境DNAデータの自動解析プログラムMiFish Pipelineの概要
ユーザーが入力し転送したデータについて、データ全体のクオリティ確認から、低クオリティ配列末端の除去、ペアドエンド・リードの連結、プライマー配列の除去などのデータ前処理を自動で行う。続いて、ノイズ配列やキメラ配列の推定除去、配列の集計とMitoFishデータベースへの配列照合解析を行うことで、配列が由来する生物種を推定し、結果を出力する。分子系統解析、アルファ多様性指標値、相対配列比率などの高次解析の結果も出力する。
〈今後の展望〉
本研究によって構築、刷新されたMitoFish Suiteプラットフォームは、魚類ミトコンドリアDNA情報に基づいた進化生態学的研究、環境DNA研究、それらに付随するデータベース探索やユーザーデータの解析、データベース登録などを、生命情報科学の側面から強力にサポートすることが期待されます。とくに、高速DNAシークエンサーで産出される大量の塩基配列データは、その解析面において、計算機の使用に親しみのない研究者や潜在的ユーザーにとってハードルが高い場合があります。MitoFish Suiteが提供するウェブツールが、生物学や水産学のみならず、環境生態学や水利工学など、様々な分野の発展に今後も貢献するものと期待されます。
〈関連文献〉
MitoFish and MitoAnnotator: A Mitochondrial Genome Database of Fish with an Accurate and Automatic Annotation Pipeline (Wataru Iwasaki, et al. Molecular Biology and Evolution 2013 )
https://doi.org/10.1093/molbev/mst141
〈研究助成〉
本研究は、文部科学省海洋資源利用促進技術開発プログラム(課題番号:JPMXD1521474594)、科研費(課題番号:22H04925)、JST CREST(課題番号:JPMJCR19S2)の支援により実施されました。
発表者
東京大学大学院新領域創成科学研究科
朱 涛(シュ トウ)特任研究員
岩崎 渉 教授 (東京大学大気海洋研究所 附属地球表層圏変動研究センター 教授)
琉球大学医学部附属実験実習機器センター
佐藤 行人 講師
千葉県立中央博物館
佐土 哲也 研究員
宮 正樹 主任上席研究員
論文情報
〈雑誌〉 Molecular Biology and Evolution
〈題名〉 MitoFish, MitoAnnotator, and MiFish Pipeline: Updates in ten years
〈著者〉 Tao Zhu, Yukuto Sato, Tetsuya Sado, Masaki Miya, Wataru Iwasaki
〈DOI〉 10.1093/molbev/msad035
〈URL〉 https://doi.org/10.1093/molbev/msad035
用語解説
(注1)環境DNA
海水、河川水、土壌などの環境媒質が含有しており、周辺に生息する生物に由来するDNAの総称。
主な由来は、生物の皮膚片や羽毛、粘液などの細胞片や、排泄物などであると考えられている。
(注2)ミトコンドリアゲノム
真核生物の細胞小器官ミトコンドリア内に存在するゲノムのこと。ミトコンドリアゲノムは、細胞あたりの分子数が細胞核ゲノムと比較して多いこと、半数体(ハプロイド)であることから、塩基配列決定などのDNA実験を比較的容易に行うことができる。このため、野生生物の種判別や集団遺伝解析などを行うマーカー遺伝子として多用される。
(注3)MiFish Primer
多様な魚類のミトコンドリアゲノムのDNA配列の一部をPCRによって読み取るために用いられる短いDNA配列。
DNAを「商品のバーコード」のように簡単に読み取ることで、読み取ったDNAの塩基配列から魚の種類が分かる。MiFish PrimerはMitoFishのデータを用いて開発され、性能の高さから世界的に使われている。
(注4)デノイジング
一般にDNA配列を決定する際には、1/1,000から1/10,000程度の確率で塩基の読み取りエラーが発生する。このために、実際の生物に由来する真正の配列とは微妙に異なった「ノイズ配列」が混入する。
このようなノイズ配列と、環境サンプル中に含まれていたレアな生物種に由来する配列をうまく区別し、データ解析の感度を向上する技術をデノイジングと呼ぶ。
(注5)アルファ多様性
あるサンプル、すなわち1つの環境において検出された生物種の多様性を、種の豊富さ(species richness)と均等度(evenness)などに基づいて数学的に評価した要約統計量。
確率論に基づいて推定されるSimpsonの多様度指数D や、エントロピー係数として推定されるShannon指数H′など、複数の指標値が考案されており、目的や用途によって使い分けられる。
お問い合わせ
新領域創成科学研究科広報室